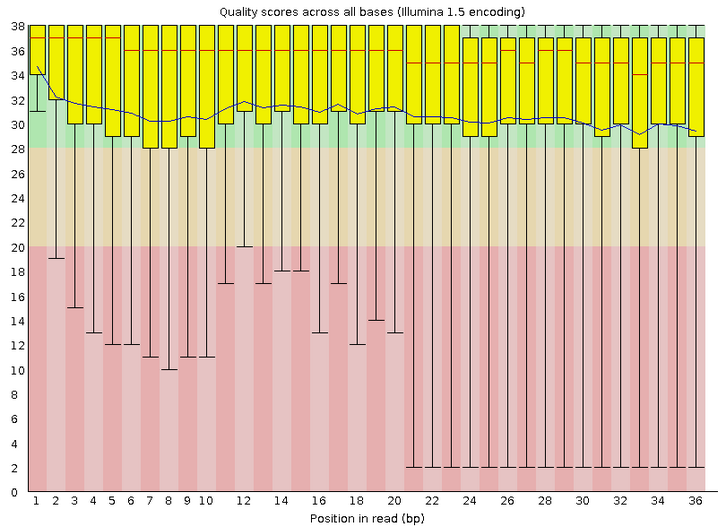
| Анализ ридов: | fastqc chipseq_chunk45.fastq |
| Картирование чтений на хромосому: | bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk22.fastq > chipseq_chunk22.sam |
| Перевод в бинарный формат .bam: | samtools view -b chipseq_chunk22.sam > chipseq_chunk22.bam |
| Сортировка по координатам в референсе начала чтения: | samtools sort chipseq_chunk22.bam 22-sorted |
| Индексация: | samtools index 22-sorted.bam |
| Подсчет чтений, откартировавшихся на геном (всего - 297): | samtools idxstats 22-sorted.bam > idxstats.out |
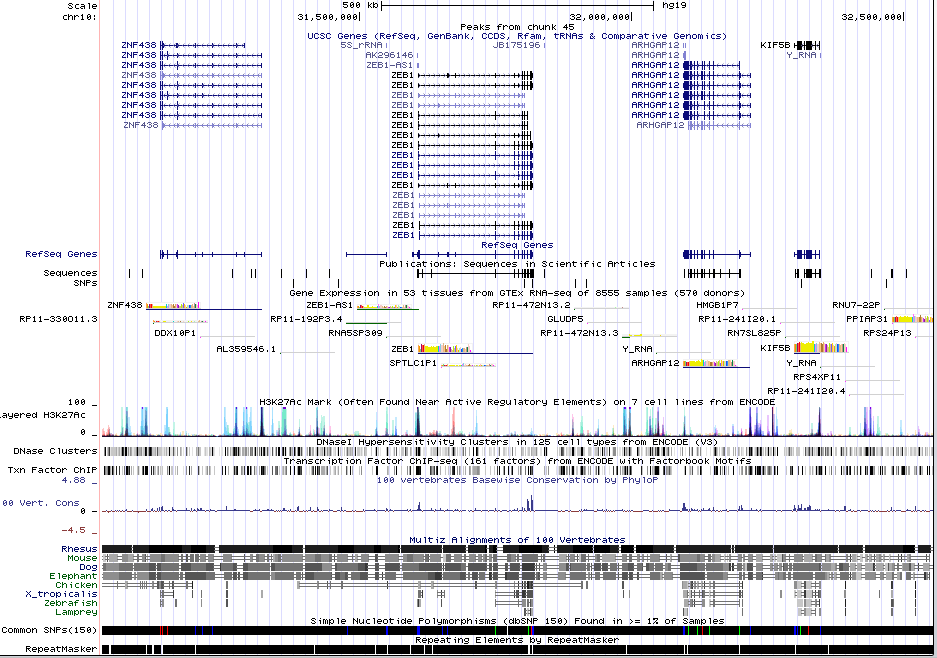
| Поиск пиков: | macs2 callpeak -t chipseq_chunkX.sorted.bam --nomodel -n 22 |