Нуклеотидный BLAST
В данном практикуме ответы на поставленные вопросы отправлялись в google-формах, однако на этой странице Вы можете ознакомиться с ними и получить дополнительные комментарии.
Таксономия и функция последовательности
Для выполнения данного задания была взята последовательность, хроматограмма которой изучалась ранее, по этой ссылке Вы можете ознакомиться с её видом в fasta-формате. И с помощью алгоритма blastn, который выравнивает нуклеотидные последовательности относительно нуклеотидных последовательностей из баз данных, было определено таксономическое положение организма, которому принадлежит данный ген. А именно, Macoma balthica, или балтийская макома - вид морских двустворчатых моллюсков из семейства теллинид. Класс (Двустворчатые), отряд (Veneroida), семейство (Теллиниды), род (Макомы). Раковина треугольной формы красного, жёлтого, зелёного или бело-коричневого цвета длиною от 2 до 3 см. [1]. На Рис. 1 показана выдача blast.
 |
Параметры выдачи и ввода: E-value 1.0E-5 и выбрано выравнивание с 99% идентичностью и 100% покрытием. У первых десятков находок E-value равен 0, что означает, что вероятность получить такое сходство случайно равна нулю. Также с помощью этого алгоритма была установлена последовательность белка, которую кодирует данный ген - cytochrome oxidase subunit 1, или ген субъединицы цитохромоксидазы, он является митохондриальным геном, состоит из 650 пар нуклеотидов.
Алгоритмы BLAST
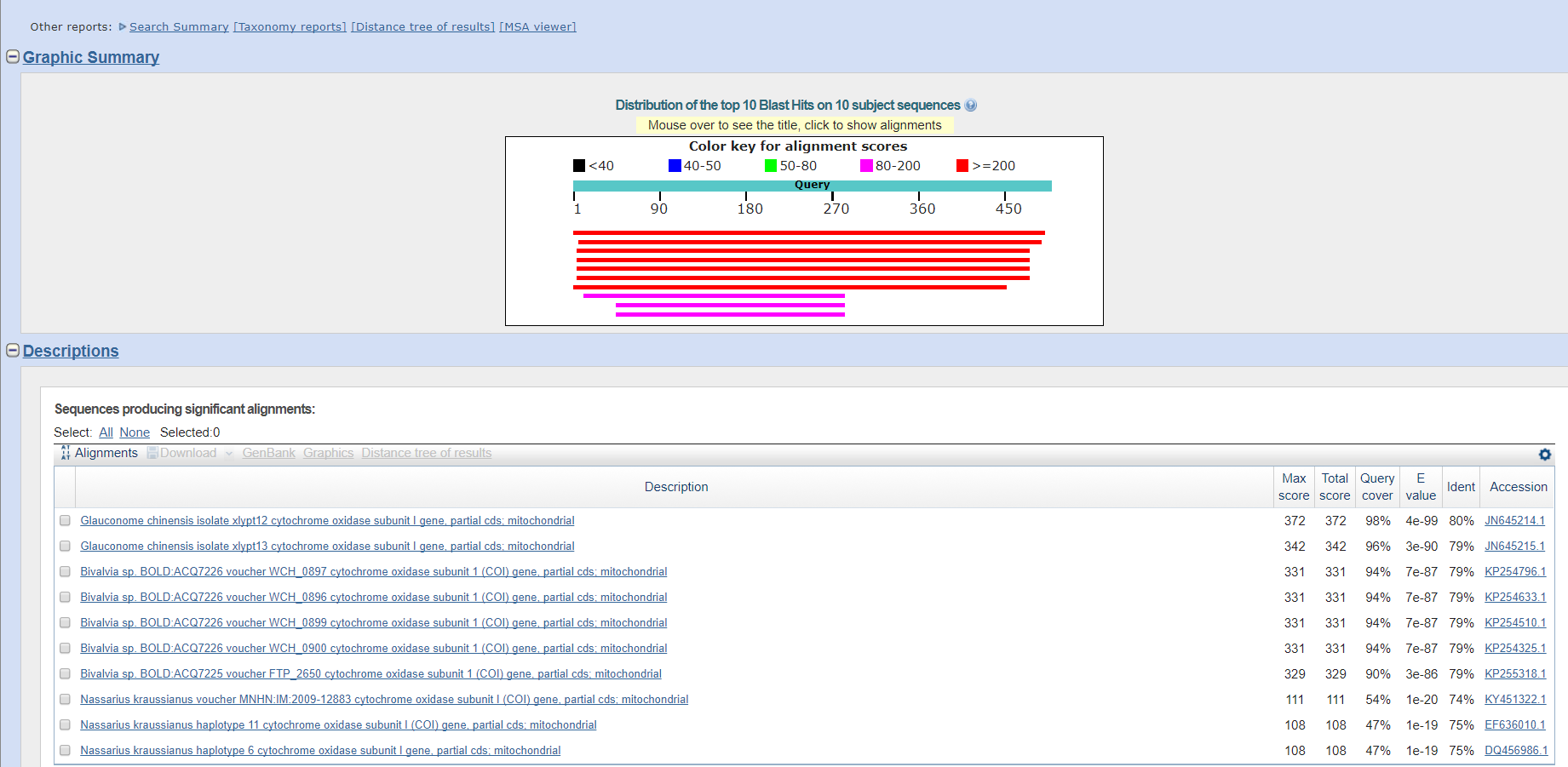
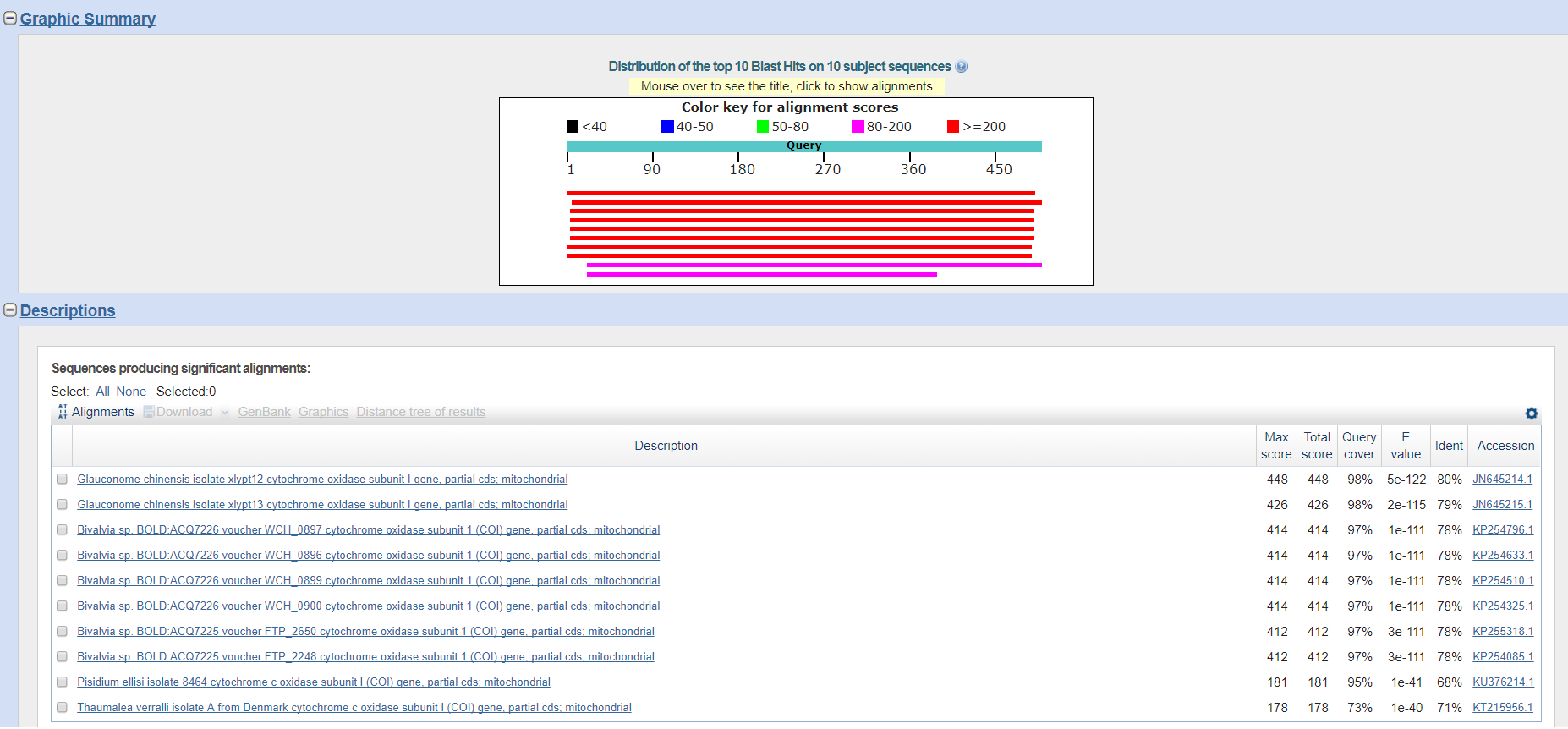
В данном задании анализировалась всё та же последовательность нуклеотидов, но с использованием 3-ёх различных алгоритмов: megablast, blastn и discontiguous megablast. Поиск производился по таким параметрам: blastn, E-value 1e-10, для обнаружения каких-то интересных находок исключен таксон - Tellinoidea, так как анализируемая последовательность белка очень распространена в разных организмах пришлось сделать такое сильное ограничение, так как при иных исключениях идентичность и площадь покрытия были в большинстве случаев 100%. В Таблице 1 приведены результаты по каждому из алгоритмов, а Рис. 2, 3, 4 иллюстрируют выдачу blast.
 |
 |
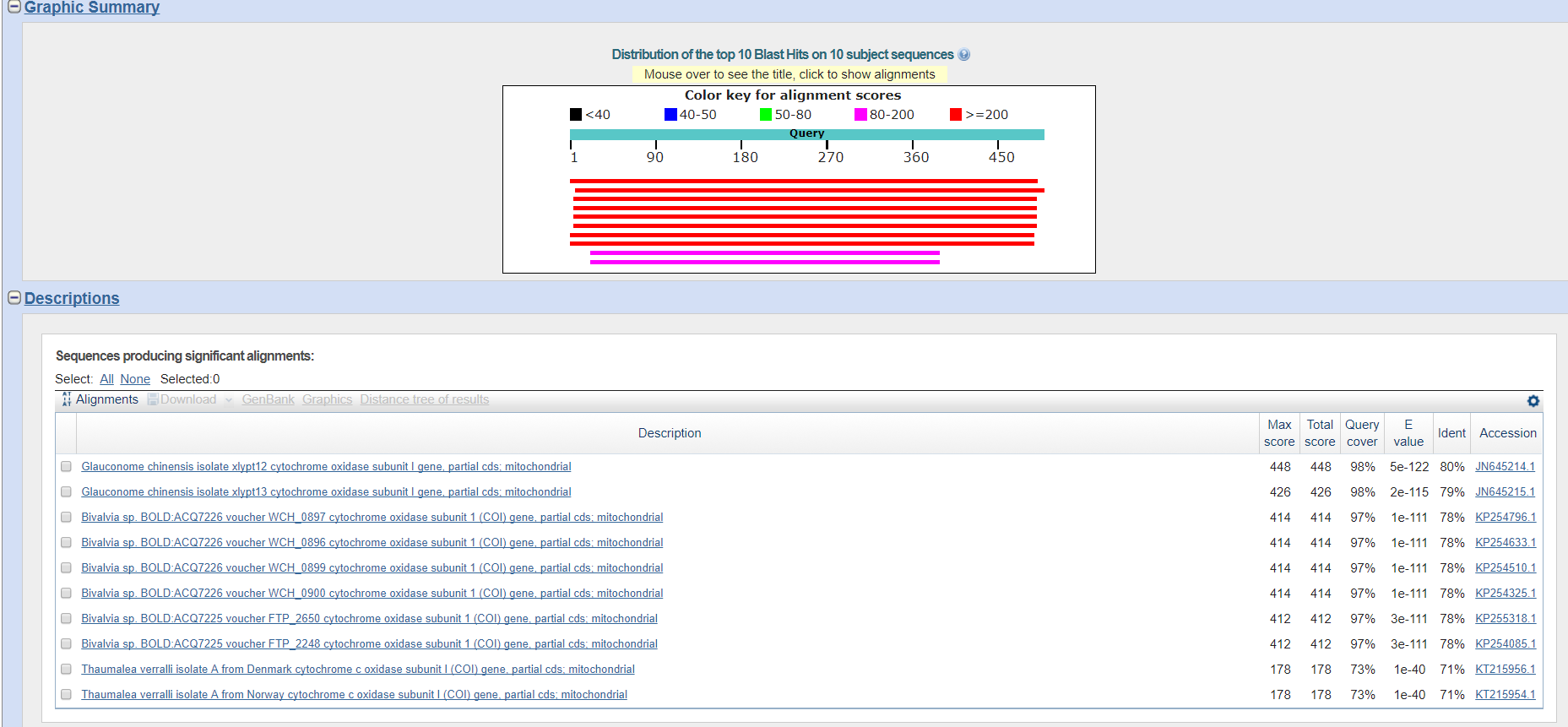 |
Как видно из таблицы и рисунков, blastn и discontiguous megablast выдают почти полностью идентичные находки, а пример последовательности, встречающейся только в этих алгоритмах - Drosophila barutani strain 245 cytochrome oxidase subunit I (COI) gene (E-value - 4e-34, идентичность 69%). Megablast более точен относительно других алгоритмов, так как в нем можно задать большую длину слова, которая используется при поиске, это отражается и при его выдаче, так как при длине 28 он ничего не находил, а в других алгоритмах находки были. Как видно, из скриншотов находки blastn и discontiguous megablast отличаются лишь одной последовательностью, что говорит о сходности их работы.
Гомологичные белки
Для выполнения данного задания был скачан геном организма Amoeboaphelidium protococcarum из афелид - таксон родственных грибам протистов из группы опистоконт [2]. И выбраны 3 белка, относительно которых проводились поиски в геноме этого организма со следующими идентификаторами:
TBB_NEUCR - тубулин - белок, из которого построены микротрубочки - белковые внутриклеточные структуры, входящие в состав цитоскелета, характерны для эукариот [3].
PRPC_EMENI - митохондриальная цитратсинтаза - фермент, катализирующий реакцию конденсации ацетата (ацетил-CoA) и оксалоацетата, в результате чего образуется цитрат [4].
HSP71_YEAST - белки теплового шока - это класс функционально сходных белков, экспрессия которых усиливается при повышении температуры или при других стрессирующих клетку условиях [5].
Затем с помощью локального tblastn, который переводит белковую последовательность в последовательность нуклеотидную и строит выравнивание с геномом организма и ищет в нём сходные последовательности. В Таблице 2 собраны результаты работы этого алгоритма и описание лучшей находки. А по этой ссылке Вы можете ознакомиться со всеми находками.
Были сделаны следующие выводы:
TBB_NEUCR - последовательности очень схожи, небольшие участки отличаются, гэпов нет. Помимо этой находки есть ещё находки с достаточно низким e - value.
PRPC_EMENI - для данного поиска характерно то, что в этом скэффолде два участка посчитаны гомологичными, причем тот, которому соответствует лучшая находка, соответствует с 86-460 а.к.о, а второй 20-85 а.к.о. Помимо этого, программа обнаружила сходную ситуацию и в 157 скэффолде, возможно, в геноме присутствует два участка, в которых есть сходные домены (характеристики находки те же). Довольно большое количество гэпов и есть непродолжительные консервативные участки.
HSP71_YEAST - находка содержит большое количество совпадающих позиций, очень мало гэпов, что свидетельствует, скорее всего, об гомологичности и выполнении одной и той же функции.
Поиск гена
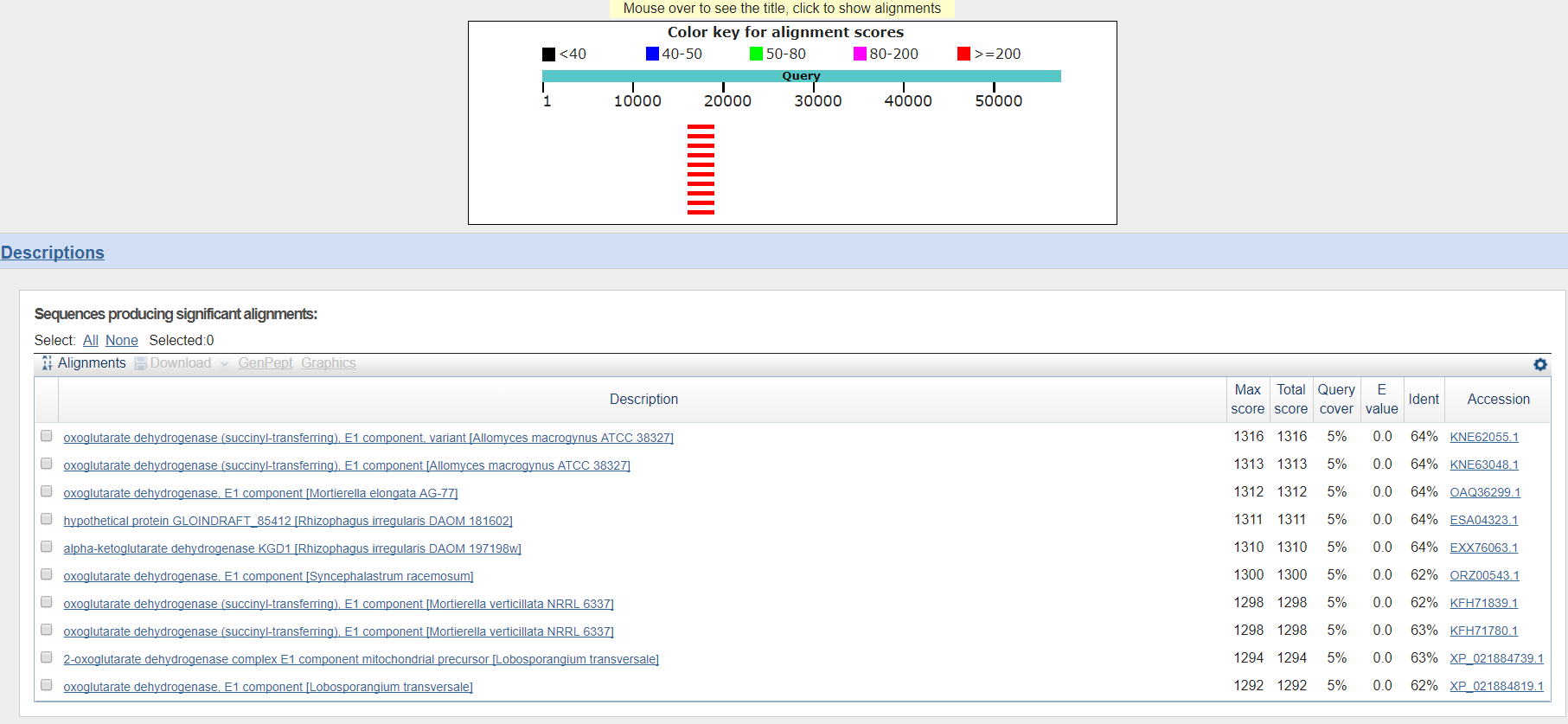
Для выполнения данного задания анализировался всё тот же геном Amoeboaphelidium protococcarum, из которого выбран scaffold-51 длиной 57241 п. н. И с помощью алгоритма blastx, который переводит последовательность нуклеотидов в аминокислотную последовательность и проводит выравнивание этой последовательности относительно белков из белковой базы данных, по следующим параметрам: таксон - Opisthokonta, e-value - 1e-05, максимальное количество находок - 50, nr, остальные параметры по умолчанию. Парметры лучшей находки: мин. координата - 16066, макс. координата - 19011, обратная ориентация. В 50 находках в одной и той же области скэффолда обнаружено сходство с последовательностью компонента E1 оксоглутарат дегидрогеназы - фермента, принимающего участие в цикле трикарбоновых кислот. E-value всех 50 находок равен 0, что так же подтверждает правильность вывода о том, что в данном участке генома закодирован именно этот белок. На Рис. 5 представлена выдача blastx.
 |
Источники:
| [1] Балтийская макома [2] Афелиды [3] Микротрубочки и тубулин [4] Цитратсинтаза [5] Белки теплового шока |