Чтение последовательностей по Сэнгеру
Метод Сэнгера — метод секвенирования (определения последовательности нуклеотидов) ДНК, также известен как метод обрыва цепи. Впервые этот метод секвенирования был предложен Фредериком Сэнгером в 1977 году [1]. Чтобы узнать больше про секвенирование по Сэнгеру можно посмотреть следующее видео, в котором доступно и понятно объясняются принципы, являющиеся ключевыми в секвенировании и по сей день. В современном мире секвенирование проводится в секвенаторах с использованием дидезоксинуклеотидов с разными флуорисцентными метками, каждая из которых выдает разные результаты при возбуждении, что позволяет проводить анализ в одной пробирке. На выходе получаются цветные хроматограммы, с которыми можно работать в разных программах. В данной работе была использована программа Chromas, которая анализирует файлы в формате .ab1 и отображает пики, соответствующие разным нуклеотидам, соответственно следующими цветами:
A - зелёный
T - красный
G - черный
C - синий
Были взяты файлы, соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК, ссылки на которые приведены ниже:
L51_COI_F_H05_WSBS-Seq-07-10-16.ab1 - прямое прочтение
L51_COI_R_H06_WSBS-Seq-07-10-16.ab1 - обратное прочтение
В программе Chromas обратное прочтение было превернуто и представлено в виде комплементарной последовательности, затем анализировались по отдельности прямое и обратное прочтения на наличие проблематичных участков. В Таблице 1 приведена информация о качестве хроматограмм (определенные на глаз) и длине нечитаемых участков на 5'- и 3'-концах.
В некоторых случаях программа затрудняется определить нуклеотид по его сигналу на хроматограмме и ставит литеру N, однако при анализе можно определить какой нуклеотид здесь идет, например, по комплементарной последовательности или очевидно виден пик, который программа не распознала. В хроматограмме можно обнаружить сложные места, которые могут быть объяснены по-разному, ниже представлены примеры таких проблемных мест с предположениями о возможных объяснениях этого.
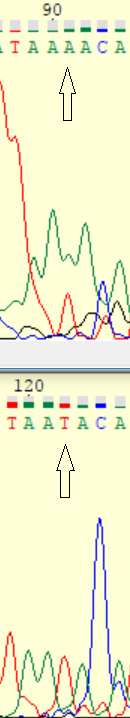 |
В данном примере видно, что в 91 позиции прямой цепи за счет слияния пиков программой был выбран аденин, в то время как в обратной последовательности на этом месте четко виден пик тимина. В хроматограмме прямой последовательности под высоким пиком аденина можно увидеть небольшой пик аденина, который не был разпознан программой. Такая ситуация может объясняться полиморфизмом, наблюдаемым в молекуле ДНК. |
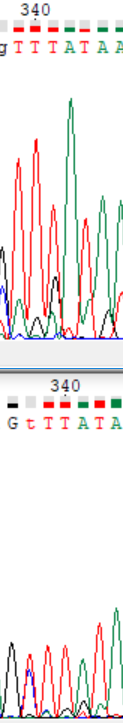 |
Данная иллюстрация показывает, каким образом может помочь в определении последовательности нуклеотидов совместные прямое и обратное прочтение цепи ДНК. Здесь в 339 позиции в прямой цепи однозначно определяется тимин, в то время как для обратной последовательности есть возможность для нахождения в этом месте тимина и цитозина, однако отсутствие пика цитозина в примере доказывает, что в 338 позиции обратной цепи должен быть тимин, что и было исправлено. |
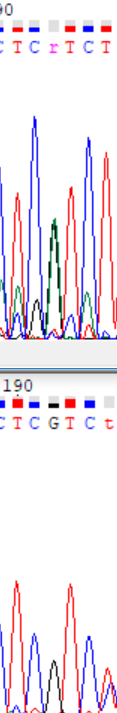 |
На данной иллюстрации в прямом прочтении (сверху) видно четкое наложение пиков аденина и гуанина, что может свидетельствовать о наличи полиморфизма в ДНК в этом случае. Для обозначения полиморфизма была использована литера r, которая означает совместное проявление аденина и гуанина. Полный список, возможных сочетаний букв можно посмотреть по этой ссылке. |
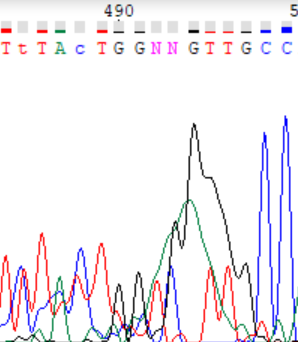 |
Эта иллюстрация отражает ситуацию, когда шумы на хроматограмме могут помешать правильному её прочтению, здесь слишком высокие пики гуанина и аденина лишают возможность точно определить нуклеотиды, которые должны располагаться в данных местах. Такая ситуация может объясняться попаданием пятна краски в детектор. |
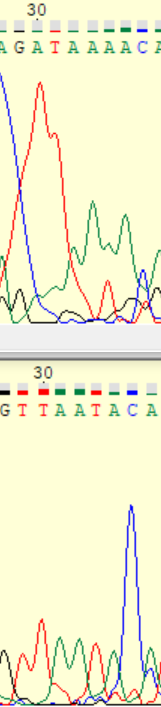 |
В позиции 34 в прямой последовательности можно увидеть отчетливый пик для аденина, под которым находится пик тимина, который и был определен при обратном прочтении ДНК, что может свидетельствовать о существовании полиморфизма в данном месте для данной позиции или же это показывает нам ошибочное прочтение при секвенировании. |
Затем из программы Chromas были сохранены последовательности в fasta-формате (ссылка), в которой маленькими буквами показаны нуклеотиды, выбранные по тем или иным соображениям, примеры обоснований которых приведены выше. С помощью программы needle из пакета EMBOSS выполнялось глобальное вырвавнивание прямой и обратной последовательностей, где для обратной последовательности взята комплементарная ей. Результат данной работе был визуализирован в программе Jalview и приведен на Рис. 1 и по ссылке.
 |
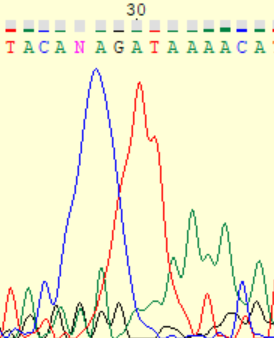
Нечитаемый фрагмент хроматограммы
Для выполнения данного задания был взят нечитаемый участок обратной последовательности, рассматриваемой выше, он приведен на Рис. 2. Возможным объяснением таких высоких пиков может быть прохождение большого количества флуорисцентного красителя через капилляр.
 |
Ссылки на исходные последовательности в fasta-формате прямая, обратная последовательности.
Источники:
| [1] Секвенирование по Сэнгеру |