Сборка генома de novo
В данном задании выполнялся анализ проекта по секвенированию грамотрицательной бактерии Buchnera aphidicola, полученного в базе данных ENA (European Nucleotide Archive) по коду доступа - SRR4240358. Buchnera aphidicola относится к типу Proteobacteria и является основным симбионтом тлей, располагающимся в бактериосомах, расположенных внутри тли [1]. В базе данных был скачан архив, содержащий проект по секвенированию генома бактерии в формате .fastq, а затем с помощью программы gunzip из пакета EMBOSS он был разархивирован.
Подготовка чтений
Сперва был создан файл adapters.fasta, в котором содержалась информация о последовательностях адаптеров для Illumina. Затем с помощью команды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240358.fastq SRR4240358_cl.fastq ILLUMINACLIP:adapters.fasta:2:7:7 |
Файл, скачанный из базы данных, был очищен от возможных остатков последовательностей адаптеров, вследствие чего его размер уменьшился с 1125 MB до 1106 MB. После этого, проводилось удаление плохих букв с концов чтений, оставив только чтения длиной не менее 30 п. н. с помощью команды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240358.fastq SRR4240358_cle.fastq TRAILING:20 MINLEN:30 |
Что привело к сокращению размера файла до 886 MB, за счёт удаления 1862418 ридов, что соответствует 17,66% от всех чтений, 8681421 (82,34%) рид остались.
Подготовка k-меров
Далее с помощью программы velveth, осуществляющей сборку генома на основании графов де Брайна, были составлены k-меры, или слова длины k, из коротких ридов с дальнейшем составлением на их основе графов. Для данного задания бралась величина k-меров, равная 29 и 25 (что соответствует длине перекрытий ридов между собой), и выполнялась команда:
velveth velv 29 -short -fastq SRR4240358_cle.fastq velveth velveth 25 -short -fastq SRR4240358_cle.fastq |
Что привело к созданию двух папок velv и velveth, содержащих 3 файла. Затем с помощью программы velvetg осуществлялась сборка генома на основе созданных k-меров и постройка непосредственно ориентированного графа:
velvetg velv velvetg velveth |
В ранее созданных папках появлялись файлы: contigs.fa, stats.txt и LastGraph, в которых содержалась информация о полученной сборке генома. В Таблице 1 представлена общая информация о сборках при k равном 29 и 25.
Таблица 1. Информация о сборках генома для k-меров разной длины. | ||||||||
k-мер |
Число вершин графа |
N50 |
Максимальная длина контига |
Число ридов |
||||
29 |
715 |
13843 |
30981 |
661591 |
||||
25 |
8528 |
1553 |
12273 |
703824 |
||||
Как видно из Таблицы 1 при длине слов равной 29 длина контига, начиная с которого покрывается больше половины генома, или N50, значительно больше оного при длине 25, что также коррелирует с большей максимальной длиной контига для 29. Однако число ридов, использованных для построения генома больше при длине слова 25, чем при длине 29. А также можно заметить большую разницу между количеством контигов, составленных при k равном 29 и 25.
Далее проводился анализ получившихся геномов, анализ для обоих k-меров сведен в Таблицу 2, где приведена информация о контигах с максимальной длиной и их покрытие. Можно заметить чем больше длина контига, тем меньше их покрытие.
Таблица 2. Информация о сборках генома для k-меров разной длины. | |||||
29 |
25 |
||||
ID |
Длина контига |
Покрытие |
ID |
Длина контига |
Покрытие |
1 |
30981 |
38.504535 |
48 |
12273 |
60.741221 |
6 |
30747 |
38.877451 |
70 |
8332 |
61.265242 |
7 |
27984 |
40.819111 |
105 |
7759 |
61.819564 |
Для дальнейшего анализа полученного генома с помощью программы Excel были подсчитаны среднее значение и медиана величин покрытия и длин контигов обоих k-меров, информация об этом собрана в Таблицу 3.
Таблица 3. Информация о покрытиях и длинах контигов для k-меров разной длины. | |||||||
29 |
25 |
||||||
Среднее значение покрытий |
Медиана покрытий |
Средняя длина контигов |
Медиана длин контигов |
Среднее значение покрытий |
Медиана покрытий |
Средняя длина контигов |
Медиана длин контигов |
288,352 |
14 |
931,82 |
8 |
131,6332 |
6 |
83,037 |
4 |
Исходя из данных, приведенных в Таблице 3, можно сделать вывод, что среди найденных контигов довольно много коротких контигов, но так же присутствуют контиги большой длины, однако для k-мера равного 25 таких контигов меньше, а преобладает число коротких, о чем говорит разница в их средних значениях и в медианах, при том, что число контигов для 25 больше. Помимо этого, видно, что медиана и среднее значения для покрытий контигов так же очень сильно отличаются, однако при проведении анализа на наличие контигов со значением отличающимся от среднего в 5 раз больше или меньше, было обнаружено, что для k-мера 29 число контигов со значением меньше в 5 раз от среднего составляет 548, а со значением больше в 5 раз - только один контиг. В Таблице 4 приведена информация об аномальном большом и о трех аномально малых контигах.
Таблица 3. Информация о покрытиях и длинах контигов для k-меров разной длины. | |||
ID |
Длина контига |
Покрытие |
Отличие от среднего |
670 |
1 |
139456 |
Больше в 483,6 раза |
188 |
397 |
7,45 |
Меньше в 7,74 |
416 |
29 |
5,48 |
Меньше в 10,5 раза |
297 |
25 |
1,32 |
Меньше в 43,69 |
Анализ генома
Для дальнейшего анализа полученной сборки проводилось сравнение самых длинных контигов с хромосомой того же организма (CP009253) с помощью программы BLASTN в NCBI megablast. Приведенные выше контиги с наибольшими длинами в Таблице 2 были выровнены относительно известного генома, результат этого анализа приведен в Таблице 4.
Таблица 4. Анализ расположения найденных контигов относительно генома Buchnera aphidicola. | ||||||||
ID |
Координаты в геноме |
Max score |
Total score |
Query cover |
E-value |
Identities |
Gaps |
Число находок |
1 |
541980 - 550219 |
6639 |
15124 |
64% |
0.0 |
6779/8332(81%) |
156/8332(1%) |
5 |
6 |
153752 - 161738 |
4741 |
12198 |
64% |
0.0 |
6346/8169(78%) |
266/8169(3%) |
3 |
7 |
2004 - 11103 |
5760 |
13683 |
73% |
0.0 |
7229/9221(78%) |
252/9221(2%) |
6 |
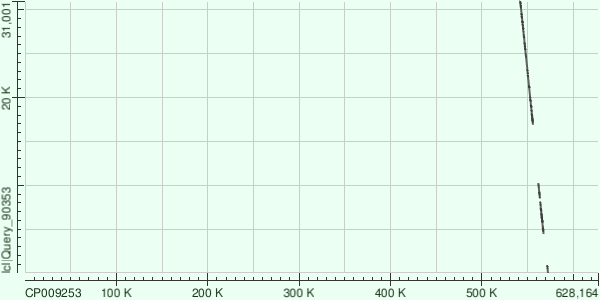
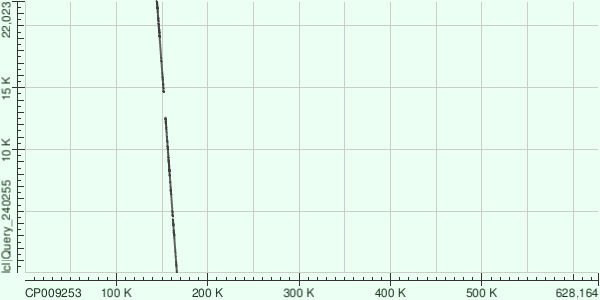
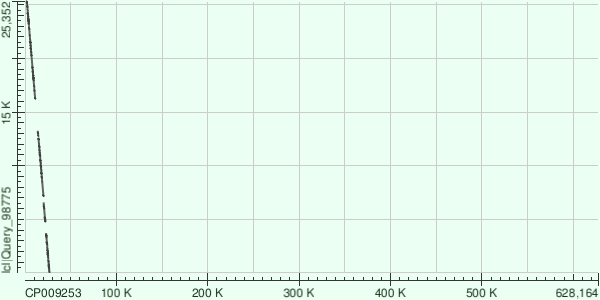
Для каждого из контигов было получено несколько находок, из которых выбраны находки с максимальным покрытием в геноме. Ниже на Рис. 1-3 представлены карты локального сходства, построенные программой BLASTN.
|
|
|
Рис. 1. Карта локального сходства контига 1 относительно CP009253. |
Рис. 2. Карта локального сходства контига 6 относительно CP009253. |
Рис. 3. Карта локального сходства контига 7 относительно CP009253. |
Согласно данным, приведенным в Таблице 4, относительно хромосомы найдено 5 находок, это отражено и в карте локального сходство, из которой мы видим, что контиг ложится частями и не полностью на хромосому, причем в обратном направлении, то есть инвертированы. Присутствуют довольно большие пропуски при сравнении с хромосомой. |
В данном случае наблюдается сходная ситуация, как для Рис. 1, контиг ложится на хромосому с разрывами в двух местах и инвертированно. Вполне возможно, что такой результат можно объяснить пропусками в секвенировании или наличием невыровненных участков. |
В данном случае наблюдается прерывистое наложение контига на хромосому, разрывы наблюдаются в 5 местах, контиг ложится инвертированно. |
Источники:
| [1]Buchnera aphidicola |