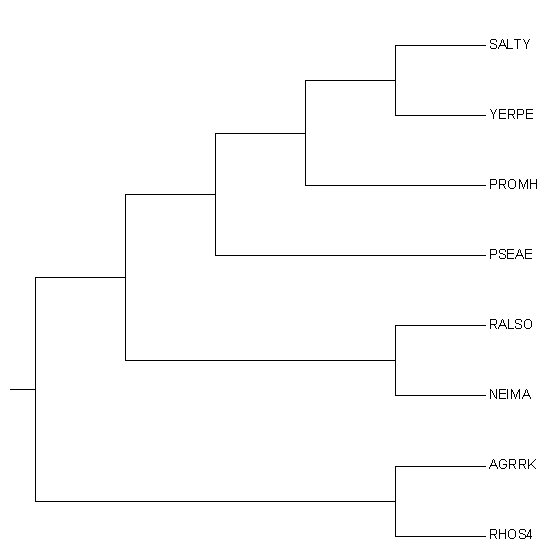Построение и анализ дерева, содержащего паралоги
Для выполнения данного задания был осуществлен поиск гомологов, то есть белков имеющих общее происхождение, белка CLPX_ECOLI - АТФ-зависимая Clp протеаза, среди бактерий, изучаемых в предыдущих практикумах и приведенных в Таблице 1.
Таблица 1. Выбранные протеобактерии. |
|
| Название | Мнемоника |
| Salmonella typhimurium | SALTY |
| Yersinia pestis | YERPE |
| Proteus mirabilis | PROMH |
| Pseudomonas aeruginosa | PSEAE |
| Ralstonia solanacearum | RALSO |
| Neisseria meningitidis | NEIMA |
| Agrobacterium tumefaciens | AGRRK |
| Rhodobacter sphaeroides | RHOS4 |
Поиск гомологов осуществлялся с помощью программы BLASTP с E-value 0.001, при этом последовательность интересующего белка сравнивалась с протеомами бактерий, в результате было найдено 26 белков, которые приведены в Таблице 2, вместе с характеристиками, выданными BLASTP.
Таблица 2. Найденные гомологи. |
||||||||
| Название | Score(bits) | E-value | Название | Score(bits) | E-value | Название | Score(bits) | E-value |
| CLPX_SALTY | 843 | 0.0 | HSLU_RALSO | 98.2 | 3e-22 | Q74RB9_YERPE | 46.2 | 3e-05 |
| CLPX_YERPE | 805 | 0.0 | HSLU_PROMH | 96.7 | 1e-21 | FTSH_SALTY | 46.2 | 3e-05 |
| CLPX_PROMH | 769 | 0.0 | HSLU_RHOS4 | 95.9 | 2e-21 | Q0WBE7_YERPE | 45.8 | 3e-05 |
| CLPX_PSEAE | 654 | 0.0 | HSLU_PSEAE | 95.9 | 2e-21 | B9J9H1_AGRRK | 45.1 | 6e-05 |
| CLPX_RALSO | 615 | 0.0 | HSLU_YERPE | 95.1 | 3e-21 | Q3J045_RHOS4 | 43.5 | 2e-04 |
| B9JD32_AGRRK | 595 | 0.0 | HSLU_AGRRK | 91.7 | 4e-20 | Q8XZ78_RALSO | 43.5 | 2e-04 |
| CLPX_RHOS4 | 568 | 0.0 | HSLU_SALTY | 91.7 | 4e-20 | Q9HV48_PSEAE | 43.5 | 2e-04 |
| CLPX_NEIMA | 557 | 0.0 | B4F2B3_PROMH | 46.6 | 2e-05 | RUVB_NEIMA | 42.7 | 2e-04 |
| Название | Score(bits) | E-value | ||||||
| Q8XY02_RALSO | 42.7 | 3e-04 | ||||||
| YIFB_SALTY | 42.4 | 3e-04 | ||||||
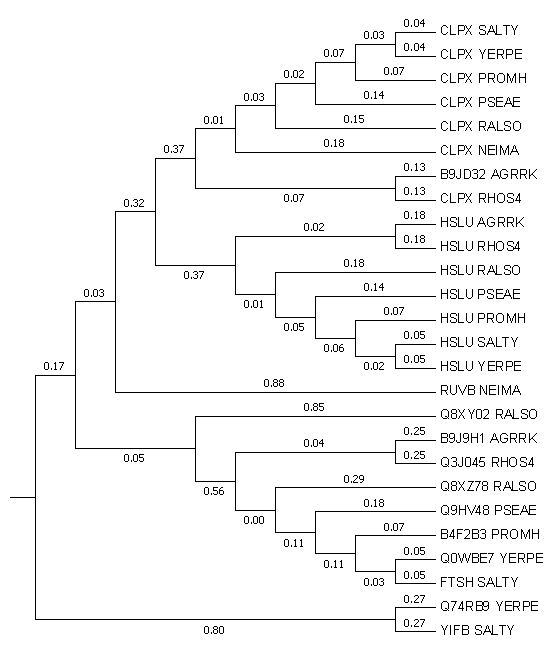
Далее с помощью сервера Muscle было построено выравнивание этих белков, которое затем было интерпретировано алгоритмом UPGMA в укорененное дерево, представленное на Рис.1, а на Рис.2 показано дерево, построенное ранее, отражающее возможную эволюцию видов данных бактерий. Ссылка на проект выравнивания.
|
|
Рис. 1. Дерево гомологов. |
Рис. 2. Дерево видов. |
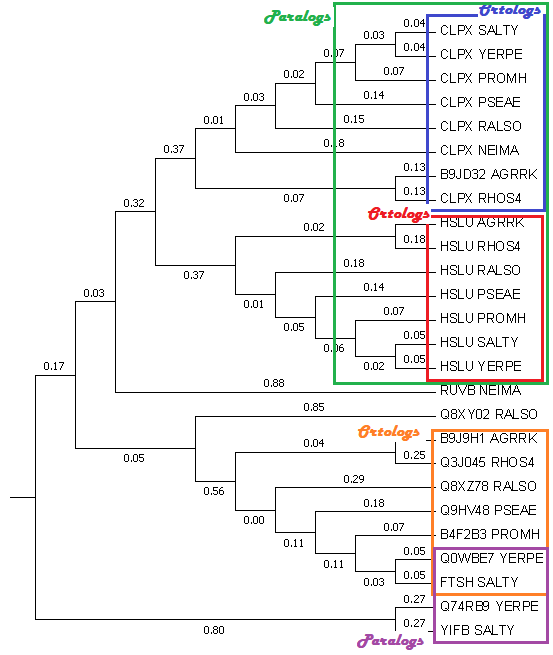
Можно заметить, что белки группы HSLU - АТФ-зависимая протеаза, характерная для многих видов бактерий - являются ортологами между собой и их разделение произошло в результате видообразования [1]. Группа белков CLPX так же является ортологичной, однако белок, который присутствует в организме Agrobacterium tumefaciens с мнемоникой B9JD32 является ортологичным этой группе. Эти белки являются гомологами и разошлись в результате видообразования. Ортологами так же являются Q0WBE7 и FTSH. Однако между собой белки из групп HSLU и CLPX являются паралогами, то есть их разделение осуществилось благодаря дупликации генов в одном и том же организме. Ещё паралогами являются Q8XZ78 и Q8XY02. На Рис.3 наглядно показаны ортологи и паралоги. Если обратить внимание на гомологичную группу белков CLPX, то можно заметить, что их топология в точности совпадает с той, которую демонстрирует дерево видов. |
|
Рис. 3. Ортологи и паралоги. |
Источники:
| [1]HSLU |