Реконструкция эволюции доменной архитектуры
Целью данного проекта было реконструировать эволюцию доменной архитектуры белков, содержащих один и тот же домен Pfam.
Построение выравнивания представителей домена Pfam белков с разной доменной архитектурой
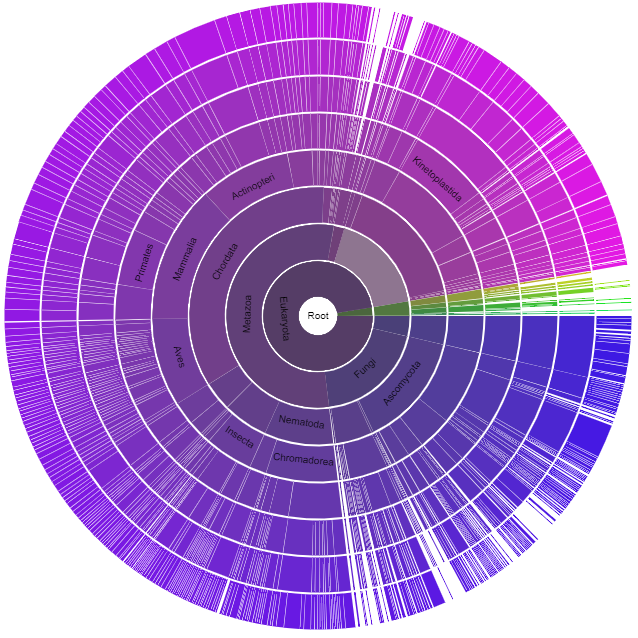
В рамках указанной выше задачи был выбран домен Peptidase_C2 (PF00648) - домен семейства белков кальпаинов, являющихся цитозольными кальций-активируемыми цистеиновыми протеазами. Они разрезают различные внутриклеточные белки и таким образом модифицируют их функции, а также участвуют в нейродегенеративных процессах и апоптозе. Действия таких протеаз практически необратимо и, скорее всего является одной из причин опасности длительного повышения уровня кальция в цитозоле [1]. Помимо этого, гиперактивации кальпаина связана с рядом заболеваний, связанных с измененным гомеостазом кальция, например, болезнь Альцгеймера [2]. В базе данных Pfam присутствует 204 различных доменных архитектур, 4873 последовательностей, 4 взаимодействий, 823 видf и 27 структур. На Рис. 1 представлена визуализация, обеспечивающая графическое представление распределения этого семейства доменов по видам, найденное в базе данных Pfam. |
 |
Сначала было построено выравнивание всех последовательностей, включающих данный домен, помимо этого для последовательности CAN1_HUMAN приведена его 3D-структура, полученная по идентификатору 2ARY. Архив с выравниванием в fasta-формате и с проектом выравнивания доступен по ссылке. Далее в работе использовались доменные архитектуры, представленные на Рис. 2.
Помимо выбранного домена, в первой архитектуре не присутствует больше никаких доменов, а во вторй присутствует домен Calpain_III (PF01067) - который соответствует домену большой субъединицы кальпаина. С помощью указанного скрипта swisspfam_to_xls.py были отобраны последовательности с выбранным доменом и представлены ввиде таблицы Excel. Команда:
python swisspfam_to_xls.py -z -i /srv/databases/pfam/swisspfam.gz -p PF00648 -o PF00648.xls |
После чего, была создана сводная таблица с помощью средств Excel, где строками являются AC всех последовательностей, а по столбцам различные домены Pfam с указанием их встречаемости в последовательностях. Далее для каждого AC в базе данных Uniprot была скачана их таксономия благодаря скрипту uniprot_to_taxonomy.py. Команда:
python uniprot-to-taxonomy.py -i uniprot.txt -o taxonomy.xls |
Таксономия была добавлена в сводную таблицу с помощью функции ВПР, а также указаны длины каждого домена в последовательностях. Ранее обозначенные выбранные для анализа доменные архитектуры были найдены в сводной таблицы и отобран таксон Eukaryota с тремя подтаксонами: Euglenozoa, Metazoa и Fungi, примерно по 20 представителей для каждого подтаксона у каждой архитектуры. Эти последовательности были отмечены знаком +, причем для каждого подтаксона были введены свои идентификаторы: Euglenozoa - E, Metazoa - M и Fungi - F, а также отмечены цифрами доменные архитектуры: цифра 1 для архитектуры, в которую входит только 1 домен и цифрой 2, в которую входят 2 домена, соответственно. После чего, с помощью ряда скриптов были получены файлы с AC выбранных белков, удобное обозначение для последовательностей, а также в дальнейшем выравнивании переименование последовательностей, обозначенные соответственно 1-3. По данной ссылке доступна получившаяся сводная таблица. Далее по полученным AC выбранных белков из построенного ранее выравнивания последовательностей с доменами были выбраны необходимые последовательности с помощью предложенного скрипта filter-alignment.py. Команда:
python filter-alignment.py -i pept.mfa -m ID_selected.txt -o selected.fasta -a "_" |
После чего, с полученным выравниванием проводилась работа, а именно: для каждой выбранной доменной архитектуры были указаны соответствующие группы и выделены по различным окраскам, так, первой архитектуре соответствует окраска ClustalX, а второй - BLOSUM62, порог консервативности выбран равным 20, были удалены последовательности, представленные очень короткими фрагментами, а также явно выравненные неправильно, удалены N- и C-концевые участки в том случае, так как в них не было хорошего выравнивания, а также большое количество гэпов. По данной ссылке доступен получившийся проект выравнивания, также его участок представлен на Рис. 3.
По полученному выравниванию видно, что последовательности первой доменной архитектуры более консервативны, чем второй, и что консервативные колонки сходных для обеих архитектур, что неудивительно, так как домены одни.
Построение филогенетического дерева последовательностей домена
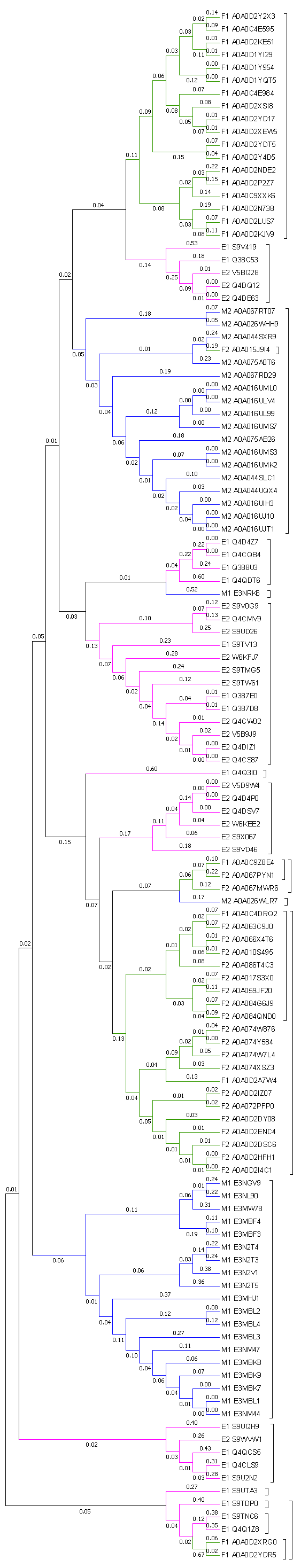
Далее с помощью метода Neighbor-Joining в программе MEGA было построено дерево, представленное на Рис. 4.
 |
|
Выше указана расшифровка идентификаторов для каждой последовательности, так как были выбраны три подтаксона дерево получилось довольно громоздким, для облегчения восприятия ветви дерева были выделены следующими цветами по разным подтаксонам: зеленым - Fungi, розовым - Euglenozoa и синим - Metazoa. Можно заметить, что в большинстве своем подтаксоны собраны в соответствующие клады, причем в кладе одного подтаксона могут присутствовать последовательности как из 1, так и из второй доменных архитектур. Помимо этого, выделяется то, что представители подтаксона Metazoa в основном сгруппированы вместе и обособлены от двух других таксонов, в то время как последовательности двух других подтаксонов встречаются вместе, что может свидетельствовать о том, что у этих доменов был общий предок, у которого после чего в результате мутаций произошло разделение, а потом эволюция шла параллельно, а так же параллельно с Metazoa, с которыми они имели общего предка (верхние клады). Так же видно, что доменные архитектуры 1 и 2 типов сходны, однако точно установить последовательность эволюционных событий сложно, как и определить общего предка, засчет того, что анализировалось много последовательностей (больше 100). Ссылка на скобочную формулу. Также по данному выравниванию было построено дерево методом bootstrap поддержки с количеством реплик 100, представлено на Рис.4. Видно, что деревья отличаются, причем акой способ построения доказывает, что определние общего предка является проблемой. Можно заметить, что некоторые клады довольно верно были определены, как и на первом дереве присутствуют клады подтаксона Metazoa для перовой доменной архитектуры, клада для Fungi второй и первой доменных архитектур, а также клада Euglenozoa.
Источники:
| [1]Кальпаин [2]Wikipedia calpain |