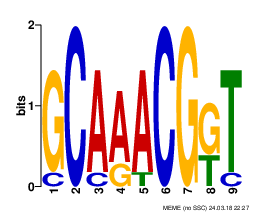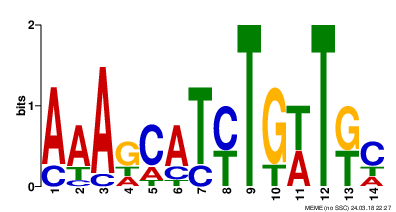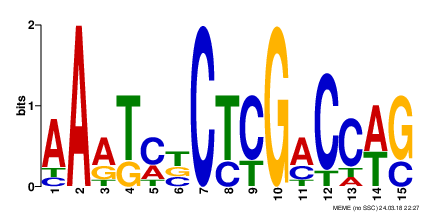Поиск мотивов с помощью программы MEME
Для выполнения данного задния была выбрана Escherichia coli - хорошо изученная гаммапротеобактерия, которая относится к домену Bacteria, типу Proteobacteria, классу Gammaproteobacteria, порядку Enterobacterales, семейству Enterobacteriaceae, роду Escherichia и виду Escherichia coli. В данной работе изпользовался штамм K12, который в базе данных Uniprot можно найти по мнемонике ECOLI. Информация о белках, выбранных для поиска сайта связывания транскрипционного фактора, регулирующего синтез пуринов, собрана в Таблицу 1.
Таблица 1. Выбранные белки. |
||||
| Entry | Entry name | Proteins name | Gene names | Coordinates |
| P0ADG7 | IMDH_ECOLI | Inosine-5'-monophosphate dehydrogenase | guaB guaR, b2508, JW5401 | complement(2631260..2632726) |
| P04079 | GUAA_ECOLI | GMP synthase [glutamine-hydrolyzing] | guaA b2507, JW2491 | complement(2629614..2631191) |
| P0AB89 | PUR8_ECOLI | Adenylosuccinate lyase | purB b1131, JW1117 | complement(1192193..1193563) |
| P0ACP7 | PURR_ECOLI | HTH-type transcriptional repressor PurR | purR b1658, JW1650 | 1739558..1740583 |
| P15254 | PUR4_ECOLI | Phosphoribosylformylglycinamidine synthase | purL purI, b2557, JW2541 | complement(2690312..2694199) |
| P0AG16 | PUR1_ECOLI | Amidophosphoribosyltransferase | purF b2312, JW2309 | complement(2434167..2435684) |
| P08179 | PUR3_ECOLI | Phosphoribosylglycinamide formyltransferase | purN b2500, JW2485 | 2620890..2621528 |
| P0A7D4 | PURA_ECOLI | Adenylosuccinate synthetase | purA adeK, b4177, JW4135 | 4409367..4410665 |
| P33221 | PURT_ECOLI | Formate-dependent phosphoribosylglycinamide formyltransferase | purT b1849, JW1838 | 1932595..1933773 |
Для выполнения данного задания из базы данных EMBL был скачан геном выбранной бактерии с AC:AP009048, далее были выбраны Upstream-регионы, которые задавались, как 100 нуклеотидов, предшествующие старт-кодонам, и записаны в файл, доступный по ссылке. Затем производился поиск мотивов с помощью программы MEME, результат можно посмотреть на данной html-странице. Команда:
ememe seq.fasta meme -nmotifs 3 -revcomp |
Информация о найденных мотивах собрана в Таблицу 2. Можно заметить, что не было найдено ни одного хорошего мотива, то есть с E-value < 0.001, однако среди трех мотивов есть и мотивы с довольно большими значениями информативности и энтропии. Из трех мотивов, на мой взгляд, наилучшим является первым, так как значение E-value для него наименьшее, а последнему мотиву, хоть и с большими значениями информативности и энтропии не информативен из-за большого E-value.
Таблица 2. Информация о мотивах. |
|||||
| Номер мотива | Информативность | Энтропия | Длина мотива | Число белков, в которых встретился мотив | E-value |
| 1 | 14.1 | 14.3 | 9 | 8 | 2.2e-002 |
| 2 | 15.7 | 15.1 | 14 | 9 | 6.1e+001 |
| 3 | 17.0 | 17.1 | 15 | 7 | 2.5e+003 |
|
|
|
Рис. 1. Мотив 1. |
Рис. 2. Мотив 2. |
Рис. 3. Мотив 1. |
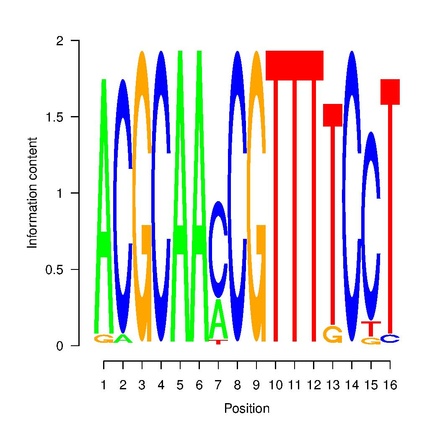
На рисунках 1-3 представлены найденные мотивы, а Рис.4 отображает LOGO для сайта связывания пуринового репрессора E.coli. Можно заметить, что сходные паттерны присутствуют в первом мотиве, в частности, он почти полностью находится в оригинальном LOGO кишечной палочки, что говорит о сходстве сайта связывания пуринового репрессора между изучаемыми видами. Так, повторяются GCAA 1-4 позиции мотива 1 и 3-6 у кишечой палочки, далее в мотиве 1 идет в подавляющем большинстве A, в то время как у E.coli далее конкурируют A и C. В следующих 2 позициях снова обнаруживается полное совпадение, а в 8 позиции в мотиве 1 конкурируют G и T, тогда как у кишечной палочки в этом месте (позиция 10) однозначно идет T, следующая позиция совпадает. |
|
Рис. 4. LOGO для сайта связывания пуринового репрессора E.coli [1]. |
Источники:
| [1]LOGO для сайта связывания пуринового репрессора E.coli |