Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
BLASTДля выполнения заданий была взята последовательность белка NAD-dependent epimerase/dehydratase из организма археи Desulfurococcus kamchatkensis 1221n (идентификатор в базе данных RefSeq YP_002427953.1). Исходная последовательность доступна в fasta-формате. Задание 1. С помощью BLASTP были найдены последовательности, сходные с исходной последовательностью. Для этого в окно для искомой последовательности (Query Sequence) я скопировала последовательность моего белка в fasta-формате. В окне Database выбрала базу данных UniProtKB/Swiss-Prot, алгоритм - blastp (protein-protein BLAST). Далее были изменены дополнительные параметры алгоритма. Чтобы получить максимальное число находок, я изменила количество выводимых на экран находок (Max target sequences 20000) остальные параметры были оставлены по умолчанию ( условия поиска). Всего было найдено 443 последовательности, из них 286 из организма бактерий, 5 из организма архей, 146 - эукариот и 6 - вирусов (Formatting options -> Organism). Из найденных последовательностей были выбраны лучшая, худшая последовательности и одна из середины. В таблице 1 представлена информация о выбранных находках и полученных выравниваниях. Сами выравнивания представлены на Рис. 1-3.


Из полученных находок гомологами исходной последователньости можно считать 299 последовательностей. За условный критерий было принято, что последовательность можно считать гомологичной, если E-value < 1e-3 и не менее 70% запроса вошло в полученное выравнивание (Query cover). Для того, чтобы найти находки-гомологи, был задан максимальный E-value 0.001 в Formatting options. На рисунке 4 изображено графическое представление результатов поиска. Задание 2. Я провела поиск для отдельного таксона. Для этого я вернулась на страницу с параметрами запроса и в поле Organism указала Homo sapiens (условия поиска). Всего найдено 7 последовательностей. Среди находок я выбрала последовательность Stero-4-alpha-carboxylate 3-dehydrogenase, decarboxylating, которая была также найдена в первом поиске последовательностей. В двух находках совпадает все (sp|Q15738.2|NSDHL_HUMAN, Score 70.1, одинаковое выравнивание), кроме значения E-value. При поиске по отдельному таксону оно равно 2e-13, а без указания организма - 3е-12. E-value показывает, насколько случайна полученная находка, и т.к. первый банк был больше, то и полученная находка более случайна. Задание 3. Для создания карты локального сходства была выбрана последовательность белка Tetraketide alpha-pyrone reductase 2 (идентификатор в UniProtKB - Q9CA28). Далее было выполнено выравнивание двух последовательностей с помощью BLAST (условия поиска). Локальная карта сходства представлена на рисунке 5. Из карты локального сходства видно совпадающий участок примерно с 10 остатка последовательности
моего белка (отложена по горизонтали) и с начала гомологичной последовательности
(отложена по вертикали). В выравнивании присутствуют 4 гэпа, что соответствует местам прерывания линии
на графике.
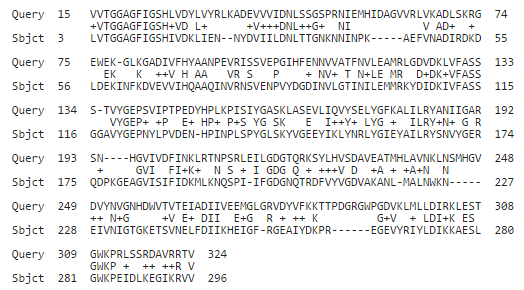
Задание 4. Из множественного выравнивания align_09.fasta была создана база данных при помощи команды makeblastdb (параметр -dbtype prot). Предварительно из выравнивания были удалены все гэпы. Итого, в базе данных было 6 последовательностей. Затем я произвела поиск в этой базе последовательность моего белка командой blastp. Информация о наиболее удачных находках представлена в таблице 2. Выравнивание представлено на рисунке 7 и 8 соответственно.
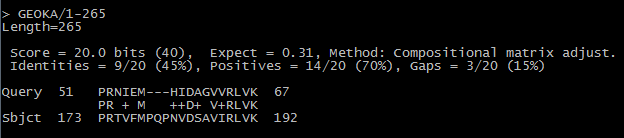
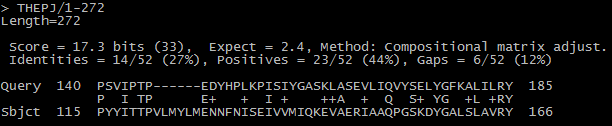
Из таблицы и изображений выравниваний видно, что длина полученных выравниваний небольшая, значение Bit score низкое относительно исследованных выравниваний в предыдущих заданиях. Значение E-value довольно большое для данной базы данных. Это позволяет сделать вывод, что данные находки случайны, и нет повода говорить о гомологии данных последовательностей. |