Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Семейства белковых доменовДля выполения данного практикума была взята аминокислотная последовательность белка НАД-зависимой эпимеразы-дегидратазы из организма Desulfurococcus kamchatkensis (идентификатор белка в базе данных RefSeq YP_002427953, в базе данных UniProt B8D2N2). Задание 1. Поиск доменов Pfam в последовательности белка. Pfam - база данных, содержащая информацию о выравниваниях белковых доменов. Был найден всего один домен - Epimerase, семейство - Epimerase (PF01370). Был сохранен Seed данного семейства (ссылка Alignments), содержащий 96 последовательностей (всего последовательностей 38903), fasta-формат. Полученное в JalView изображение Seed представлено на рис.1.  Задание 2. В данном выравнивании я выбрала в качестве блока колонки в позициях 1-11. Изображение блока представлено на рис.2, а также в окне Jalview block. 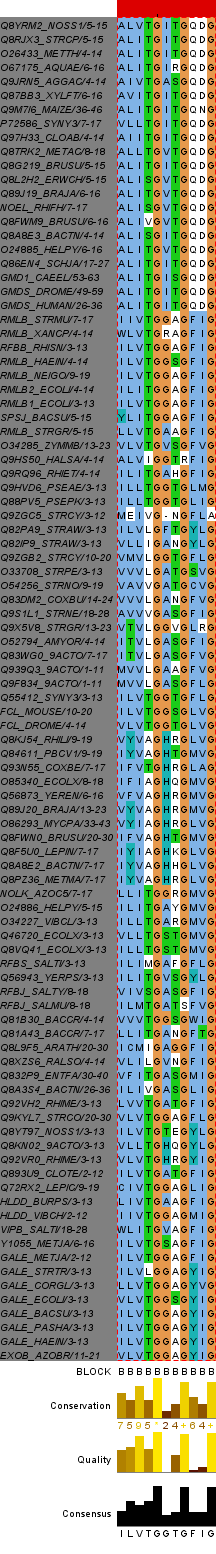 Блок в fasta-формате представлен здесь. С помощью Jalview была получена консенсусная последовательность блока (fasta-формат). По последовательностям на участке данного блока с помощью сервера http://weblogo.berkeley.edu/ был получен logo данного мотива, показывающий распространенность различных остатков в каждой из позиций (Рис. 3). 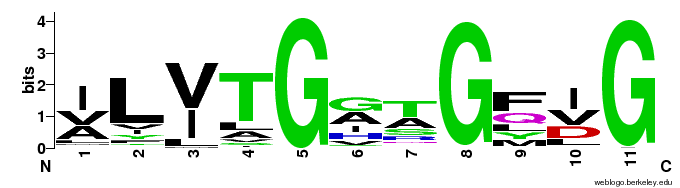
Задание 4. Паттерны. Требовалось с помощью сервиса http://prosite.expasy.org/scanprosite построить сильный и слабый паттерны для одного из блоков или кластеров выравнивания и найти число последовательностей, содержащих мотив с данным паттерном, в БД SwissProt. По сильному паттерну было найдено 35 последовательности. 25 последовательностей соответствуют гомологичным последовательностям из семейства, с которым я работала в предыдущем практикуме по PSI-BLAST. Остальные 10 последовательностей являются изморфными формами этих белков. Для поиска был использован следующий паттерн: [IVAL]-[LVI]-[VIL]-T-G-[AIV]-[TS]-G-[FLM]-[IVL]-G. Одной буквой обозначены позиции, в которых колонки были абсолютно консервативными. Функционально консервативные колонки были расширены до всех гидрофобных. В 7 позиции были выбраны гидрофильные аминокислоты: серин и треонин. Для составления слабого паттерна я заменила 6, 7 и 9 позиции символом "х", так как там наблюдается вариативность кислот. Полученный слабый паттерн: [IVAL]-[LVIF]-[VIL]-[LAV]-G-x-x-G-x-[IVL]-G. По нему в базе данных SwissProt было найдено 426 последовательностей. По слабому паттерну находятся также белки-представители других семейств. Проект в формте jvp. |