Предсказание парных выравниваний
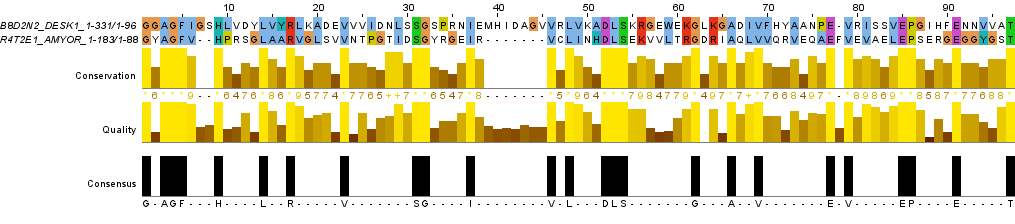
Для выполнения заданий было взято множественное выравнивание №09, последовательность
6 белков, которые представлены в данном
fasta-файле. Последовательности были отсортированы с помощью дерева методом Neighbour Joining Using BLOSUM62.
Затем были получены изображения выравнивания с раскраской BLOSUM62 с порогом консервативноcти 30. Проект в формате .jvp можно скачать по ссылке.
Из исходного множественного выравнивания (рис.1) были выбраны 2 наименее схожие последовательности: OCEIH; GEOKA. Последовательности были выбраны с помощью метода главных
компонент (Principal component analysis) по наибольшему расстоянию между точками, соответствующими последовательностям. Полученное парное выравнивание
последовательностей представлено на рисунке 2.
Рис.1. Исходное множественное выравнивание последовательностей из файла align_09.fasta с раскраской BLOSUM62 с порогом консервативности 30.
Рис.2. Полученное парное выравнивание последовательностей OCEIH и GEOKA; раскраска ClustalX.
Далее с помощью программ needle и water были построены парные выравнивания выбранных последовательностей. Needle строит глобальное выравнивание, water - локальное.
На рисунках 3 и 4 представлены глобальное и локальное выравнивания соответственно (стандартные параметры: матрица BLOSUM62, gap opening penalty = 10.0, gap extension penalty
= 0.5. Gap opening penalty - штраф за открытие гэпа; gap extension penalty - штраф за длину гэпа).
Выравнивания в fasta-формате: needle; water.
Рис.3. Глобальное парное выравнивание последовательностей OCEIH и GEOKA, полученное с помощью программы needle с параметрами gap opening penalty = 10.0,
gap extension penalty = 0.5; раскраска ClustalX.
Рис.4. Локальное парное выравнивание последовательностей OCEIH и GEOKA, полученное с помощью программы water с параметрами gap opening penalty = 10.0,
gap extension penalty = 0.5; раскраска ClustalX.
Затем я изменила параметры характеристик gap opening penalty и gap extension penalty. Для глобального выравнивания я уменьшила штраф за открытие гэпа (gap opening
penalty = 1.0). Полученное выравнивание представлено на рисунке 5, fasta-формат.
Рис.5. Глобальное парное выравнивание последовательностей OCEIH и GEOKA, полученное с помощью программы needle с параметрами gap opening penalty = 2.0,
gap extension penalty = 0.5; раскраска ClustalX.
Для локального выравнивания был уменьшен штраф за длину гэпа (gap extension penalty = 0.2), но получившееся выравниваниие ничем не отличалось от представленного
на рисунке 4. Поэтому я уменьшила штраф и за открытие гэпа (gap opening penalty = 4.0). Полученное выравнивание представлено на рисунке 6, fasta-формат.
Рис.6. Локальное парное выравнивание последовательностей OCEIH и GEOKA, полученное с помощью программы water с параметрами gap opening penalty = 4.0,
gap extension penalty = 0.2; раскраска ClustalX.
Далее я построила выравнивания последовательностей двух заведомо негомологичных белков. Были взяты последовательности с идентификаторами в базе данных
UniProt: B8D2N2 (НАД-зависимая эпимераза-дегидратаза) и R4T2E1 (дУТФ (дезоксиуридин-трифосфат) пирофосфатаза). Последовательности в fasta-формате: B8D2N2, R4T2E1.
Выравнивания были построены с помощью программ needle и water со стандартными параметрами. Изображения глобального и локального выравниваний последовательностей представлены
на рисунках 7 и 8 соответственно. Также выравнивания представлены в fasta-формате: needle,
water.
Рис.7. Глобальное парное выравнивание последовательностей белков с идентификаторами B8D2N2 и R4T2E1 в базе данных UniProt, полученное с помощью
программы needle с параметрами gap opening penalty = 10.0,
gap extension penalty = 0.5; раскраска ClustalX.
Рис.8. Локальное парное выравнивание последовательностей белков с идентификаторами B8D2N2 и R4T2E1 в базе данных UniProt, полученное с помощью
программы water с параметрами gap opening penalty = 10.0,
gap extension penalty = 0.5; раскраска ClustalX.
На рисунке 9 представлено сравнение выравнивания последовательностей OCEIH и GEOKA, полученное из множественного выравнивания(рис.2), с выравниванием, построенным с помощью программы
needle с параметрами gap opening penalty = 2.0, gap extension penalty = 0.5 (рис.5).
Рис.9. Сравнение парных выравниваний последовательностей OCEIH и GEOKA: верхнее, построенное с помощью программы needle с параметрами
gap opening penalty = 2.0, gap extension penalty = 0.5; нижнее, полученное из множественного выравнивания; раскраска ClustalX.
Из сравнения видно, что имеется несколько участков различия. Один из них (позиции с 97 по 109), окруженный с обеих сторон совпадающими колонками обоих
выравниваний, представлен на рисунке 10. Всего 82 различающиеся позиции.
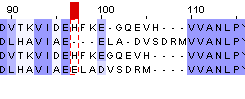 Рис.10. Участок различия сравниваемых выравниваний с 97 по 109 позиции; раскраска BLOSUM62 Score.
Рис.10. Участок различия сравниваемых выравниваний с 97 по 109 позиции; раскраска BLOSUM62 Score.
В таблице 1 представлена информация о числе и проценте консервативных колонок, колонок со сходными аминокислотными остатками, числе гэпов в выравнивании, полученном из
множественного, в выравниваниях, построенных для гомологичных последовательностей OCEIH и GEOKA при помощи программ needle и water, а также в выравниваниях, построенных
для негомологичных последовательностей.
| Выравнивание |
Длина выравнивания |
Число консервативных колонок |
Процент консервативных колонок |
Число колонок со сходными остатками |
Процент колонок со сходными остатками |
Число гэпов |
| Полученное из множественного |
270 |
156 |
57,78% |
48 |
17,78% |
1 |
| Глобальное для OCEIH и GEOKA |
270 |
156 |
57,78% |
48 |
17,78% |
1 |
| Локальное для OCEIH и GEOKA |
264 |
156 |
59,09% |
48 |
18,18% |
1 |
| Глобальное для OCEIH и GEOKA с измененными параметрами |
281 |
160 |
56,94% |
47 |
16,73% |
11 |
| Локальное для OCEIH и GEOKA с измененными параметрами |
267 |
158 |
59,18% |
47 |
29,75% |
3 |
| Глобальное для последовательностей белков с идентификаторами B8D2N2 и R4T2E1 в базе данных UniProt |
376 |
35 |
9,31% |
14 |
3,72% |
8 |
| Локальное для последовательностей белков с идентификаторами B8D2N2 и R4T2E1 в базе данных UniProt |
97 |
25 |
25,78% |
13 |
13,4% |
3 |
Данные для таблицы получены при помощи программы infoalign (EMBOSS).
По данным таблицы и по выравниваниям можно сделать некоторые выводы:
- процент консервативных колонок в выравниваниях гомологичных последовательностей OCEIH и GEOKA достигает порядка 60%, по чему можно судить о гомологичности
последовательностей, а в выравниваниях негомологичных последовательностей - колеблется от 9% до 25%;
- можно считать, что локальное выравнивание в целом лучше, чем глобальное. Наиболее значительные отличия возникают при выравнивании негомологичных последовательностей:
при глобальном выравнивании процент консервативных колонок равен 9,31%, а при локальном это значение достигает 25,78%. Это вызвано тем, что при построении
локального выравнивания гомологичные участки ищутся не во всей последовательности, а только в ее части;
- в выравниваниях негомологичных последовательностей гэпов гораздо больше, а консервативных и сходных позиций мало. Поэтому для таких
последовательностей наиболее подходит локальное выравнивание, т.к. найти случайные совпадения на небольшом участке гораздо проще;
- из таблицы 1 видно влияние изменения параметров при построении парных выравниваний. Так уменьшение штрафа за открытие гэпа в глобальном выравнивании привело к
увеличению чила гэпов. Важно отметить, что не смотря на большое количество гэпов, количество позиций, занятых гэпами, равно 23, т.к. штраф за продолжение гэпа достаточно
большой. А при изменении параметров локального выравнивания, а именно уменьшении штрафа за продолжение гэпа, появилось небольшое количество более длинных гэпов.
|