Выравнивание геномов
Задание 1. Карта сходства хромосом двух родственных бактерий.
Для выполнения задания я взяла бактерии Mycoplasma capricolum subsp. capricolum ATCC 27343
(хромосома) и Mycoplasma leachii 99/014/6
(хромосома).
Карта локального сходства отражает крупные эволюционные события, такие как вставки, делеции, транслокации, инверсии.
Она отбражает информацию о том, какой участок генома одной бактерии соответствует определенному участку генома другой бактерии.
Для построения карты локального сходства я использовала blast2seq (выравнивание 2 последовательностей), алгоритм blastn,
на сайте NCBI. Результат представлен на рис.1.
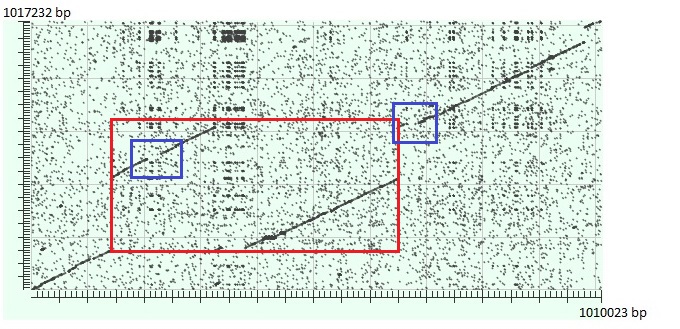 Рис.1. Карта локального сходства последовательностей хромосом Mycoplasma capricolum subsp. capricolum ATCC 27343
(по Ox) и Mycoplasma leachii 99/014/6 (по Oy). Координаты участков не указаны из-за сильной компактизации изображения (слишком длинные участки).
Рис.1. Карта локального сходства последовательностей хромосом Mycoplasma capricolum subsp. capricolum ATCC 27343
(по Ox) и Mycoplasma leachii 99/014/6 (по Oy). Координаты участков не указаны из-за сильной компактизации изображения (слишком длинные участки).
Особенности:
- На данной карте представлено выравнивание прямых последовательностей (линии идут из левого нижнего угла в верхний правый);
- Красной рамкой выделена транслокация довольно больших участков;
- Синей рамкой выделены места, где произошла вставка в хромосому Mycoplasma leachii 99/014/6 или делеция в хромосоме Mycoplasma
capricolum subsp. capricolum ATCC 27343, т.к. последовательность хромосомы Mycoplasma leachii 99/014/6
длинее последовательности хромосомы Mycoplasma capricolum subsp. capricolum ATCC 27343 примерно на 7000 п.н.
Задание 2. Описание сходства и различия геномов близкородственных бактерий.
В данном задании требовалось построить нуклеотидный пангеном (NPG) с помощью пакета NPG-explorer. Нуклеотидные
пангеном - специальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей.
Для выполнения заданий я выбрала 4 штамма бактерии Clostridium perfringens: Clostridium perfringens str. 13 (CP1),
Clostridium perfringens ATCC 13124 (CP2), Clostridium perfringens F262 (CP3).
- Описание синтеничных участков - g-блоков - и их перестановок в геномах:
Информация была взята из global-blocks/blocks.gbi. Число блоков: 22.
Информация о блоках в виде таблицы
Информация для определения последовательности глобальных блоков в каждом геноме была получена
global-blocks/blocks.blocks.
Порядок блоков в каждой хромосоме для всех геномов (выравнивание)
Рис.1. Выравнивание блоков
Из выравнивания видно, что 14 блоков у всех 3 штаммов расположены в одном месте и совпадают.
У штамма CP3 в блоках g3x4777 и g3x4091 наблюдается инверсия.
У штамма CP2 наблюдается транслокация блоков g3x101, g3x104. Также можно заметить транслокацию блоков g3x398 в штамме CP1 и g3x127 в штамме CP3.
- Описание ядра геномов - s-блоков:
s-блоки - стабильные (коровые) блоки. Информация была получена из файла
pangenome/pangenome.info.
- Число блоков: 280;
- Суммарная длина и процент от длины генома в среднем: 2685922 (72.72%);
- Сходство геномов (процент консервативных позиций в объединенном выравнивании s-блоков): 0.980141
- Описание повторов на примерах r-блоков:
r-блоки - блоки с повторами, по крайней мере, в одном геноме.
- Число блоков: 99;
- Суммарная длина и процент от длины генома в среднем: 27154 (0.73%);
- Сходство геномов: 0.94838
| Блок |
Число фрагментов |
Длина |
Процент консервативных колонок |
Число генов |
| r19x233 |
19 |
233 |
96,99% |
18 |
| r4x190 |
4 |
190 |
95% |
1 |
- Описание крупных делеций на примерах h-блоков:
h-блоки - "полустабильные" блоки - по одному фрагменту из части геномов.
| Блок |
Число фрагментов |
Длина |
Процент консервативных колонок |
Количество генов |
У кого нет |
| h2x1190 |
2 |
1190 |
96,51% |
2 |
Clostridium perfringens F262 |
| h2x1908 |
2 |
1908 |
99.71% |
4 |
Clostridium perfringens str. 13 |
| h2x7728 |
2 |
7728 |
98.86% |
15 |
Clostridium perfringens F262 |
- Описание уникальных последовательностей:
u-блоки - уникальные последовательности из одного генома, у них нет гомологов среди всех геномов, кроме самой себя.
| Блок |
Число фрагментов |
Длина |
Количество генов |
У кого есть |
Аннотация |
Горизонтальный перенос от другой бактерии |
| u1x7782 |
1 |
7782 |
3 |
Clostridium perfringens ATCC 13124 |
cell wall surface anchor family protein; sortase family protein; conserved hypothetical protein |
Горизонтальный перенос возможен, т.к. последовательность блока выровнялась на геномы других видов бактерий. Также
blast нашел вектор экспрессии pSG590_5. Похожие гены есть у плазмодиев. |
| u1x2623 |
1 |
2623 |
4 |
Clostridium perfringens ATCC 13124 |
2-dehydro-3-deoxyphosphogluconate; kinase, pfkB family; 4-deoxy-l-threo-5-hexosulose-uronate;
putative glucuronyl hydrolase |
Горизонтальный перенос возможен, т.к. последовательность блока выровнялась на геномы других видов бактерий. |
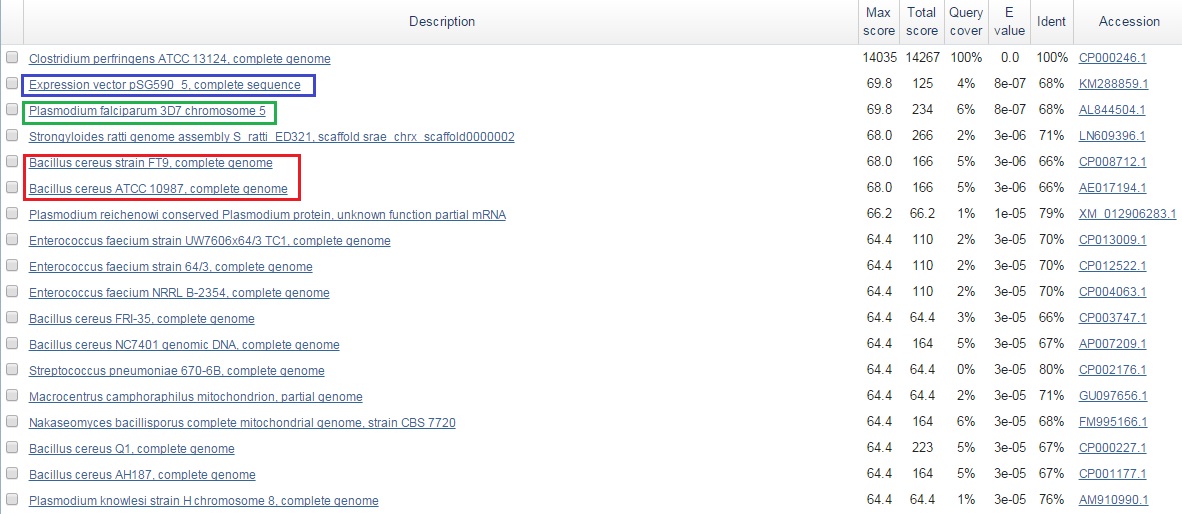 Рис.2. Результаты выдачи blastn для последовательности блока u1x7782. Красным цветом выделены бактерии других видов,
синим цветом выделен вектор экспрессии, зеленым - плазмодий.
Рис.2. Результаты выдачи blastn для последовательности блока u1x7782. Красным цветом выделены бактерии других видов,
синим цветом выделен вектор экспрессии, зеленым - плазмодий.
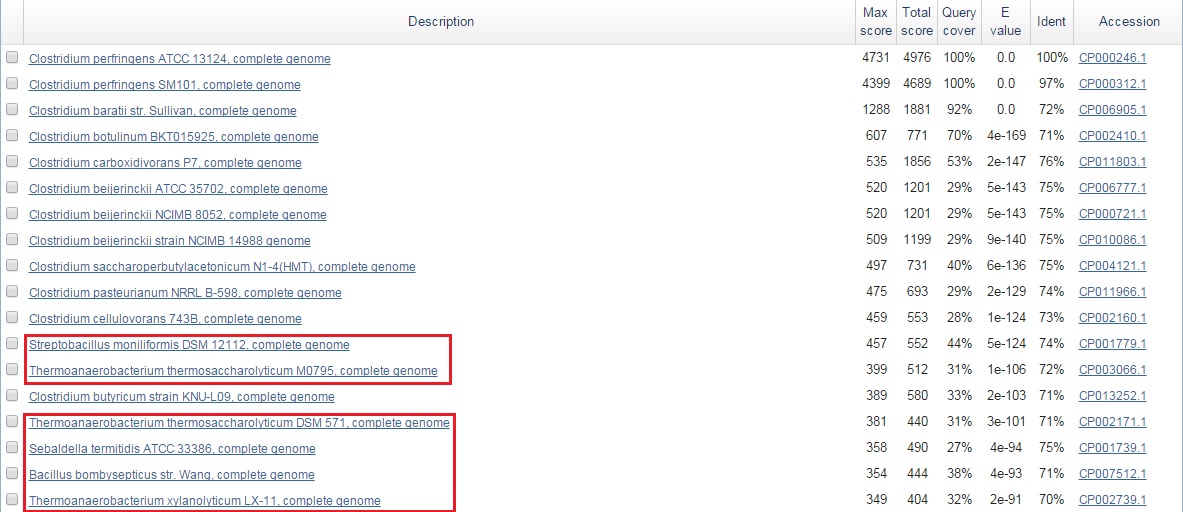 Рис.3. Результаты выдачи blastn для последовательности блока u1x2623. Красным выделены бактерии
других видов.
Рис.3. Результаты выдачи blastn для последовательности блока u1x2623. Красным выделены бактерии
других видов.
- Примеры расхождений между аннотациями генов из одного блока:
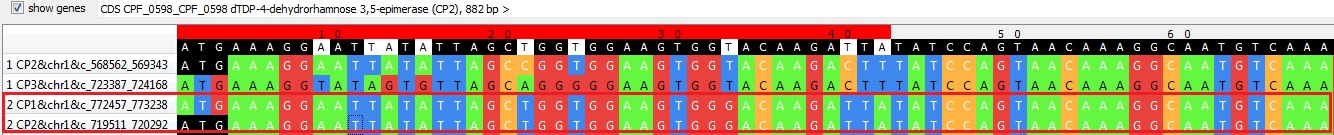
- В блоке r4x782 представлен случай, когда в одном геноме ген аннотирован, а в другом последовательность
нет (рис.4, выделены красным цветом). В геноме бактерии Clostridium perfringens ATCC 13124 последовательность является геном
dTDP-4-дегидрорамноза-3,5-эпимеразы. Та же самая последовательность в геноме бактерии Clostridium perfringens str. 13 не аннотирована.
Рис.4. Участок выравнивания последовательностей блока r4x782.
- В блоке r6x103 последовательность, показанная на рис.5, повторяется 2 раза в геноме бактерии
Clostridium perfringens ATCC 13124.
В первом случае ген кодирует ацетилтрансферазу из семейства GNAT, во втором случае - белок B, участвующий в транспорте двухвалентного железа.
Рис.5. Участок выравнивания последовательностей блока r6x103.
|