Банки нуклеотидных последовательностей
Часть I.
Задание 1. Качество сборки генома эукариотического организма.
Для этого задания я выбрала Danio rerio. Для поиска использовала NCBI Browse by organism.
Danio rerio - модельный организм для изучения развития позвоночных, эволюционной биологии и некоторых генетических заболеваний человека.
 Рис.1. Danio rerio
Рис.1. Danio rerio
- Число сборок генома: 2;
- Число проектов по секвенированию: 1;
- Число образцов: 2.
Данные были получены из соответствующей таблицы, приведенной на странице Genome Assembly and Annotation report (табл.1).
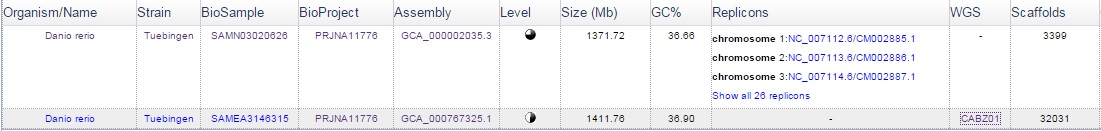 Таблица 1. Информация о количестве сборок генома, проектов по секвенированию и числе образцов.
Таблица 1. Информация о количестве сборок генома, проектов по секвенированию и числе образцов.
Далее нужно выбрать наиболее полную сборку. Как видно из таблицы, наболее полная сборка GCA_000002035.3 (колонка Level), но для нее не представлена
таблица контигов в колонке WGS. Поэтому я выбрала сборку GCA_000767325.1.
Описание образца:
- Идентификаторы: BioSample: SAMEA3146315;
- Организм: Danio rerio,вид пресноводных лучепёрых рыб семейства карповых (подробная систематика приведена на рис.2);
- Характеристика: порода Tuebingen;
- Проект: PRJNA11776 Danio rerio
- Представление результатов: EBI; 2014-11-30.
 Рис.2. Описание образца SAMEA3146315 на сайте NCBI
Рис.2. Описание образца SAMEA3146315 на сайте NCBI
Описание проекта:
- Accession: PRJNA11776 ID: 11776;
- Институт Сэнгера начал секвенирование 1,7 Gb генома Danio rerio в 2001, используя секвенирование методом дробовика (shotgun sequencing).
The Genome Reference Consortium (GRC) взял на себя техническое выполнение сборки генома Danio rerio.
Целями этой группы являются: исправить регионы, которые искажены, закрыть оставшиеся гэпы, произвести альтернативные сборки структурных вариантов
локусов.
- Тип данных проета: секвенирование генома и сборка;
- Параметры исследования:
- охват и чистота образца (Scope): популяция (Multiisolate);
- материал: геном;
- тип полученной информации: полный геном;
- метод: секвенирование;
- Данные о последовательности: количество нуклеотидов (35456), геномных ДНК (3423);
- Публикации: PubMed, PMC;
- Регистрация данных: 28-Jan-2005.
Информация о проекте представлена на рис.3.
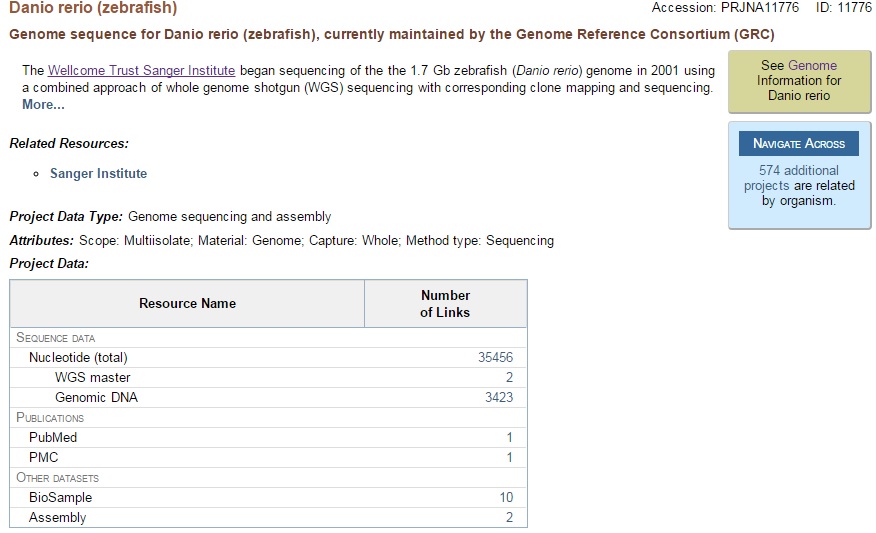 Рис.3. Информация о проекте PRJNA11776, представленная на сайте NCBI.
Рис.3. Информация о проекте PRJNA11776, представленная на сайте NCBI.
- Число контигов сборки: 119,179;
- N50 для контигов: 24,925;
- L50 для контигов: 16,539;
- Число скэффолдов сборки: 32,031;
- N50 для скэффолдов: 613,723;
- L50 для скэффолдов: 633;
- Самый длинный контиг: CABZ01060317, длина: 215,016;
- Самые короткие контиги: CABZ01021198, CABZ01083106, CABZ01088203, длина: 501;
- Таблица контигов;
- Таблица скэффолдов представлена не была;
- Ссылка на последовательность контига CABZ01000015 в формате fasta.
Задание 2. Таблица митохондриальных генов мха Racomitrium emersum.
- Запрос в БД Nucleotide (NCBI): Racomitrium emersum[ORGN] AND mitochondrion[FILT];
- Для получения информации о числе генов РНК и белков из записи с последовательностью генома я перешла на страницу описания генома
по ссылке Genome в разделе Related information. Из табл.2 видно, что число генов РНК: 27; число генов белков: 39 (всего: 66).
 Таблица 2. Информация о геноме мха Racomitrium emersum
Таблица 2. Информация о геноме мха Racomitrium emersum
- Для получения списка всех генов я перешла по ссылке Gene в разделе Related information. Ссылка на файл со списком генов, отсортированным по началу в геноме:
список геномов.
Задание 3. Описание 10 ключей, используемых в таблицах особенностей.
| Ключ |
Описание |
Пример |
| D-loop |
Петля смещения. Область в митохондриальной ДНК, в которой короткая РНК взаимодействует с одной цепью ДНК,
отстраняя комплементарную вторую цепь. Так же описывает замещение одной цепи дуплекса ДНК на другую цепь в реакции, катализируемой белком RecA |
D-loop 15424..16300
/note="control region" |
| misc_difference |
Характеристика последовательности отличается от представленной на записи и не может быть описана другим ключом |
misc_difference 534
/note="compared to genom"
/replace="t" |
| misk_feature |
Область биологического интереса, которая не может быть описана другим ключом; новая или редкая характеристика |
misc_feature 2470..2472
/gene="CENPC"
/gene_synonym="CENP-C; CENPC1; hcp-4; MIF2"
/experiment="experimental evidence, no additional details
recorded"
/note="Phosphoserine; propagated from UniProtKB/Swiss-Prot
(Q03188.2); phosphorylation site" |
| mobile_element |
Область генома, содержащая подвижный элемент |
mobile_element complement(50911..51040)
/mobile_element_type="MITE:SM3A"
|
| primer_bind |
Нековалентное связывание праймера c сайтом инициации репликации, транскрипции или обратной транскрипции |
primer_bind 7..24
/note="mt12SrRNA1363d" |
| polyA_site |
Сайт на РНК-транскрипте, к которому могут присоединяться адениновые остатки, обеспечивающие послетранскрипционное полиаденилирование |
polyA_site 3349
/gene="CENPC"
/gene_synonym="CENP-C; CENPC1; hcp-4; MIF2" |
| regulatory |
Любой участкой ДНК, который принимает участие в регуляции транскрипции или трансляции |
regulatory 3117..3122
/regulatory_class="polyA_signal_sequence"
/gene="CENPC"
/gene_synonym="CENP-C; CENPC1; hcp-4; MIF2" |
| rep_origin |
Ориджин репликации, фрагмент молекулы нуклеиновой кислоты, с которого начинается её репликация |
rep_origin 5160..5191
/note="L-strand origin of replication" |
| source |
Указывает биологический источник указанного промежутка последовательности. Этот ключ обязателен. Допускается более чем 1 источник ключа |
1..190214555 /organism="Homo sapiens"
/mol_type="genomic DNA"
/db_xref="taxon:9606"
/chromosome="4" |
| 3'UTR |
1) Участок на 3'-конце зрелого транскрипта (следующего за стоп-кодоном), который не транслируется в белок;
2) участок на 3'-конце генома РНК вируса (следующий за последним стоп-кодоном), которые не транслируется в белок
| 3'UTR complement(16555..17256)
/locus_tag="Esi_0005_0003"
|
Часть II.
Задание 4. BLASTN.
В данном задании нужно установить какому гену принадлежит последовательность,
полученная в практикуме 6, и таксономию организма. Для этого использовался BLASTN ("samewhat similar sequences") по банку nr.
На рис.4 изображены 12 первых находок. Они достоверны: E-value = 0.0, покрытие входной последовательности около 90%, процент
идентичности свыше 90% (всего находок с E-value = 0.0 47 штук).
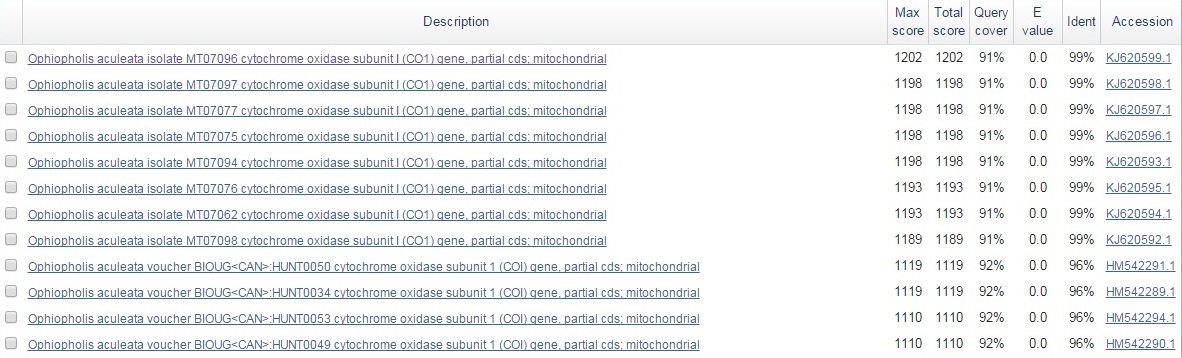 Рис.4. Результат работы blastn. Отображены первые 12 находок.
Рис.4. Результат работы blastn. Отображены первые 12 находок.
Как видно из рисунка, первые находки принадлежат к виду Ophiopholis aculeata. Можно сделать вывод, что находки представляют собой вид
Ophiopholis aculeata из рода Ophiopholis (класс Офиуры). Более подробная систематика представлена на рис.5. и на
сайте.
 Рис.5. Систематика Ophiopholis aculeata.
Рис.5. Систематика Ophiopholis aculeata.
 Рис.6. Ophiopholis aculeata.
Рис.6. Ophiopholis aculeata.
Для дальнейшей работы были выбраны первые 8 находок, процент идентичности которых составляет 99%. Все они являются последовательностями митохондриального гена,
кодирующего субъединицу 1 цитохромоксидазы. В таблице 3 представлены выравнивания выбранных последовательностей с исходной.
Проект в формате Jalview доступен по ссылке.
Выводы:
- Полученная последовательность, в практикуме 6, - митохондриальный ген субъединицы 1 цитохромоксидазы;
- Организм Ophiopholis aculeata;
- Число замен на 100 п.н. - 1 (рис.7);

- Число замен на 100 п.н. в лучшей находке из гарантированно другого вида - 14 (рис.8).

|

















