Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Эволюционные доменыЗадание 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектурой. Описание выбранного домена из Pfam:
Ссылка на страницу домена в Pfam Список доменных архитектур с выбранным доменом Выравнивание в формате fasta семейства доменов Описание выбранных доменных архитектур Архитектура 1: 499 последовательностей 
Домены: Med26, TFIIS_M, TFIIS_C Архитектура 2: 120 последовательностей 
Домены: TFIIS_M, SPOC Описание выбранного таксона В качестве таксона выбран Eukaryota. В качестве подтаксонов были выбраны Viridiplantae и Metazoa. Файл Excel содержит сводную таблицу с информацией о доменной архитектуре и таксономии (лист "Сводная таблица"). Затем нужно было выбрать около 20 представителей каждого подтаксона с каждой доменной архитектурой. Результат: лист "выбранные 20" файла Excel и проект JalView, содержащий выравнивания 4 групп последовательностей исходного домена (группы по подтаксонам и доменным архитектурам). В выравниваниях - раскраска ClustalX с порогом консервативности 20 % во всех группах. Предварительно из выравнивания были удалены пара последовательностей, которые были выровнены сильно хуже других. N- и C-концевые участки были удалены, поскольку там не было хорошего выравнивания. Задание 2. Построить филогенетическое дерево последовательностей домена. По полученному выравниванию в программе MEGA было построено филогенетическое дерево последовательностей методом максимального правдоподобия. На Рис.1 - изображение данного дерева с выделенными группами. Скобочная формула по ссылке. 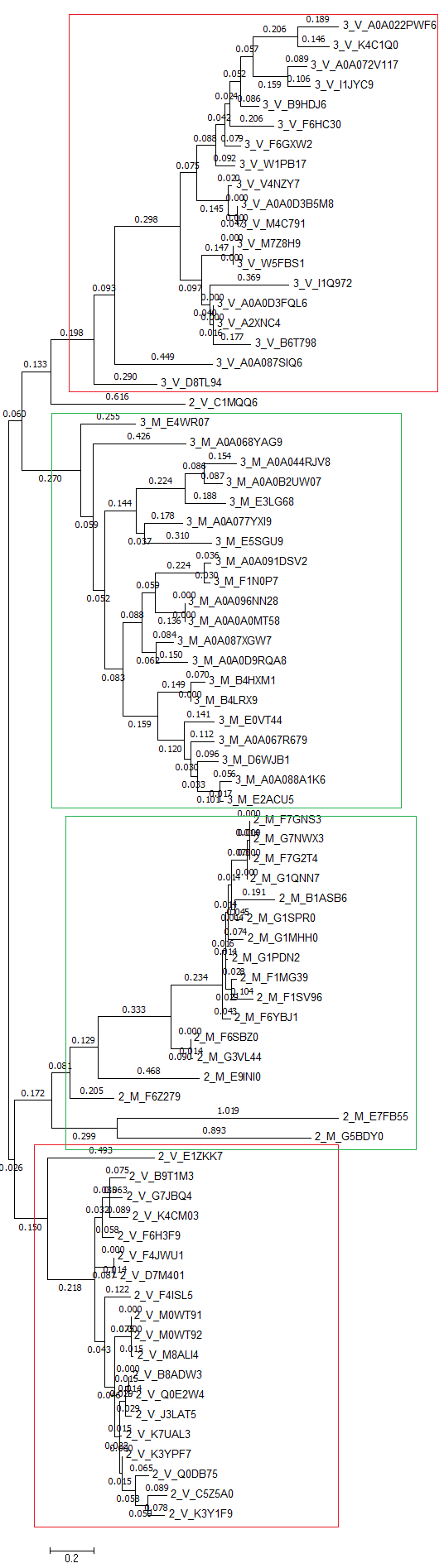
Внутри разделения по доменной архитектуре дерево хорошо разбивается на клады. Задание 3. Построить профиль подсемейства и охарактеризовать качество его работы. Для подсемейства 2_V был построен HMM-профиль (ссылка). На основе результатов поиска был получен файл Excel (на листе "Данные" содержатся данные об E-value для каждой находки и столбец с правильными находками (столбец "Результаты работы программы"). На основе этих данных была построена ROC-кривая (Рис.2). 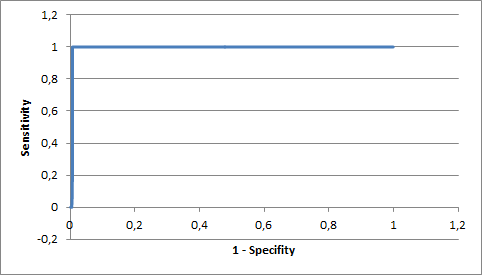
ROC-кривая получилась очень крутая. По ней был выбран порог 1,30E-26. HMM-профиль можно использовать для выделения подсемейства.
|