Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Укоренение и бутстрэпЗадание 1. Укоренение в среднюю точку Выбранные представители
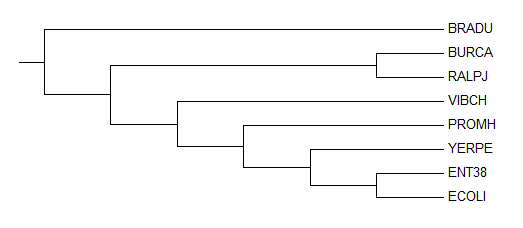
На рис.2 и 3 представлены филогенетические деревья, реконструированные по семейству белков. Для этого был выбран белок фактор элонгации трансляции Ts. Затем в программе Jalview было построено выравнивание, и было реконструировано дерево с помощью метода Neighbor Joining Using % Identity. .png)
Данный метод строит неукоренненые деревья, как видно из рис.2. В данном дереве можно найти отличие от дерева, построенного с помощью программы MEGA: листья YERPE и PROMH образуют отдельную кладу. На рис.3 представлено дерево выбранных организмов, для которого было выполнено укоренение в среднюю точку с помощью программы retree пакета PHYLIP. Полученное укоренение совпадает с укоренением дерева, полученного с помощью программы MEGA. 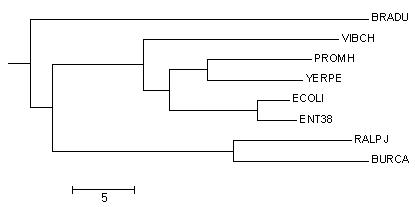
|