Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Реконструкция филогении по нуклеотидным последовательностям. Паралоги.Задание 1. Построение дерева по нуклеотидным последовательностям В данном задании требовалось построить филогенетическое дерево бактерий, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Последовательности 16S рибосомальной РНК каждой из бактерий были получены из базы полных геномов NCBI ( ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/), из файлов с расширением frn, которые содержат последовательности всех РНК.
Полученные последовательности были выровнены методом Muscle. Далее в программе MEGA было реконструировано дерево методом Maximum Likelihood. 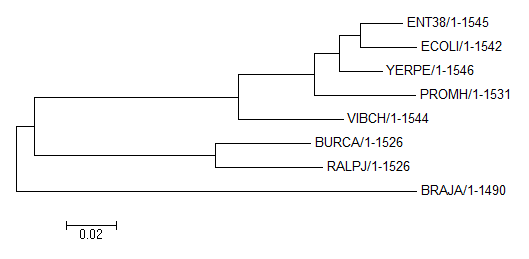
Полученное дерево совпадает с правильным (практикум 1). Задание 2. Построение и анализ дерева, содержащего паралоги В данном задании требовалось найти в выбранных бактериях гомологов белка CLPX_ECOLI. Для этого был проведен поиск программой blastp с E-value 0.001. Затем было получено выранивание последовательностей белков методом Muscle, и реконструировано дерево полученных гомологов методом Neighbor-Joining. 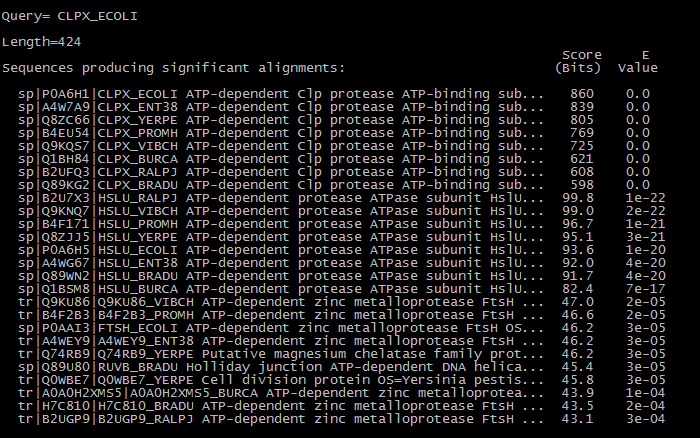
Белки, встретившиеся в выдаче blast:
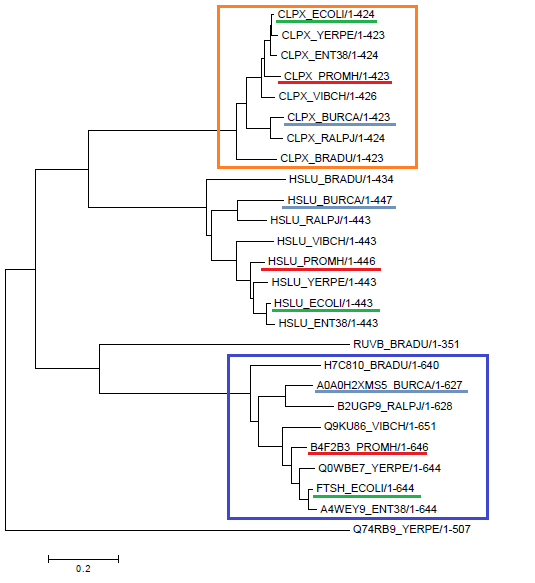
Паралогами называются два гомологичных белка из одного организма (красным выделены все гомологи Proteus mirabilis, которые попарно являются паралогами; аналогично зеленым цветом выделены все гомологи Escherichia coli и голубым - гомологи Burkholderia cenocepacia). Ортологи - два гомологичных белка из разных организмов, и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. На рис.3 синим и оранжевым цветами выделены группы попарно ортологичных белков. Также на дереве отражены эволюционные события:
|