Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
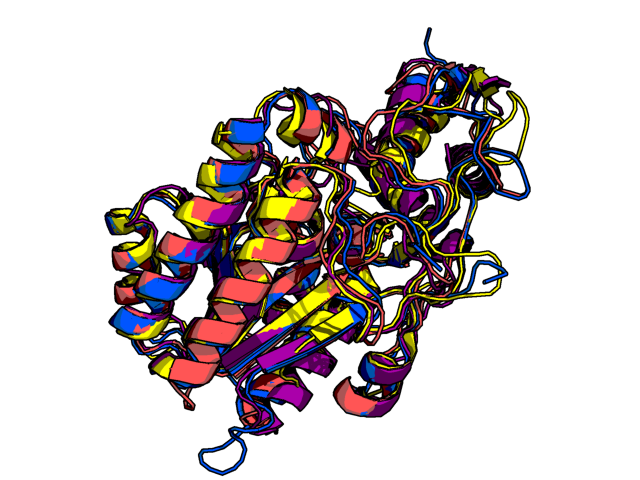
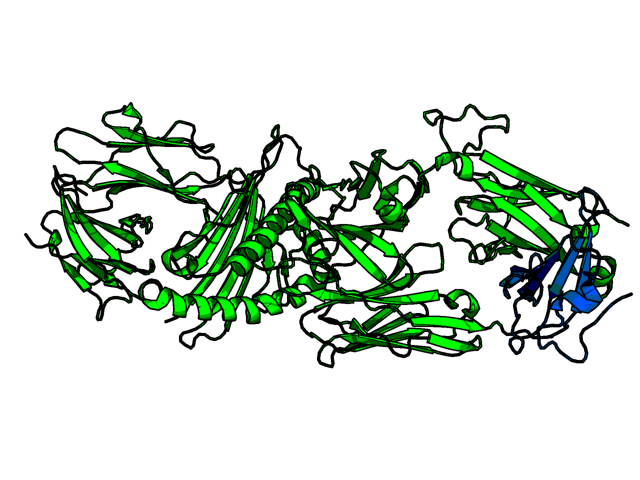
Совмещение структурПоиск структурных гомологов С помощью сервиса PDBeFold был произведен поиск структурных гомологов белка GalE из археи Pyrobaculum calidifontis JCM 11548 (PDB 3KO8). Были выбраны 3 гомолога из разных организмов с RMSD между 0,8 и 2,5 и длиной выравнивания более 50% от длины белка GalE: Thermus thermophilus HB8 (цепь А, PDB 2p5y), Homo sapiens (цепь B, PDB 1EK6), Trypanosoma brucei (цепь D, PDB 1GY8). На рис. 1 показано полученное совмещение структур.
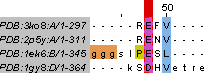
На рис. 2 представлено полученное структурное выравнивание. 
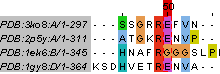
Было построено выравнивание последовательностей программой Muscle с параметрами по умолчанию (рис. 3). 
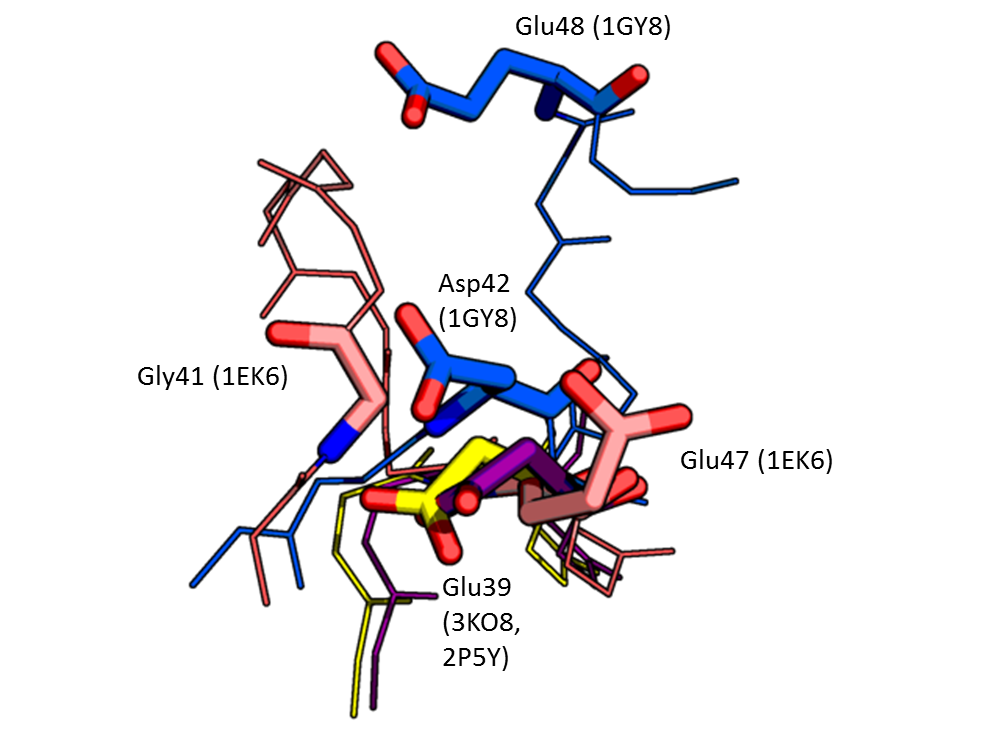
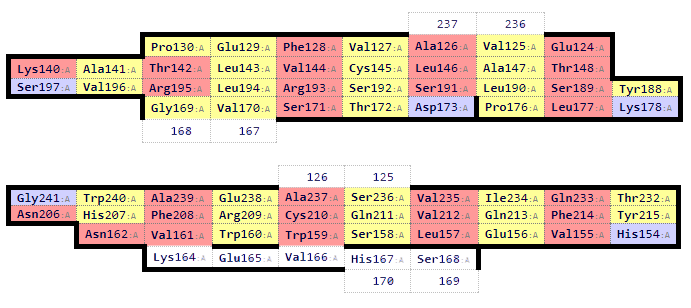
Полученные выравнивания отличаются. Один из примеров приведен на рис. 4, остатки из разных белков выровнены по структуре, но не выровнены программой Muscle. На рис. 5 представлено пространственное расположение данных остатков. Видно, что остатки, которые предлагает программа, находятся далеко от остатков из той же колонки, поэтому выравнивание по структуре можно считать более точным.
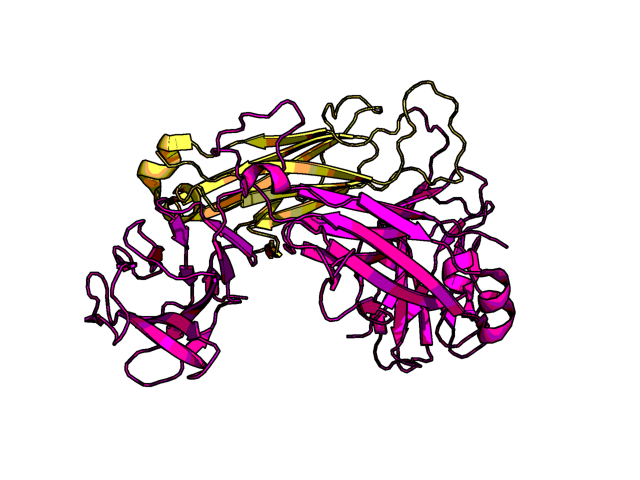
Совмещение по заданному выравниванию Для задания были выбраны 2 структуры константного домена T-клеточного рецептора: из цепи α - 2BNQ (участок D: 115 - 204), из цепи β - 2AXH (участок A: 119 - 244). На рис. 6-7 показаны выбранные структуры и участки.
С помощью сервера SheeP были получены карты β-листов для выбранных участков (рис. 8-9).
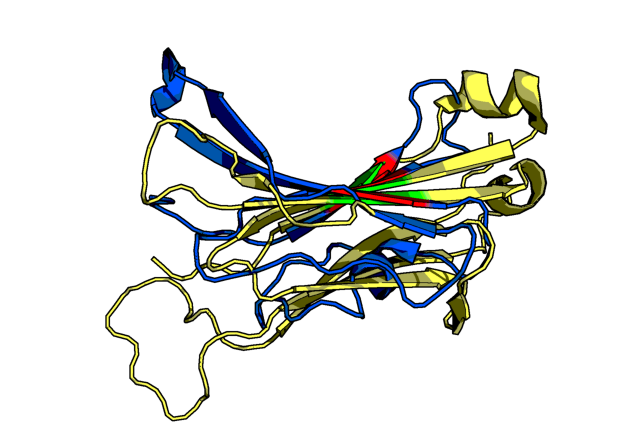
В каждой карте был найден консервативный остаток цистеина: Cys137 в цепи α и Cys145 в цепи β. Эти остатки были использованы для выравнивания. На рис. 10 показано полученное выравнивание. Файл с совмещением можно скачать по ссылке. Команды: select alpha, 2bnq and chain D and resi 124-126+136-138+177-179 and name CA select beta, 2axh and chain A and resi 126-128+144-146+191-193 and name CA pair_fit alpha, beta
Из рис. 10 видно, что направление и расположение β-тяжей совпадают, но не все элементы вторичной структуры совпадают, поэтому говорить о сходстве топологий нельзя. |