TRNA(GLU)
GLUTAMYL-TRNA SYNTHETASE
из организма THERMUS THERMOPHILUS.
Для исследования была выбрана цепь C, представляющая glutamyl-тРНК со следующей последовательностью:
[501] ggccccaucgucuagcgguaggacgcggcccucucaaggccgaaacggggguucgauucccccuggguucacca [576]
В последовательности на 3'-конце имеется триплет CCA, к которому присоединяется аминокислота. (координаты можно посмотреть в pdb-файле)
* Акцепторный стебель состоит из участка 501-507 и комплементарного ему участка 566-572.
* T-стебель состоит из участка 549-553 и комплементарного ему участка 561-565.
* D-стебель состоит из участка 510-513 и комплементарного ему участка 522-525.
* Антикодоновый стебель состоит из участка 539-543 и комплементарного ему участка 527-531.
RasMol
Был создан скрипт для программы RasMol, который отображает акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым.Скрипт: stem.spt
Файл PDB, на котором следует запускать скрипт: TRNA.PDB (только РНК)
|
|
define stem_acceptor 501-507,566-572 define stem_t 549-553, 561-565 define stem_d 510-513, 522-525 define stem_anticodon 539-543, 527-531 background white select all wireframe off cpk off backbone 100 select stem_acceptor color red select stem_t color green select stem_d color blue select stem_anticodon color orange select not (stem_acceptor, stem_t, stem_d, stem_anticodon) color grey |
Можно отметить также
a) Вариабельная петля, судя по всему, отсутствует.
b) В Т-петле остатки тимидина отсутствуют.
c) В D-петле дигидроуридины отсутствуют.
*Дополнительное задание. Похоже, что антикодон составляют [534]-CUC-[535]
Неканонические пары
Неканоническая пара U-G (513-522)7 (0.004) C:.507_:[..A]Ax----U[..U]:.566_:C (0.006)
8 (0.004) C:.549_:[..G]G-----C[..C]:.565_:C (0.002)
принадлежащих концу акцепторного стебля и началу T стебля соответственно.
7 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.56( 0.89) 2.56( 0.89)
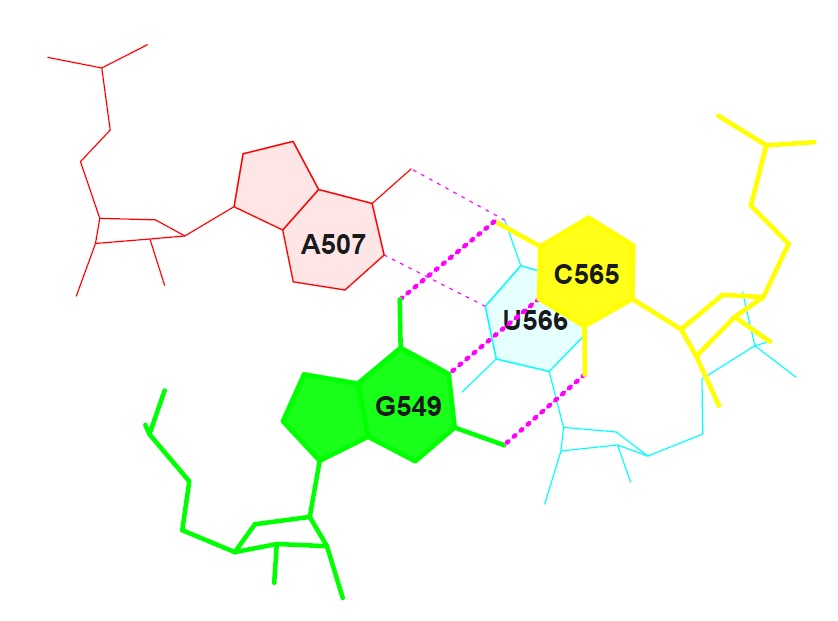
Две найденные пары не сильно перекрываются (всего 2.56 А), таким образом вероятность появления стекинг-взаимодействия оценивается как низкая.
2. Дополнительная связь между основаниями D и T петель:
28 (0.004) C:.519_:[..G]Gx---xC[..C]:.556_:C (0.002)
Запустим программу mfold по последовательности. При запуске программы можно изменять значение параметра P.
Этот параметр устанавливает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального.
В моем случае это оказалось не принципиально, поскольку даже при задании P=1 было получено вполне реальное изображение, а повышение P дало только дополнительные варианты менее похожие на реальность:
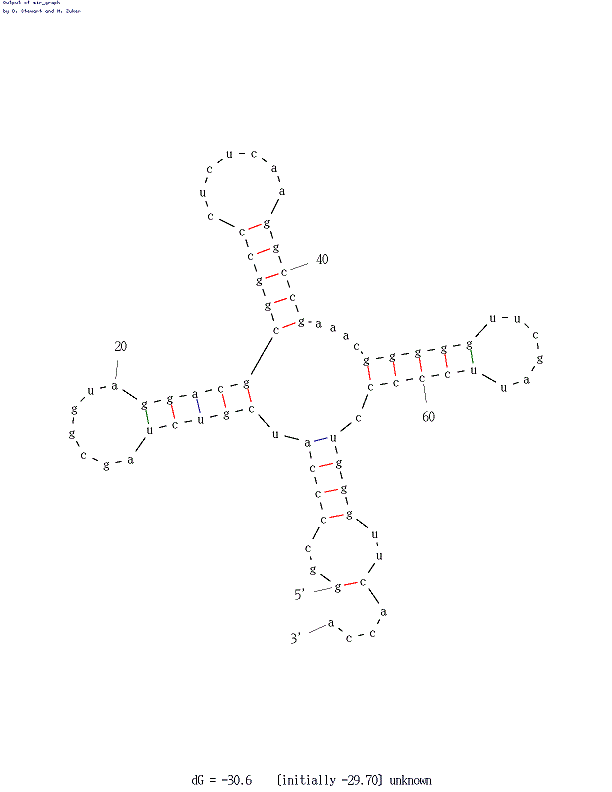
В противном случае пришлось бы постепенно повышать значение P до получения приемлемого изображения.
| Участок структуры | Позиции в структуре (по результатам mfold / find_pair) | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель |
5' 501-507 3' / 501-507
5' 564-570 3' / 566-572 Всего 5 пар (2 пары выпали) | Предсказано 5 пар из 7 реальных (со сдвигом) |
| D-стебель |
5' 509-513 3' / 510-513
5' 521-525 3' / 522-525 Всего 5 пар | Предсказано 5 пары из 4 реальных |
| T-стебель |
5' 547-551 3' / 549-553
5' 558-62 3' / 561-565 Всего 5 пар | Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель |
5' 538-542 3' / 539-543
5' 526-530 3' / 527-531 Всего 5 пар | Предсказано 5 пар из 5 реальных |
| Общее число канонических пар нуклеотидов | 19 | 18 |
Таким образом, mfold почти правильно предсказал вторичную структуру тРНК, если не считать произошедшего сдвига.
Торсионные углы
| &alpha P-O5’ | &beta O5’-C5’ | &gamma C5’-C4’ | &delta C4’-C3’ | &epsilon C3’-O3’ | &xi O3’-P | &chi C1’-N | |
| тРНК 1ffy | -78,12 | 175,59 | 69,83 | 85,46 | -150,32 | -76,28 | -159,71 |
| ДНК A | -62 | 173 | 52 | 88 / 3 | 178 | -50 | -160 |
| ДНК B | -63 | 171 | 54 | 123 / 131 | 155 | -90 | -117 |
Вывод: по торсионным углам данная тРНК больше похожа на A-форму ДНК, чем на B