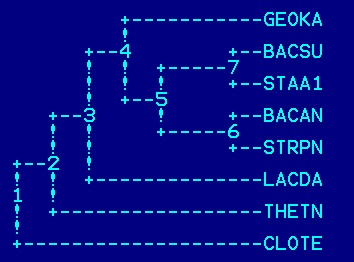
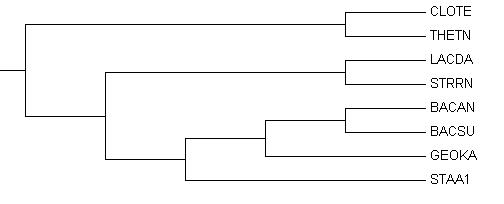
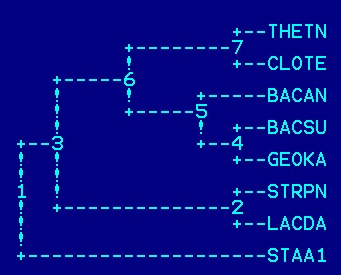
| Мнемоника | AC записи EMBL | Координаты FT, в которой описано 16S рРНК |
| BACSU | CP002468 | 2221825..2223383 |
| BACAN | AE017334 | 9335..10841 |
| CLOTE | AE015927 | 176113..177621 |
| STAA1 | AP009324 | 531922..533476 |
| LACDA | CP000412 | 43705..45265 |
| GEOKA | BA000043 | 10421..11973 |
| THETN | AE008691 | 53858..55384 |
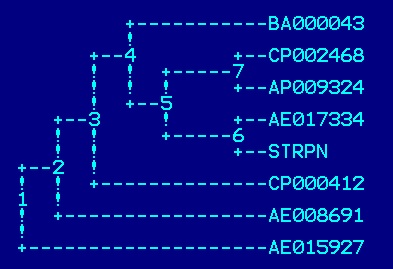 ==>
==>