Создание репрезентативной выборки гомологов белка MANB1_BACSU
С помощью программы BLAST был проведен поиск гомологов белка MANB1_BACSU среди эу- и прокариот отдельно. Критерием отбора хитов служило то, что гомология с исследуемым белком не должна быть чрезмерно высокой, чтобы выравнивание было достаточно информативным. Параметры поиска были таковыми (Табл.1):
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
|---|---|---|---|---|---|
| По прокариотам | blastp | Reference proteins | Exclude "Eukaryotes" + "Firmicutes" | 0.001 | 500 |
| По эукариотам | blastp | Reference proteins | "Eukaryotes" | 1 | 100 |
В итоге нашлись 188 гомологов среди прокариот (7 из них архейные) и 22 гомолога среди эукариот. На рис.1,2 представлены филогенетические деревья гомологов, найденных в результате поиска:
 Рис.1 Филогенетическое дерево хитов среди эукариот
Рис.1 Филогенетическое дерево хитов среди эукариот
 Рис.2 Филогенетическое дерево хитов среди прокариот
Рис.2 Филогенетическое дерево хитов среди прокариот
Из них были отобраны 29 прокариотических и 22 эукариотических хитов. Количество эукариотических хитов ограничивалось порогом e-value равным единице. Результаты приведены в табл.2:
| Archaea | ||
|---|---|---|
| Филум | Количество белков | Название организма |
| Euryarchaeota | 3 | Methanospirillum hungatei JF-1 |
| Halalkalicoccus jeotgali B3 | ||
| Halococcus hamelinensis 100A6 | ||
| Bacteria | ||
| Филум | Количество белков | Название организма |
| Proteobacteria | 3 | Cystobacter fuscus |
| Chondromyces apiculatus | ||
| Corallococcus coralloides DSM 2259 | ||
| Fibrobacteres | 2 | Fibrobacter succinogenes subsp. succinogenes S85 |
| Dictyoglomi | 2 | Dictyoglomus turgidum DSM 6724 |
| Dictyoglomus thermophilum H-6-12 | ||
| Chloroflexi | 2 | Herpetosiphon aurantiacus DSM 785 |
| Cyanobacteria | 2 | Gloeocapsa sp. PCC 7428 |
| Cyanothece sp. PCC 7425 | ||
| Actinobacteria | 4 | Micromonospora aurantiaca ATCC 27029 |
| Streptomyces venezuelae ATCC 10712 | ||
| Cellulomonas flavigena DSM 20109 | ||
| Stackebrandtia nassauensis DSM 44728 | ||
| Verrucomicrobia | 2 | Opitutus terrae PB90-1 |
| Bacteroidetes | 5 | Bacteroides fragilis 638R |
| Niastella koreensis GR20-10 | ||
| Bacteroides xylanisolvens XB1A | ||
| Solitalea canadensis DSM 3403 | ||
| Fibrella aestuarina BUZ 2 | ||
| Spirochaetes | 2 | Treponema saccharophilum |
| Planctomycetes | 1 | Phycisphaera mikurensis NBRC 102666 |
| Ignavibacteria | 1 | Melioribacter roseus P3M |
| Eukaryotes | ||
| Царство | Количество белков | Название организма |
| Animals | 1 | Strongylocentrotus purpuratus |
| Fungi | 21 | Leptosphaeria maculans JN3 |
| Podospora anserina S mat+ | ||
| Verticillium albo-atrum VaMs.102 | ||
| Sordaria macrospora k-hell | ||
| Chaetomium globosum CBS 148.51 | ||
| Myceliophthora thermophila ATCC 42464 | ||
| Aspergillus nidulans FGSC A4 | ||
| Aspergillus flavus NRRL3357 | ||
| Aspergillus oryzae RIB40 | ||
| Botryotinia fuckeliana B05.10 | ||
| Penicillium chrysogenum Wisconsin 54-1255 | ||
| Aspergillus niger CBS 513.88 | ||
| Puccinia graminis f. sp. tritici CRL 75-36-700-3 | ||
| Botryotinia fuckeliana B05.10 | ||
| Neurospora crassa OR74A | ||
| Sclerotinia sclerotiorum 1980 | ||
Множественное выравнивание гомологов белка MANB1_BACSU
 Рис.3 Множественное выравнивание гомологов белка MANB1_BACSU
( полный размер изображения )
Рис.3 Множественное выравнивание гомологов белка MANB1_BACSU
( полный размер изображения )
Выравнивание доступно в формате .jar, а также в формате .fasta
Результаты анализа множественного выравнивания гомологов белка MANB1_BACSU
Учитывая то, что в выравнивании использовались последовательности эукариотических организмов из сильно ограниченной выборки с высоким значением e-value, и не проводилось улучшения выравнивания, консервативность выравниваия довольно высока. Хотя некоторые блоки просто не имеют смысла.
За небольшим исключением, все вторичные структуры соотносятся с консервативными участками. Самое плохое выравнивание наблюдается в начальном участке - там, где находится сигнальная последовательность белка, что не удивительно, поскольку у каждого организма процесс сортинга (пост-/котрансляционный транспорт) может сильно различаться, соответственно и сигнальная последовательность.
В выравнивании присутстут большое количество паралогов.
Лишь незначительная часть вторичных структур приходятся на колонки гэпов - всего один бета-тяж. Большинство же соответсвует выделенным мной блокам, которым соответствуют колонки выровненных участков последовательностей с минимальным количеством гэпов.
Что касается остатков, связывающих лиганд, то четкой консервативности ни в одном из них не наблюдается. Возможно, это вызвано тем, что лигады, представленные в pdb структуре на самом деле являются артефактами, как, например, В-цепь данного белка, являющаяася результатом неправильной
обработки данных рентгеноструктурного анализа.
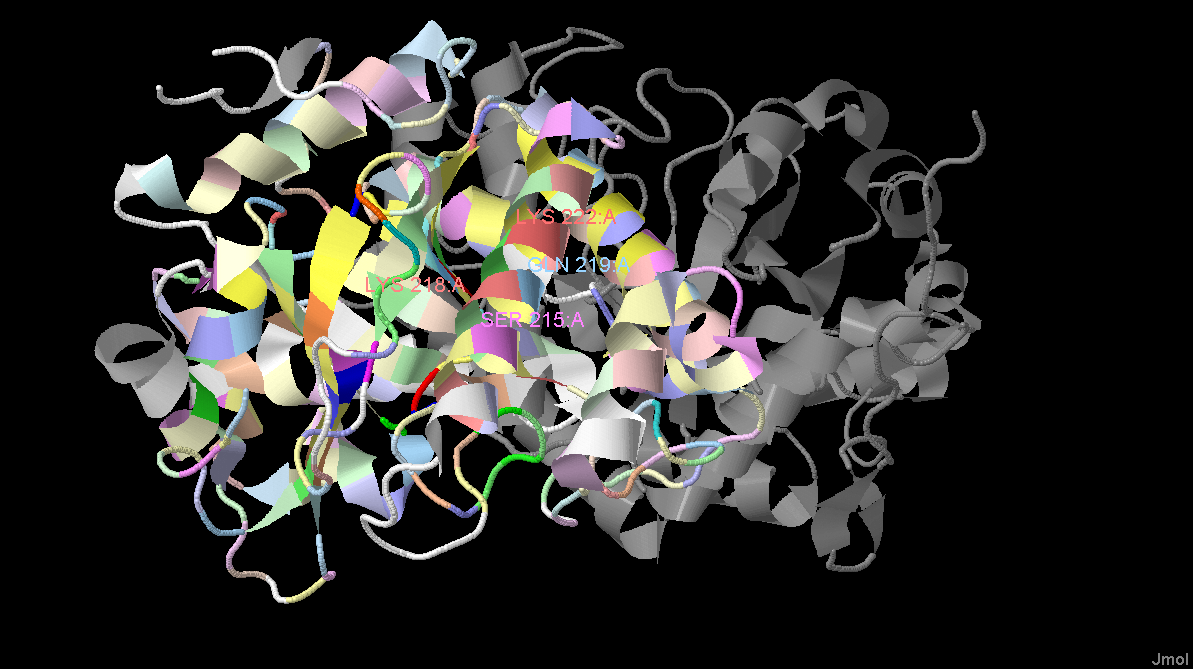 Рис.4 Трехмерная структура исследуемого белка. Показаны номера остатков, связывающих
лиганд ( полный размер изображения )
Рис.4 Трехмерная структура исследуемого белка. Показаны номера остатков, связывающих
лиганд ( полный размер изображения )