Задание №1
В задании требовалось c помощью команды define программы JMol задать следующие множества атомов в ДНК-белковом комлексе структуры 1LQ1:
1. Множество атомов кислорода 2'-дезоксирибозы (set1);
2. Множество атомов кислорода в остатке фосфорной кислоты (set2);
3. Множество атомов азота в азотистых основаниях (set3).
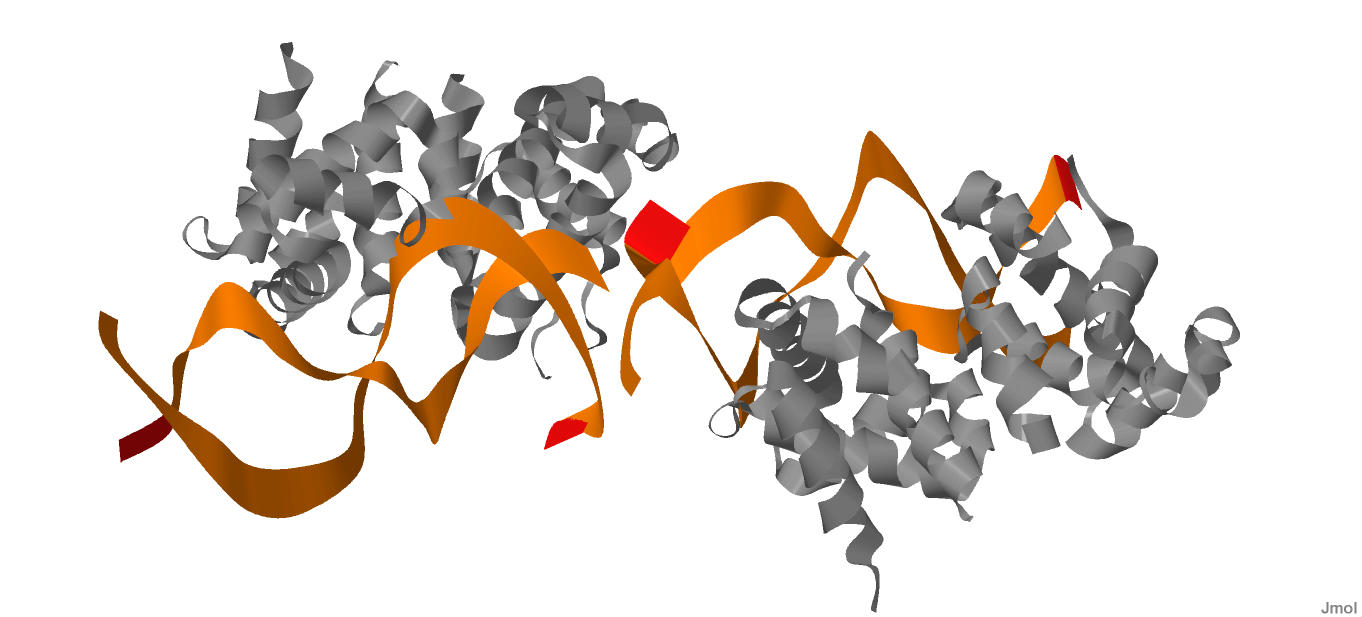 Рисунок 1. Весь ДНК-белковый комплекс в модели "ribbons".
Рисунок 1. Весь ДНК-белковый комплекс в модели "ribbons".
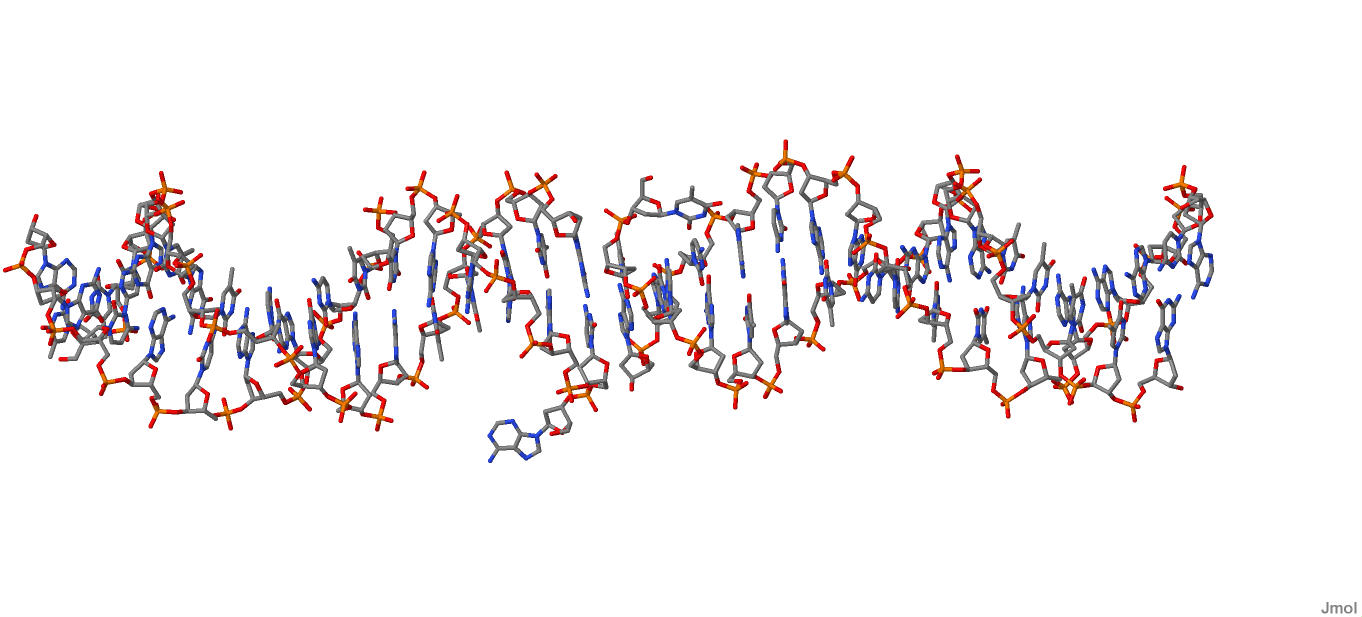 Рисунок 2. ДНК.
Рисунок 2. ДНК.
 Рисунок 3. Множество атомов кислорода 2'-дезоксирибозы (set1)
Рисунок 3. Множество атомов кислорода 2'-дезоксирибозы (set1)
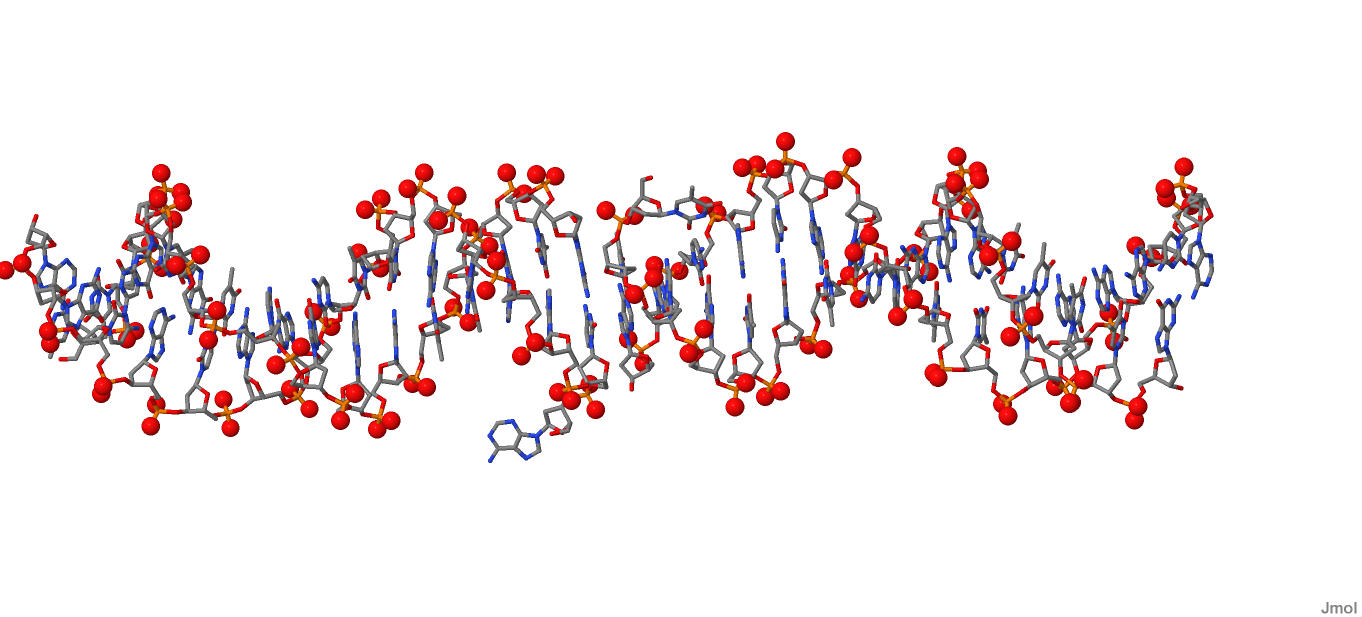 Рисунок 4. Множество атомов кислорода в остатке фосфорной кислоты (set2)
Рисунок 4. Множество атомов кислорода в остатке фосфорной кислоты (set2)
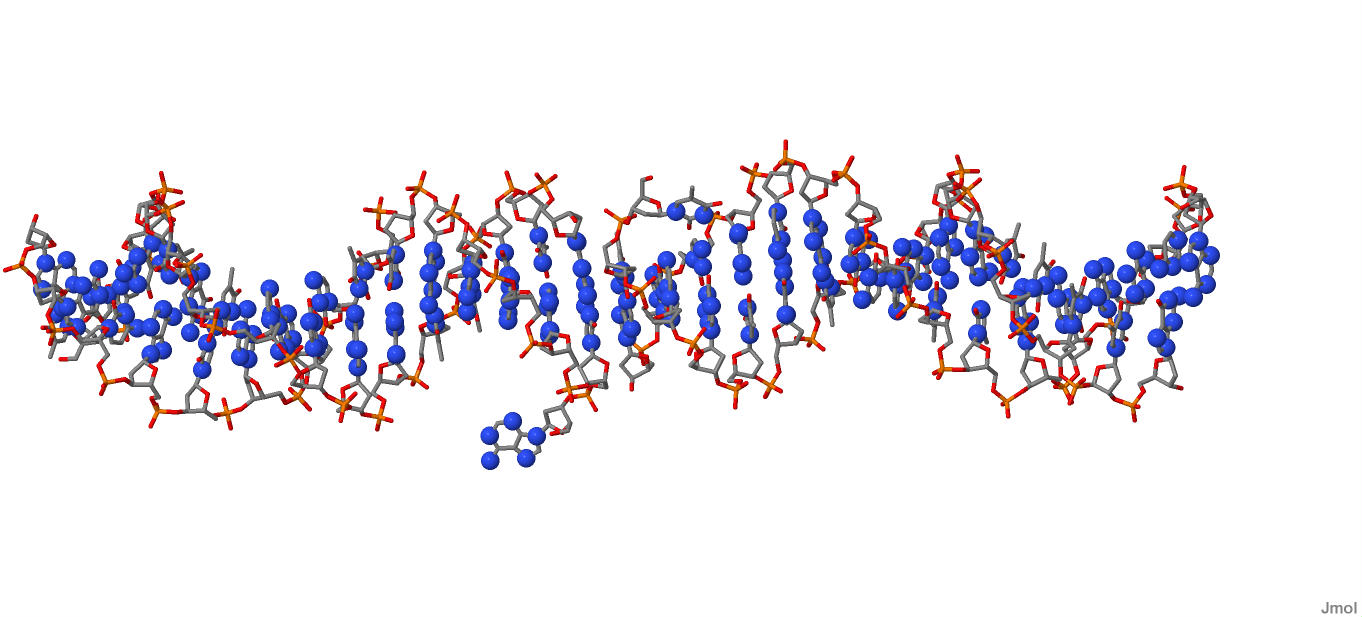 Рисунок 5. Множество атомов азота в азотистых основаниях (set3)
Рисунок 5. Множество атомов азота в азотистых основаниях (set3)
Скрипт для для просмотра изображений в JMol
Упражнение №2
Контакты атомов белка с |
Полярные |
Неполярные |
Всего |
остатками 2'-дезоксирибозы |
3 |
48 |
51 |
остатками фосфорной кислоты |
35 |
0 |
35 |
остатками азотистых оснований со стороны большой бороздки |
15 |
37 |
52 |
остатками азотистых оснований со стороны малой бороздки |
0 |
8 |
8 |
Как нетрудно заметить, неполярных контактов больше. Наиболее часто атомы белка контактируют с остатками 2'-дезоксирибозы и с остатками азотистых оснований со стороны большой бороздки.
Упражнение №3
Схема ДНК-белковых контактов, полученная с помощью программы nucplot
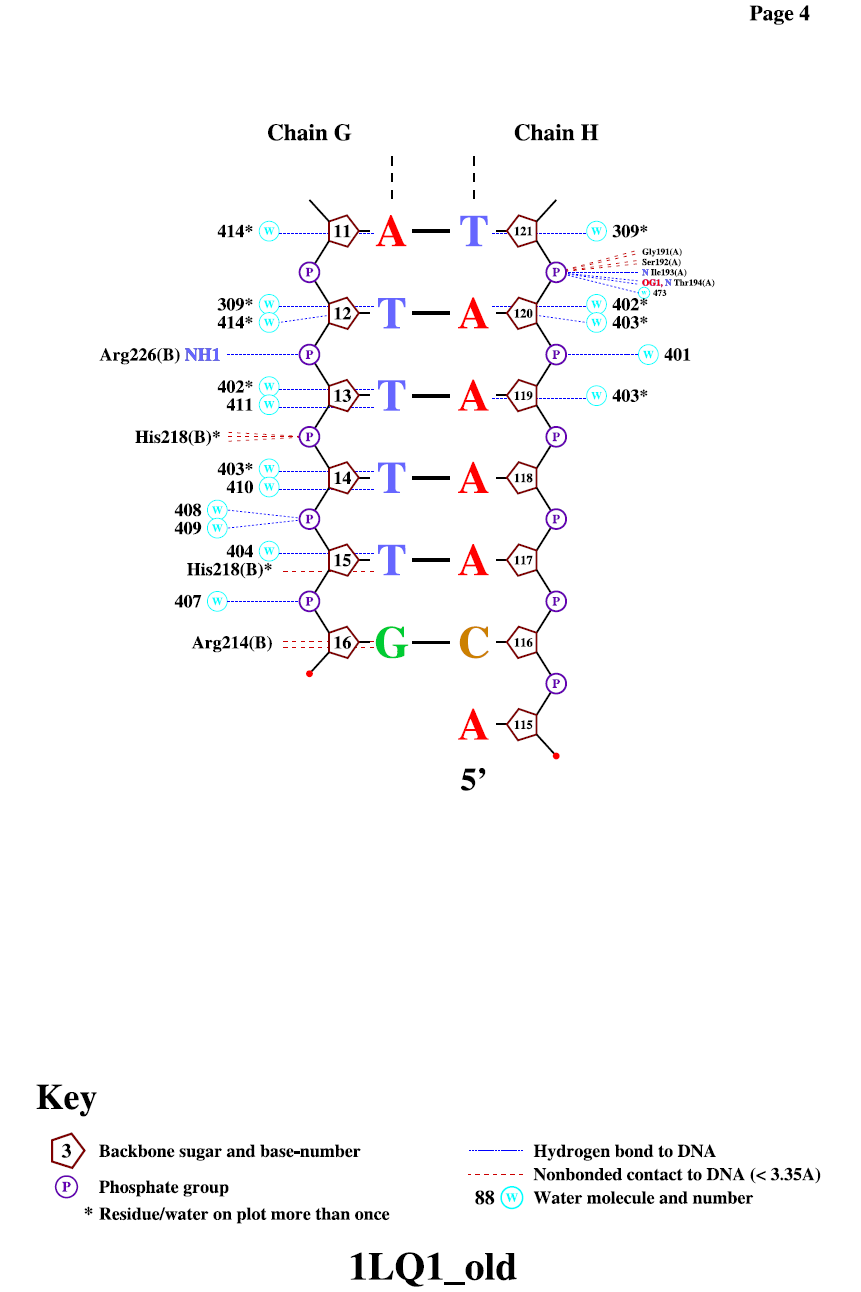 Рисунок 6. Схема ДНК-белковых контактов.
Рисунок 6. Схема ДНК-белковых контактов.
Упражнение №4
На основе полученной схемы нужно выбрать:
1. Аминокислотный остаток с наибольшим числом указанных на схеме (рис. 6) контактов с ДНК:
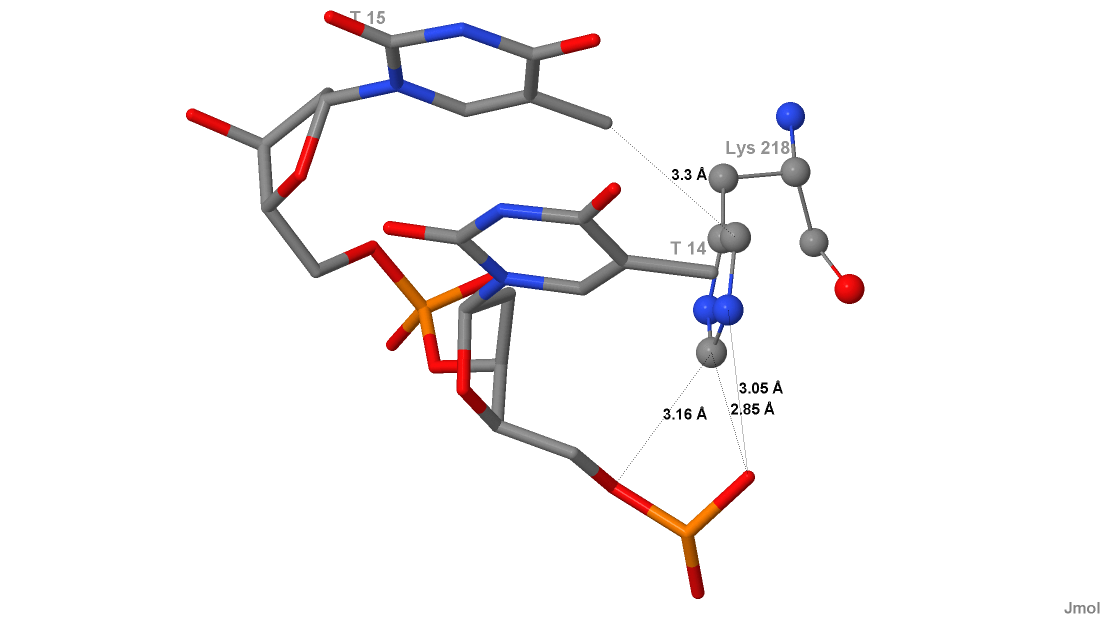 Рисунок 7. Гистидин 218 имеет 4 контакта с ДНК.
Рисунок 7. Гистидин 218 имеет 4 контакта с ДНК.
2. Аминокислотный остаток наиболее важный для распознавания последовательности ДНК:
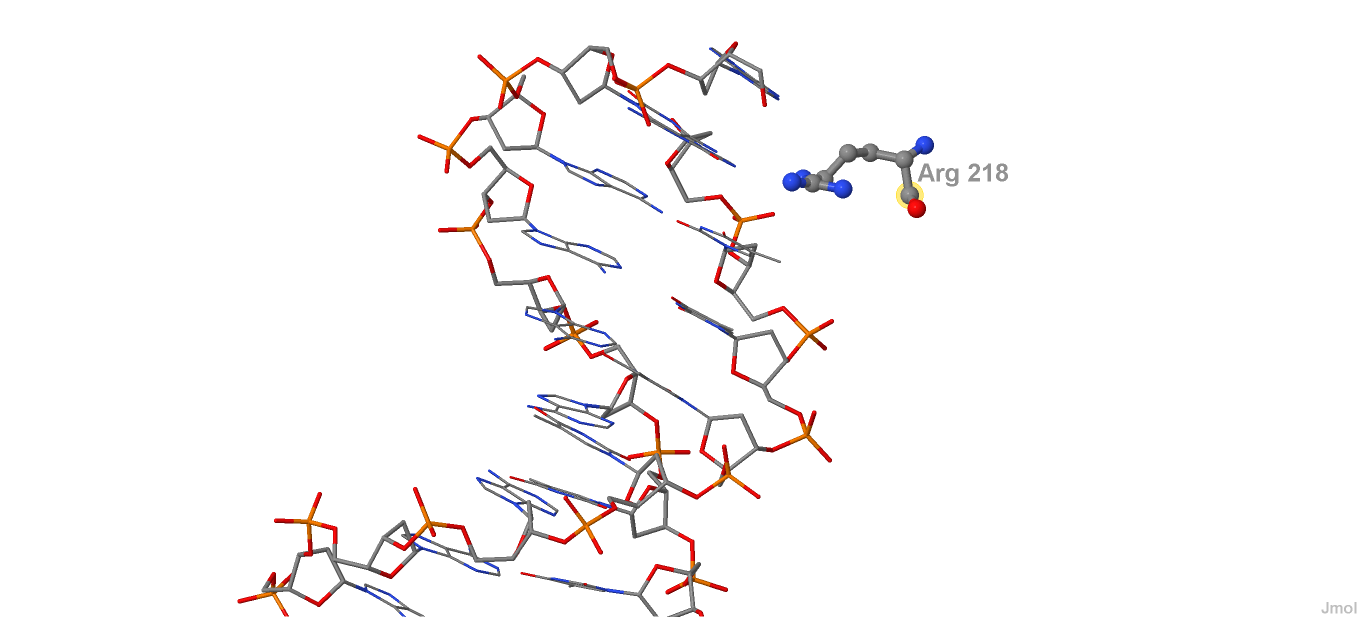 Рисунок 8. Аргинин 214. Мне кажется, что именно этот остаток наиболее
важен в распознавании последовательности ДНК, поскольку:
Рисунок 8. Аргинин 214. Мне кажется, что именно этот остаток наиболее
важен в распознавании последовательности ДНК, поскольку:1. Во-первых, он контактирует с большой бороздкой ДНК;
2. Во-вторых, хотя и на схеме показано, что образуемые им связи не водородные, тем не менее, его гуано-группа, обращенная в сторону большой бороздки ДНК, неизбежно будет образовывать с ней водородные связи, которыми и распознается последовательность ДНК.
Задание №2
Упражнение №1
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
При стандартных установках программа не находила комплементарных пар, но задав Minimum score threshold = 20, был получен файл sequence.inv. Найдены акцепторный и антикодоновый стебли. Акцепторный почти полностью совпадает с реальным, а у антикодонового стебля основания смещены на две позиции относительно реального.
Упражнение №2
Предсказание вторичной структуры тРНК по алгоритму Зукера
Воспользумся веб-версией программы mfold. Из дополнительных опций выберем Р=15 и дополним выдачу изображением в формате .gif:
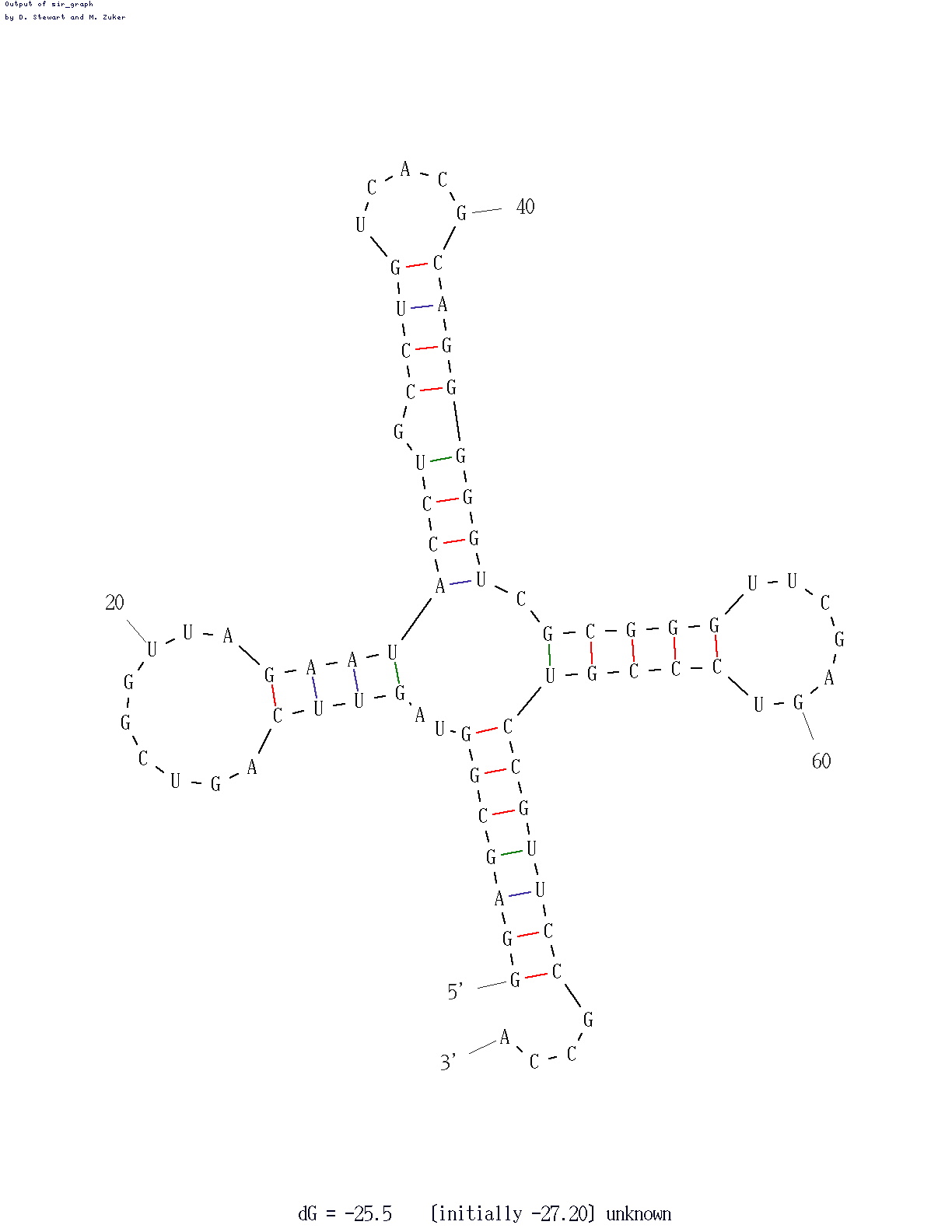 Рисунок 9. Изображение тРНК, полученное программой mfold.
Рисунок 9. Изображение тРНК, полученное программой mfold.
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
5'-601-607-3' |
Предсказано 6 пар из 7 реальных |
7 пар (6 канонических) |
D-стебель |
5'-610-613-3' |
Предсказано 0 пар из 4 реальных |
4 пары (3 канонические) |
T-стебель |
5'-649-653-3' |
Предсказано 0 пар из 5 реальных |
5 пар (4 канонические) |
Антикодоновый стебель |
5'-638-644-3' |
Предсказано 0 пар из 7 реальных |
8 пар (7 канонических) |
Общее число канонических пар нуклеотидов |
18 |
13 |
20 |