Программа getorf пакета EMBOSS
Выполним команду getorf d89965.entret -minsize 30 -find 1, чтобы получить набор трансляций всех открытых рамок последовательности, данной в записи D89965 банка EMBL, которые определены при использовании стандартного кода и одновременно удовлетворяют следующим условиям:
- длина более 30 аминокислотных остатков;
- начинаются со старт-кодона (или начала последовательности) и заканчиваются стоп-кодоном (или концом послеледовательности).
Из всех найденных рамок кодирующей последовательности, приведенной в поле FT записи EMBL соответствует прямая рамка D89965_3
Создав файл с последовательностью записи Swiss-Prot, на которую ссылается данная запись EMBL, выясним, какой из полученных открытых рамок соответствует эта последовательность. Это рамка D89965_5, причем обратная.
Чем же вызвано это противоречие? Дело, возможно, в следующем. Авторы записи EMBL занимилась поиском белка в желудке крысы с определенными параметрами. После того, как они заметили активность белка со свойствами, похожими на свойства искомого, путем гомогенизации желудка и стандартных молекулярно-биологических методов выделили и секвенировали мРНК, соответствующее этому белку. Однако до этого уже была известна последовательность подобного белка. Запись о ней хранилась в банке Swiss-Prot. Но белок принадлежал бактерии E. coli. После этого выяснилось, что белок, полученный из желудка крысы принадлежал на самом деле косполиту E. coli. Тем самым, получается, что авторы проаннотировали найденный им белок неверно, чем и вызвано полученное в ходе нашей работы противоречие.
Файлы-списки
Запущенные команды и их параметры:
seqret sw:adh*_* adh.fasta
infoseq adh.fasta -only -usa > adh.infoseq
grep -f pattern.txt adh.infoseq > adh_mi.infoseq
grep -f pattern.txt adh.infoseq > adh_my.infoseq
seqret @adh_my.infoseq mysequences.fasta
Файл с последовательностями моих алкогольдегидрогеназ: mysequences.fasta
EnsEMBL
При поиске по последовательности через BLAST/BLAT открывается изображение положения гена на соответствующей хромосоме:
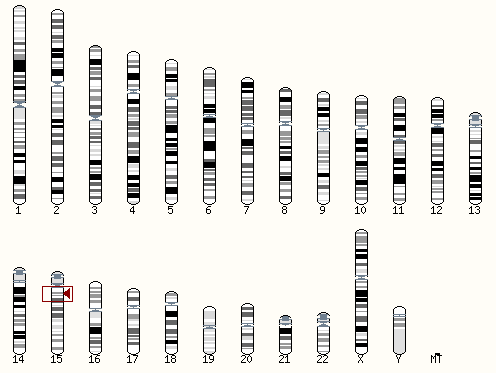
Рисунок 1. Кариотип
По гиперссылке "Contig view" можно перейти на страницу с детальным описанием участка ДНК, с которым провелось выравнивание.
Ниже представлено изображение детального описания участка генома в области гена. В нем указана разметка ДНК в мегабазах, контиги и закодировавнные гены. Псевдогены выделены серым цветом, РНК гены - фиолетовым, Транскрипт - синим и участки, определенные как EnsEMBL так и Havana - желтым.
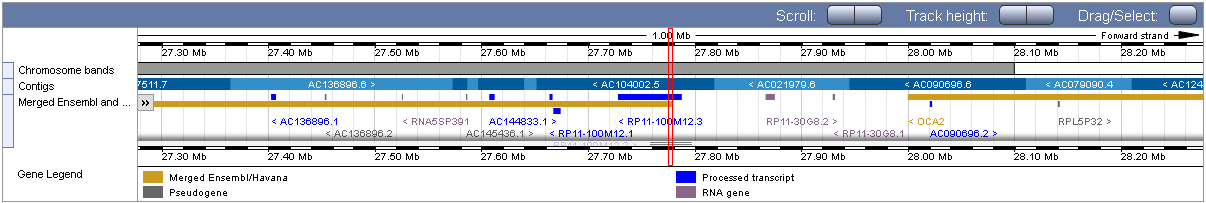 Рисунок 2. Детальное описание области гена
Рисунок 2. Детальное описание области гена
Ниже приведена схема контига, содержащий исследуемый ген. Изображении выделено разными цветами: гены, кодирующие белки- красным или желтым; псевдогены- серые; некодирующие гены- голубые; РНК-кодирующие гены- светло-фиолетовые. Там же указано процентное содержание GC-пар оснований в виде графика.
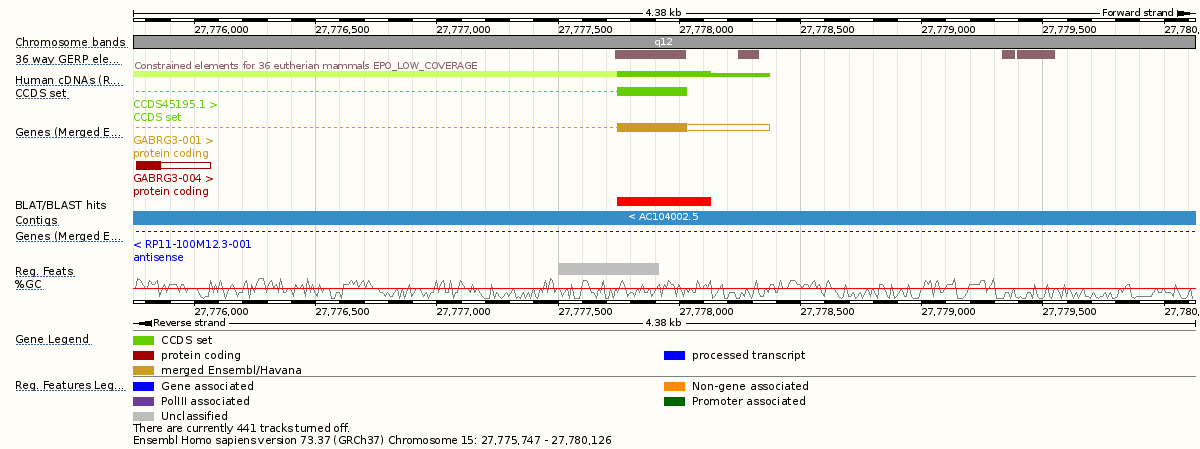 Рисунок 3. Схема контига
Рисунок 3. Схема контига