Задание 1
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определим,
к каким таксонам относятся бактерии, отобранные в предыдущем задании.
Все отобранные бактерии относятся к Cellular organisms, Bacteria, Firmicutes. Дальнейшие различия в таксономии представлены в таблице 1:
Таксоны |
Название |
Мнемоника |
Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
Bacillus subtilis |
BACSU |
Clostridia; Clostridiales; Clostridiaceae; Clostridium |
Clostridium botulinum |
CLOB1 |
Clostridia; Clostridiales; Clostridiaceae; Clostridium |
Clostridium tetani |
CLOTE |
Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
Lactobacillus delbrueckii |
LACDA |
Bacilli; Bacillales; Listeriaceae; Listeria |
Listeria monocytogenes |
LISMO |
Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
Staphylococcus aureus |
STAA1 |
Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
Staphylococcus epidermidis |
STAES |
Определим на изображении филогенетического дерева, полученного в предыдущем задании, ветви, выделяющие какие-нибудь таксоны:
Задание 2
Из Swiss-Prot для выбранных бактерий получим последовательности белков - факторов высвобождения пептидной цепи 1(RF1).
Поместим последовательности в один fasta-файл rf1_bacteria.fasta, отредактировав названия последовательностей,
оставив только мнемонику видов.
Далее выровняем последовательности с помощью JalView (воспользуемся Web Service, Muscle with Defaults)
Ниже представлено изображение (рис. 2) выравнивания в блочной форме с раскраской по проценту идентичности:
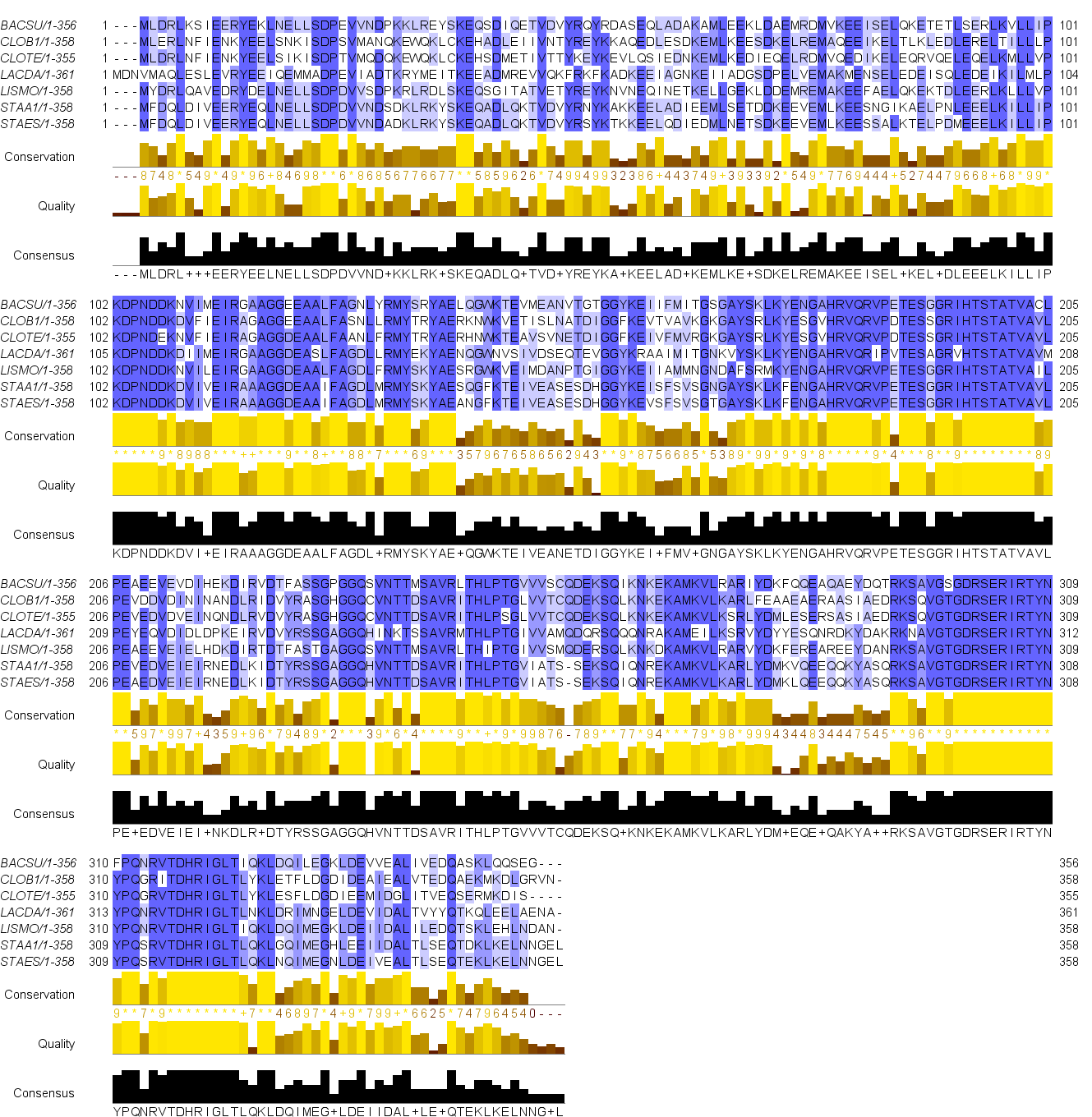 Рисунок 2. Выравнивание белковых последовательностей RF1 выбранных бактерий
Рисунок 2. Выравнивание белковых последовательностей RF1 выбранных бактерий
Выравнивание доступно в форматах FASTA и JalView.
Задание 4
Деревья были реконструированы четырьмя методами, доступными из JalView (Меню =>Calculate => Calculate tree).
Каждое дерево сохранено в Newick-формате в файл с соответствующим названием. Изображения получены с помощью программы Mega.
1. Average Distance Using Percentage Identity
Здесь неправильно выделены ветви Clostridia, Bacilli и Bacilliales.
В дереве №1, построенном программой JalView есть все ветви, что и в правильном.
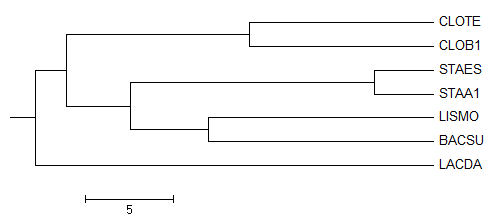 Дерево №1. Average Distance Using Percentage Identity
Дерево №1. Average Distance Using Percentage Identity
2. Neighbour Joining Using Percentage Identity
Ничем, кроме расстояний, не отличается от дерева №1.
 Дерево №2. Neighbour Joining Using Percentage Identity
Дерево №2. Neighbour Joining Using Percentage Identity
Average Distance Using BLOSUM62
Не отличается от дерева №1.
 Дерево №3. Average Distance Using BLOSUM62
Дерево №3. Average Distance Using BLOSUM62
Neighbour Joining Using BLOSUM62
Здесь имеется неразрешенная веть, поэтому количество нетривиальных ветвей будеть больше. Будут такие же ветви, что и в правильном дереве:
1) {CLOTE,CLOB1} против {LACDA,LISMO,BACSU,STAA1,STAES}
2) {CLOTE,CLOB1,LACDA} против {STAA1,STAES,LISMO,BACSU}
3) {LISMO,BACSU} против {CLOTE,CLOB1,LACDA,STAA1,STAES}
4) {STAA1,STAES} против {CLOTE,CLOB1,LACDA,LISMO,BACSU}
И появятся "новые" ветви: 1) {CLOTE,CLOB1,STAA1,STAES} против {LACDA,LISMO,BACSU} 2) {LISMO,BACSU,LACDA} против {STAA1,STAES,CLOTE,CLOB1} 2) {STAA1,STAES,LACDA} против {CLOTE,CLOB1,LISMO,BACSU}
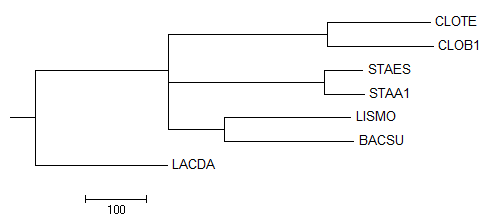 Дерево №4. Neighbour Joining Using BLOSUM62
Дерево №4. Neighbour Joining Using BLOSUM62
Судя по всем этим деревьям, можно сделать предположение, что общий предок белка RF1 разделился на два паралога, один из которых достался только LACDA.
Ортологи данного белка, принадлежащие бактериям из Clostridiales эволюционировали очень сильно по сравнению с остальными.
Задание 5
Выравнивание импортировано в программу Mega (при импорте выбран "Analysis"). Реконструировано дерево методом "Maximum Parsimony". Укоренено дерево в наиболее правильную, с моей точки зрения, ветвь (меню Subtree => Root).
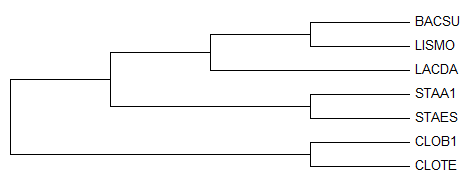 Рисунок 7. Дерево, построенное на основе выравнивания
Рисунок 7. Дерево, построенное на основе выравнивания
Общие ветви
1) {CLOTE,CLOB1} против {LACDA,LISMO,BACSU,STAA1,STAES}
2) {STAA1,STAES} против {CLOTE,CLOB1,LACDA,LISMO,BACSU}
3) {LISMO,BACSU} против {CLOTE,CLOB1,LACDA,STAA1,STAES}
Отличия от правильного дерева:
1) {BACSU,LISMO,LACDA} против {STAA1,STAES,CLOTE,CLOB1,STAA1,STAES}
В правильном дереве была ветвь:
1) {CLOTE,CLOB1,LACDA} против {BACSU,LISMO,STAA1,STAES,STAA1,STAES}