Задание 1. Укоренение в среднюю точку
Укореним в среднюю точку дерево, построенное при выполнении предыдущего занятия методом Neigbour Joining Tree Using BLOSUM62.
Для этого воспользуемся программой retree пакета PHYLIP:
1. Скопируем файл с деревом в Newick-формате в файл с именем intree;
2. Запустим retree.exe;
3. Далее - M - для укоренения;
4. Наконец - W - для записи в выходной файл.
Выходному файлу outtree добавим расширение .nwk и откроем программой MEGA:
Укорение произошло в правильную точку: так, что, полученное дерево идентично с исходным.
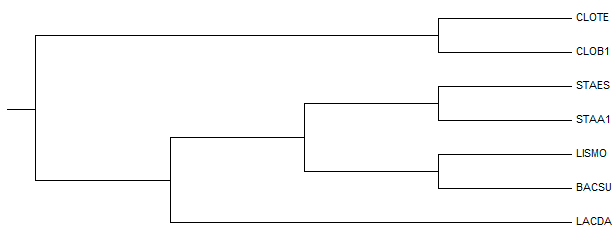 Рисунок 1. Дерево, укорененное в среднюю точку программой retree пакета PHYLIP
Рисунок 1. Дерево, укорененное в среднюю точку программой retree пакета PHYLIP
Задание 2. Использование внешней группы
Метод Maximum Parsimony определяет оптимальное дерево так, чтобы свести к минимуму количество мутаций.
Но расстояний между листьями нет, поэтому нельзя выбрать самый длинный путь и провести укоренение в среднюю точку.
Можно воспользоваться укоренением с помощью внешней группы. Такой группой будет тот же белок из другой бактерии - Escherichia coli (ECOLI).
Все отобранные бактерии - грамположительные, а кишечная палочка - грамотрицательная.
1. Добавим к файлу с невыровненными последовавтельностями белков последовательность белка из кишечной палочки;
2. Далее выровним с помощью сервиса Muscle with Defaults программы JalView;
3. Импортируем выравнивание в программу MEGA;
4. Построим дерево методом "Maximum Parsimony";
5. Наконец вырезаем ветвь, ведущую к ECOLI.
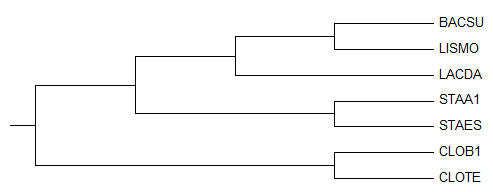 Рисунок 2. Дерево, построенное с использованием внешней группы
Рисунок 2. Дерево, построенное с использованием внешней группы
Видно, что укоренение произощло в правильную точку.
Неверная нетривиальная ветвь:
(BACSU,LISMO,LACDA) против(STAES, STAA1, CLOB1, CLOTE)
Задание 3. Бутстрэп
Проведем бутстрэп-анализ филогении белков, используя один из методов, доступных программе MEGA. Для этого в окне, которое открывается после вызова программы, в меню "Test of Phylogeny" выберем "Bootstrap method". Число реплик выберем 100.
Original tree:
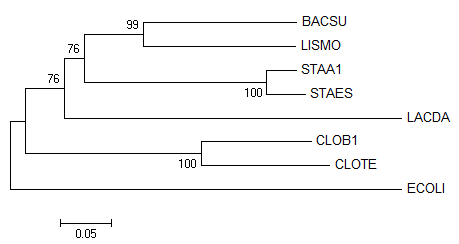 Рисунок 2. Original tree
Рисунок 2. Original tree
Bootstrap consensus tree:
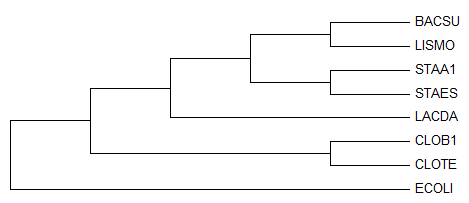 Рисунок 2. Bootstrap consensus tree
Рисунок 2. Bootstrap consensus tree
Деревья "Original tree" и "Bootstrap consensus tree" ничем не отличаются и имеют правильные тополгоии. Значит, построение по части данных (100 реплик по случайной половине имеющихся столбцов выравнивания) примерно такое же по достоверности, как и по полноценным данным.