Описание трансмембранных доменов
Ссылки:
1. Orientation of Proteins in Membranes (OPM) Database;
2. Protein Data Bank of Transmembrane (PDBTM) Proteins;
3. Transporter Classification Database (TCDB).
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков были определены параметры, перечисленные в Таблице 1:
| PDB ID | Тип | Локализация | Организм | Толщина гидрофобной части мембраны, Å | Медиана числа остатков в одном трансмембранном участке |
| 2iww | Баррель | Внешняя мембрана клеточной стенки грамотрицательных бактерий | E. coli | 24.7 ± 1.4 | 10 |
| 2jk4 | Баррель | Внешняя митохондриальная мембрана | H. sapiens | 23.4 ± 2.3 | 8 |
| 1bxw | Баррель | Внешняя мембрана клеточной стенки грамотрицательных бактерий | E. coli | 25.4 ± 1.9 | 9,5 |
| 2hac | Спираль | Плазматическая мембрана | H. sapiens | 31.9 ± 2.3 | 21 |
| 2m0b | Спираль | Плазматическая мембрана | H. sapiens | 31.8 ± 2.5 | 19 |
| 1nrj | Спираль | Цитоплазматическая строна мембраны эндоплазматичекого ретикулума | S. cerevisiae | 3.9 ± 1.4 | 21 |
Отбор гомологов
Для белка с PDB ID 4BOG (цепь A) с помощью PSI-BLAST был произведён поиск гомологов. Белок со структурой 4BOG принадлежит скату Torpedo marmorata. При поиске гомологов были исключены белки из типа Chordata. В итоге было отобрано 12 хитов из разных таксономических групп. Затем названия последовательностей были отредактированы с сохранением ID белка и соответствующего ему организма (скачать FASTA).
Анализ структуры выданного белка
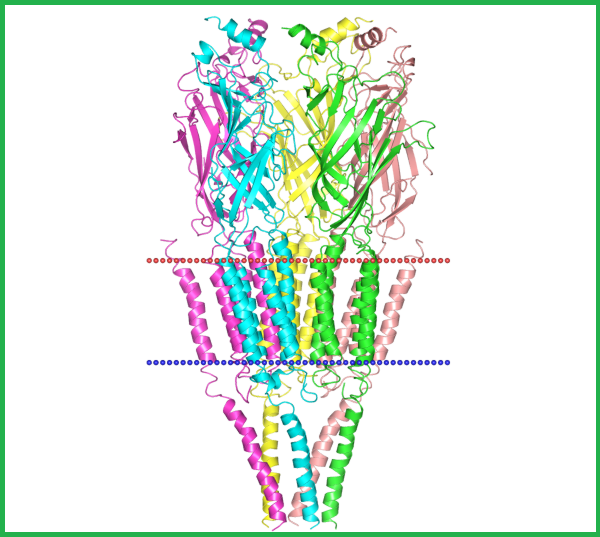
Рисунок 1. Белок Q6S3I0_TORMA (идентификатор PDB 4BOG, цепь B)
Информация о белке со структурой 4BOG (цепь B), полученная с помощью баз данных OPM и TCDB, представлена в таблице 2:
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей |
| 4BOG | Torpedo marmorata | Плазматическая мембрана | 1.A.9 | 3 ± 0°
Отдельно для пяти субъединиц: 15°, 14°, 10°, 8°, 14°. |
4 |
Описание каждого из полей TC-кода для белка 4BOG:
3 – Каналы/поры;
3.D – Каналы альфа-типа;
3.D.4 – Семейство нейротрансмиттерных рецепторов, цис-петель, лиганд-зависимых ионных каналов (LIC);
Записей конкретно для белка со структурой 4BOG в базе TCDB не существует.
Анализ множественного выравнивания трансмембранных белков
С помощью сервиса muscle на сервере kodomo было построено множественное выравнивание отобранных гомологов. Затем результат был загружен в программу JalView. К выравниванию были добавлены аннотации TM_REAL и TM_PREDICTED. TM_REAL содержит указания на участки выравнивания отвечающие трансмембранным спиралям в белке со структурой 2BG9 (репрезентативная структура белка с PDB ID 4BOG. Источник: ссылка), которая была прикреплена к последовательности белка Q6S3I0_TORMA (исходного) в выравнивании в программе JalView. TM_PREDICTED обозначает предсказанные с помощью TMHMM участки трансмембранных спиралей в гомологе K1Q8W5_CRAGI.
После применения цветовой схемы, позволяющей визуально различать гидрофобные и гидрофильные остатки (Hydrophobicity: гидрофобные остатки выделены красным цветом), вместе с зависимостью интенсивности цвета от консервативности позиции (By Conservation, было использовано значение 20% в качестве порога) было получено следующее изображение (Рисунок 2):
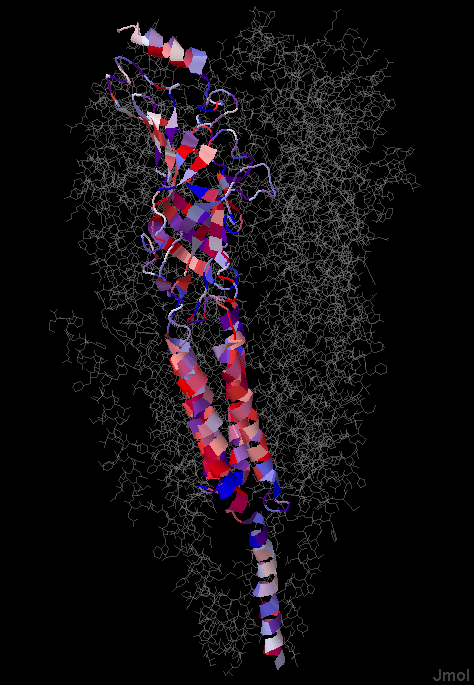
Рисунок 2. Белок Q6S3I0_TORMA. (2BG9, цепь B)
Часть белка, ориентированная в n-сторону мембраны, расположена внизу. Участки, относящиеся к коровым (не по взаимному расположению, а по последовательности) трансмембранным спиралям, консервативны, но участки спиралей, находящиеся на концах последовательности, значительно менее консервативны. при этом наиболее часто в спиралях встречаются остатки лейцина, изолейцина, валина, аланина, фениалаланина, метионина, глицина и пролина (как видно, трансмембранные участки действительно состоят в основном из гидрофобных остатков). Среди участков между спиралями встречаются как консервативные участки, так и неконсервативные (относительно порога в 20% для цветового отображения консервативности).
Стоит отметить, что в трансмембранных спиралях также нередко встречаются остатки серина, аспарагина, тирозина (полярны), и даже гистидина (полярен и положительно заряжен) и глутаминовой кислоты (полярен и заряжен отрицательно). Эти остатки как правило консервативны (в особенности консервативны остатки гистидина), поэтому закономерно предположить, что их наличие связано с функциональной ролью белка.
Выравнивание в формате .jar (проект JalView) (Рисунок 3):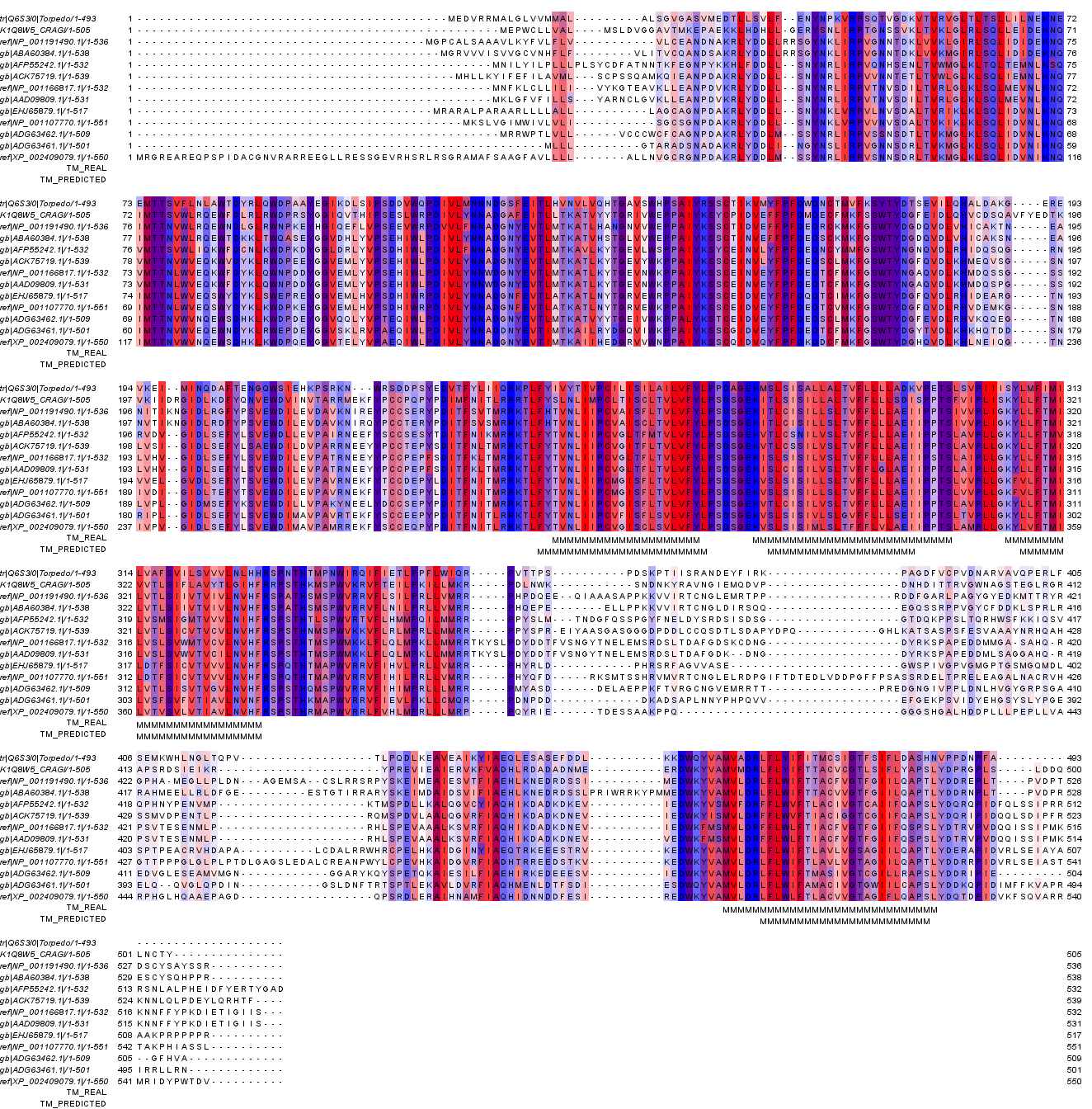
Рисунок 3. Множественное выравнивание гомологов в программе JalView.
Если сравнивать результаты программы TMHMM и реальную структурную информацию по совпадению на выравнивании, то можно отметить, что все трансмембранные спирали, кроме двух крайних, были предсказаны. Предсказания и структурная информация совпадает полностью, но структурня часть в большинстве случаев больше на несколько остатков, возможно, это вызвано тем, что крайние аминокилслоты часто выходят за пределы мембраны и поэтому нередко бывают полярными, что не подходит под критерий отбора предсказаний в программе TMHMM. Программа оставила полностью не замеченными две спирали. Не удивительно, поскольку они не являются трансмембранными.