Выбор структуры
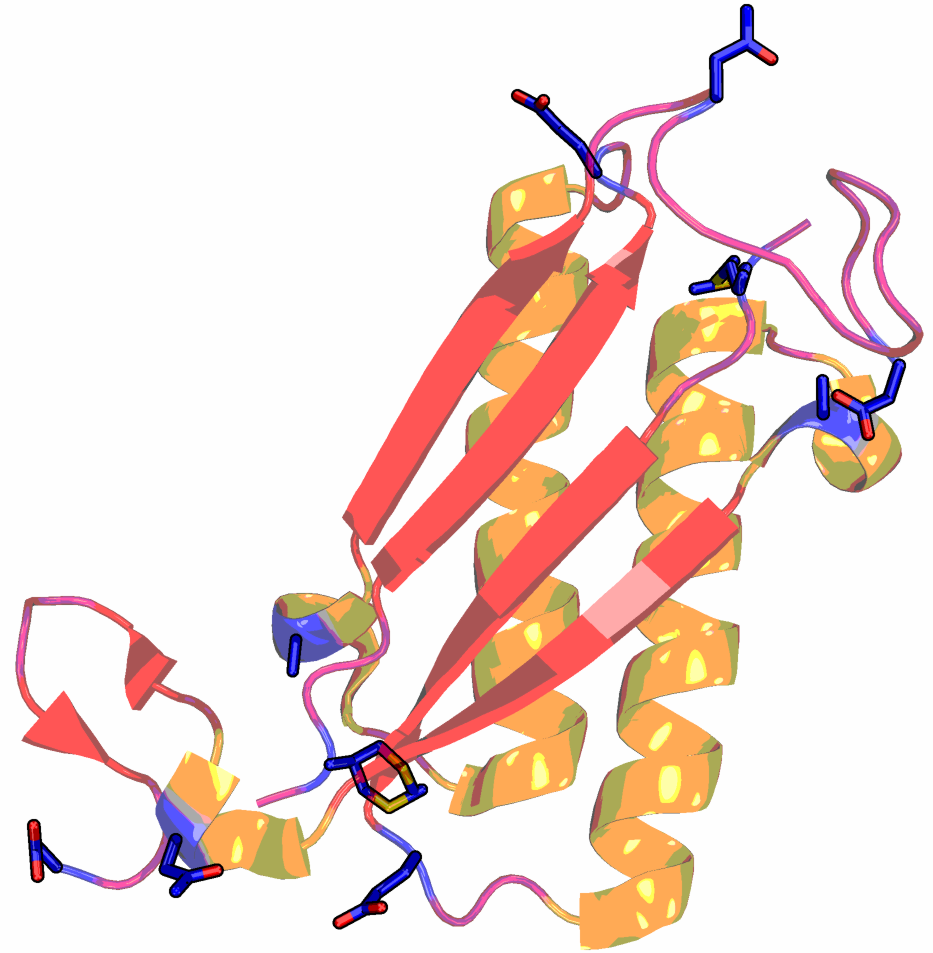
Для работы была выбрана структура фермента 4-оксалокротонат таутомеразы из бактерии Methylibium petroleiphilum PM1 (PDB ID: 4FAZ Разрешение: 1.57 Å Длина цепи: 62).
 |
 |
 |
|
 |
Этот фермент необычен тем, что его мономер длиной всего 62 аминокислотных остатка является самым маленьким среди всех известных ферментов.
Выбранная структура удовлетворяет следующим требованиям:
- 1. Информация о структуре содержится в сервере EDS. Это равносильно тому, что в PDB, помимо модели структуры, помещены экспериментальные данные - файл структурных факторов(Structure factors).
- 2. Имеются подходящие структурные гомологи белка. Это было проверено с помощью сервера PDBeFold. Всего было найдено 208 совпадений. Если ограничиться параметрами 0.8Å ≤ RMSD ≤ 3.0Å и 31 ≤ N_align≤ 55, то остается 14 разных структур.
Изображение электронной плотности вокруг полипептидной цепи
Очевидно, что для визуализации электроной плотности не достаточно иметь лишь координат атомов белка. Поэтому, во-первых, с сервера EDS был загружен файл с картой электронной плотности для 4FAZ. Во-вторых, собственно для самой визуализации структуры и электронной плотности белка была использована программа PyMOL.
Итак, с помощью карты электронной плотности можно оценивать качество модели пространственной структуры, представленной в PDB файле. С целью, чтобы эта оценка была более качественной, были использованы три уровня подрезки (1.0σ, 1.5σ и 2.0σ) изоповерхности электронной плотности, который означает превышение сигнала над шумом и измерется статистически - в терминах среднеквадратических отклонений. Также существует параметр carve. Этот параметр нужен для того, чтобы отсекать сигнал от соседних атомов. Он представляет собой ни что иное, как расстояние от выбранного множества атомов, в пределах которого будет учитываться электронная плотность. Значение этого параметра в данной работе всегда будет составлять 2.0Å.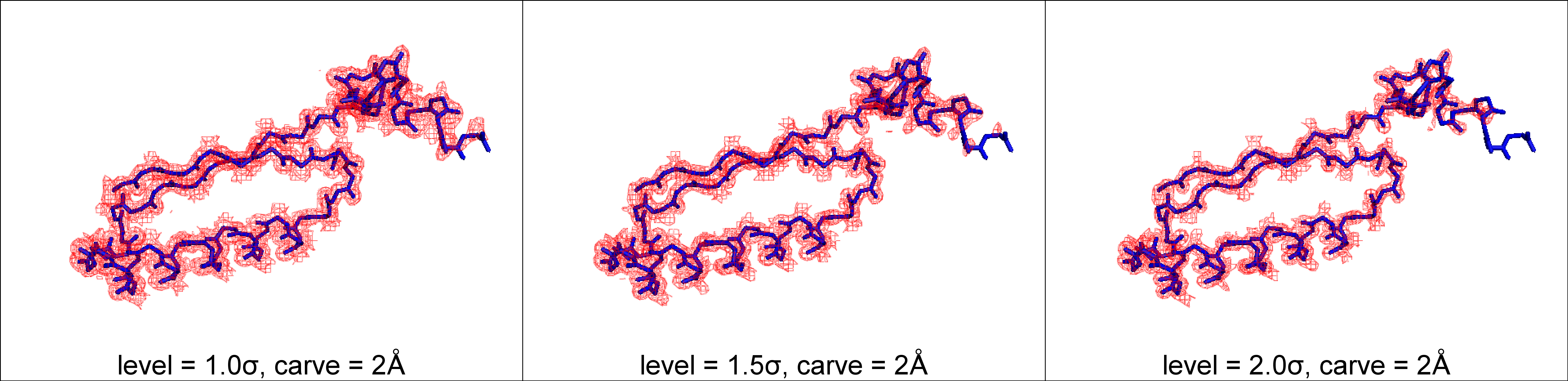 Рисунок 2.
Электронная плотность вокруг полипептидной цепи мономера 4-ОТ при разных уровнях подрезки (1.0σ, 1.5σ и 2.0σ).
Синим выделена полипептидная цепь (без боковых цепей аминокислот). Красным - электронная плотность при разных уровнх подрезки.
Рисунок 2.
Электронная плотность вокруг полипептидной цепи мономера 4-ОТ при разных уровнях подрезки (1.0σ, 1.5σ и 2.0σ).
Синим выделена полипептидная цепь (без боковых цепей аминокислот). Красным - электронная плотность при разных уровнх подрезки.
На рисунке видно, что электроннная плотность в целом хорошо соответствует модели. Однако для всех уровней подрезки в C-конце полипептидной цепи электронная плотность пропадает.
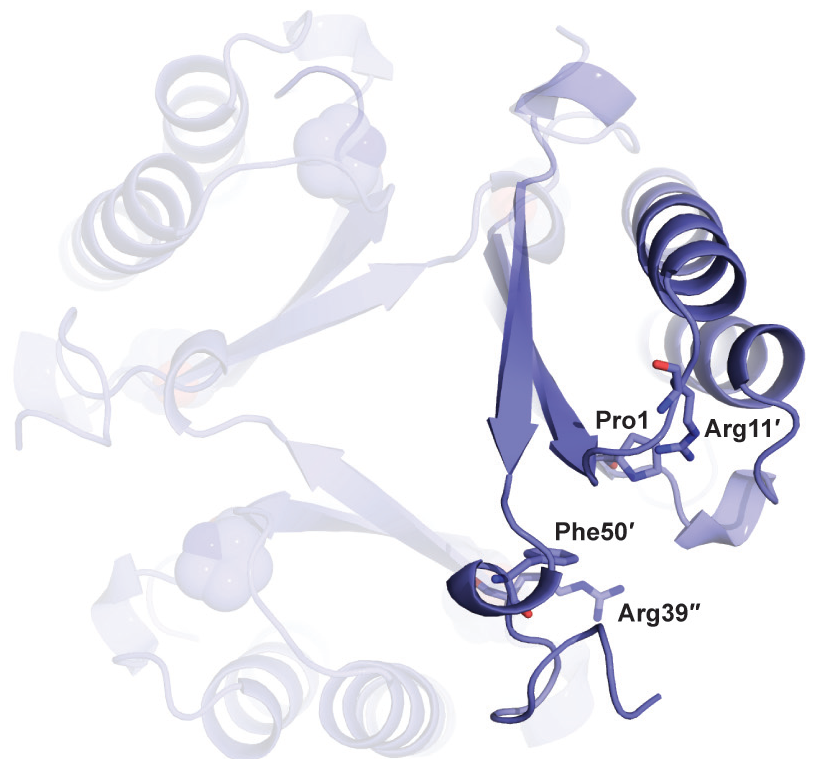
Изображения электронных плотностей для аминокислотных остатков, входящих в активный центр 4-ОТ: Pro1, Arg39, Trp50
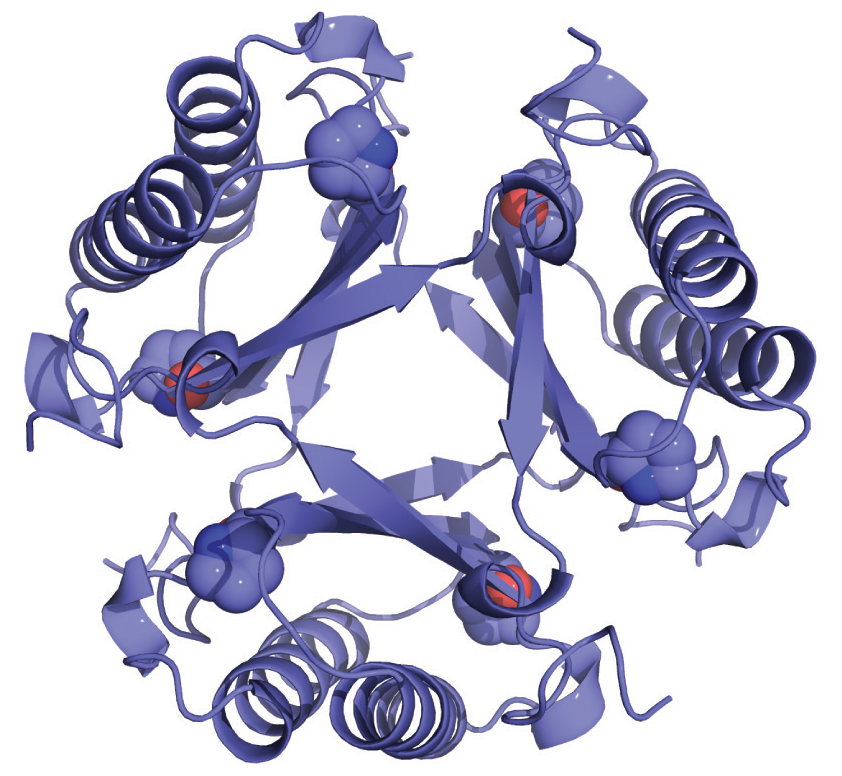
На самом деле, в активный центра фермента 4-ОТ входит 6 аминокислотных остатков от одного мономера. Вообще, фермент представляет собой гомогесамер и имеет три активных центра, расположенных между мономерами.
Ниже на изображениях показана электронная плотность вокруг аминокислотных остатков пролина (Pro1); аргинина (Arg39) и триптофана (Trp50) для трех уровней подрезки: 1.0σ, 1.5σ и 2.0σ (рис.3-5):
Pro1
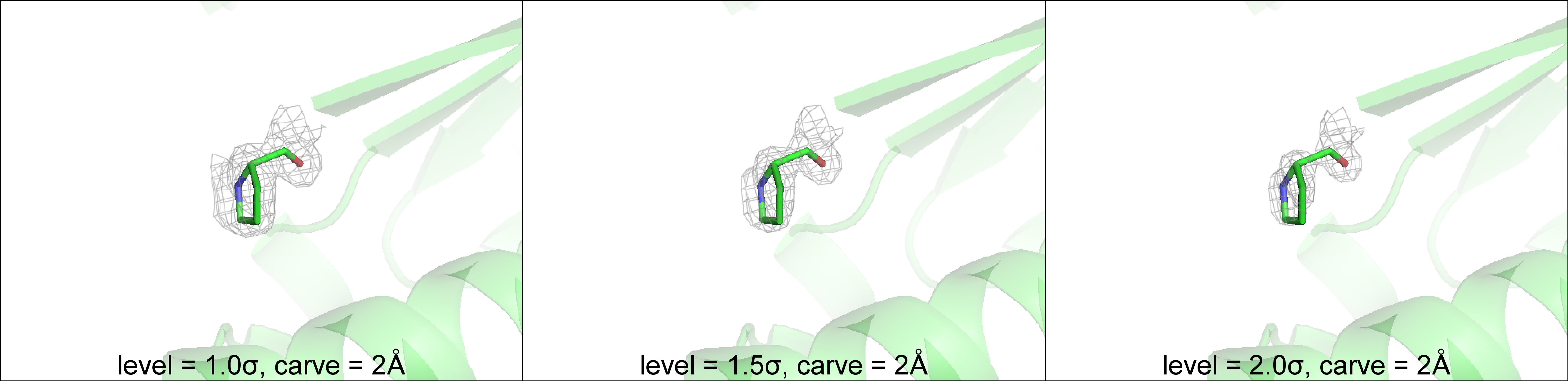 Рисунок 3. Электронная плотность вокруг Pro1.
Стержневая модель.
Рисунок 3. Электронная плотность вокруг Pro1.
Стержневая модель.
Arg39
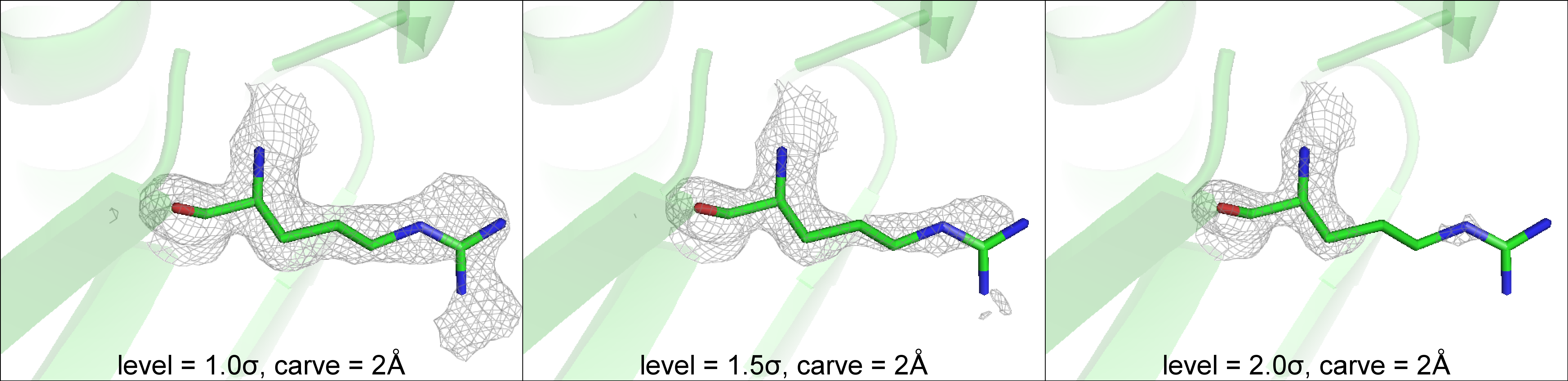 Рисунок 4. Электронная плотность вокруг Arg39.
Стержневая модель.
Рисунок 4. Электронная плотность вокруг Arg39.
Стержневая модель.
На приведённых изображениях (рис.3 и рис.4) по паттерну электронной плотности видно положение боковых групп аминокислот, однако наблюдаются и случаи несоответствия со структурой. Например, электронная плотность бензольного колца, входящего в состав индола боковой цепи триптофана даже при уровне подрезки в 0.2σ восстанавливается не полностью (рис.5):
Trp50
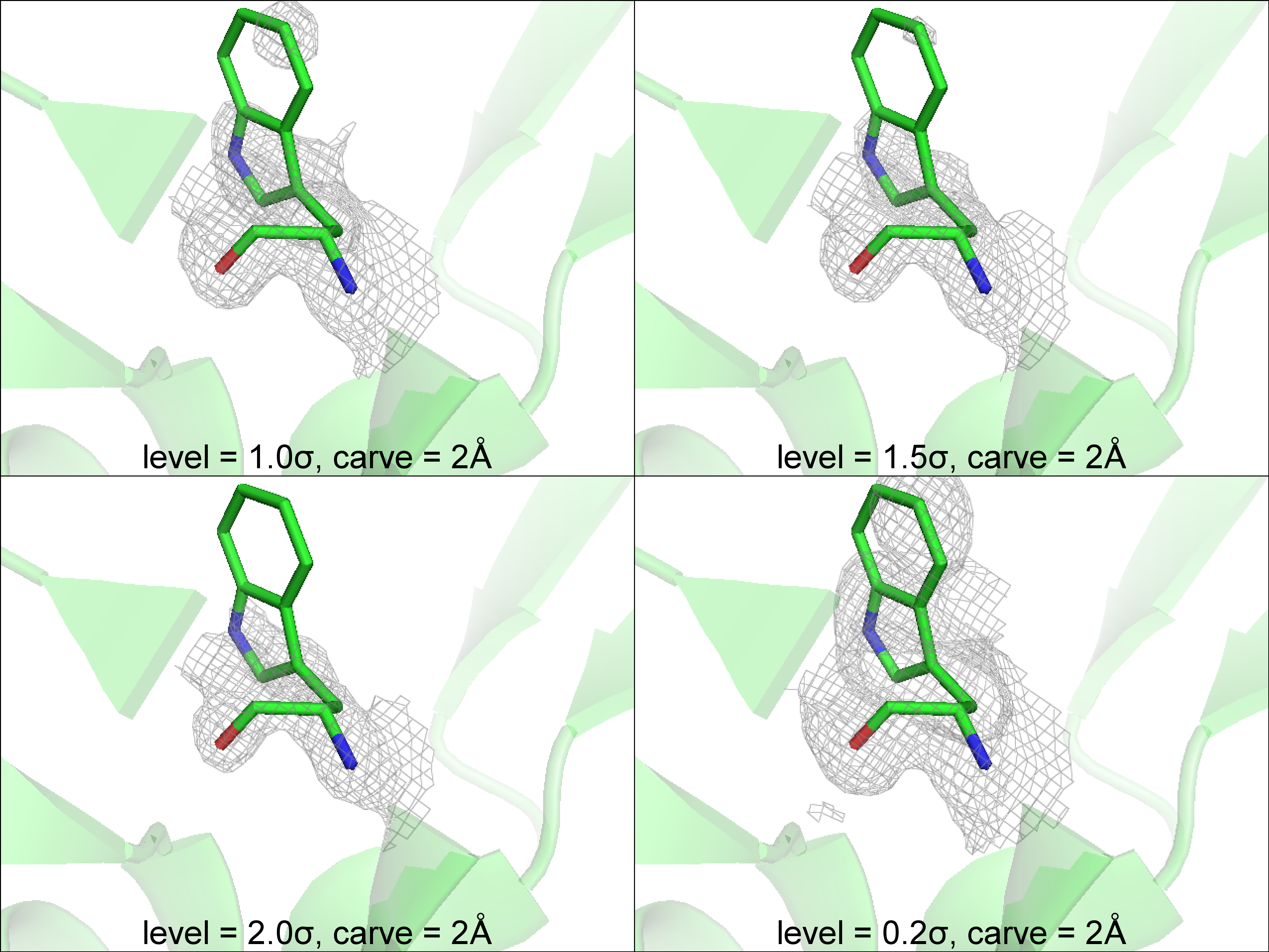 Рисунок 5. Электронная плотность вокруг Trp50.
Стержневая модель.
Рисунок 5. Электронная плотность вокруг Trp50.
Стержневая модель.
Заключение
На основании визуализации электронной плотности вокруг полипептидной цепи (рис. 2) и отдельных аминокислотных остатков (рис. 3,4,5) можно заключить следующее: разрешение структуры 4FAZ не является достаточным для того, чтобы наблюдать отдельные атомы на изображении карты электронной плотности, однако по этой карте виден ход полипептидной цепи и угадывается положение боковых групп аминокислот.