Совмещение структур 4FAZ и структурных гомологов
С помощью сервиса PDBeFold были выбраны четыре структурных гомолога для цепи A структуры 4FAZ: 3M21:A, 2ORM:B, 3EJ7:H, 2X4K:B, построено их совмещение (Рис. 1) и множественное выравнивание последовательностей по совмещению структур.
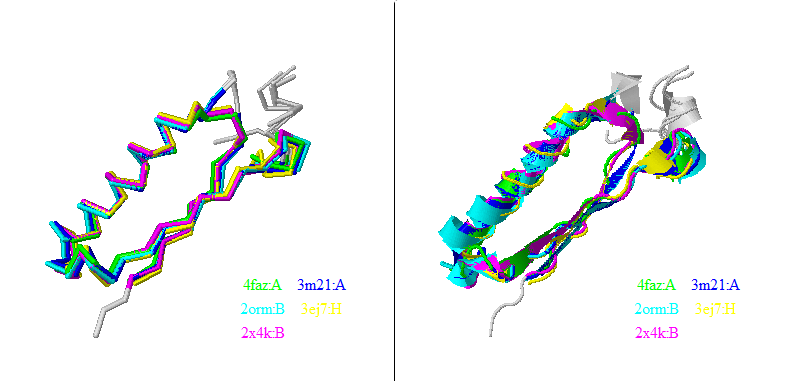 Рисунок 1. Cовмещение структур гомологов 4FAZ в JMol,
предоставляемое сервисом PDBeFold. Справа: backbone-диаграмма, слева: cartoon-диаграмма.
Рисунок 1. Cовмещение структур гомологов 4FAZ в JMol,
предоставляемое сервисом PDBeFold. Справа: backbone-диаграмма, слева: cartoon-диаграмма.
Выравнивание по структуре (большие буквы в файле с выравниванием) можно сравнить с выравниванием последовательностей, выполненным в программе JalView (с помощью Tcoffee со стандартными параметрами):
 Рисунок 2. Выравнивание последовательностей гомологов 4FAZ
в JalView; окраска по проценту идентичности.
Рисунок 2. Выравнивание последовательностей гомологов 4FAZ
в JalView; окраска по проценту идентичности.
Выравнивания последовательностей по структуре и по последовательнсти практически не отличаюся (Рис. 3):
 Рисунок 3. Сверху: выравнивание последовательностей
в JalView, Снизу: выравнивание последовательностей по структуре. Окраска по проценту идентичности.
Рисунок 3. Сверху: выравнивание последовательностей
в JalView, Снизу: выравнивание последовательностей по структуре. Окраска по проценту идентичности.
Поиск структурных гомологов для домена 1dek A: 33-154
В сервисе PDBeFold был осуществлен поиск стрктурных гомологов для домена 1dek A:33-154 со стандартными параметрами(Рис. 4):

Рисунок 4. Параметры поиска структурных гомологов для домена 1dek A:33-154 в PDBeFold.
Нашлись 33 находки (см. результат), среди которых искомого домена не было. Как видно, параметры по умолчанию требуют, чтобы структуры совпадали не меньше, чем на 70%. Для многодоменного белка искомый домен занимает меньше 70% общей длины белка, поэтому сам белок находиться не будет. При снижении порога до 50% (Рис. 5) искомый домен нашелся (см. результат).
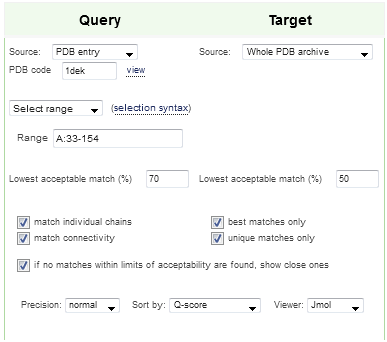
Рисунок 5. Параметры повторного поиска структурных гомологов для домена 1dek A:33-154 в PDBeFold.
Совмещение по заданному выравниванию
Для работы были выбраны структуры 1oga, region d: 118-202 и 1oga, region e: 119-245 – моедели константного домена α- и β-цепей, соответственно, человеческого T-клеточного рецептора (T-cell receptor - TCR).
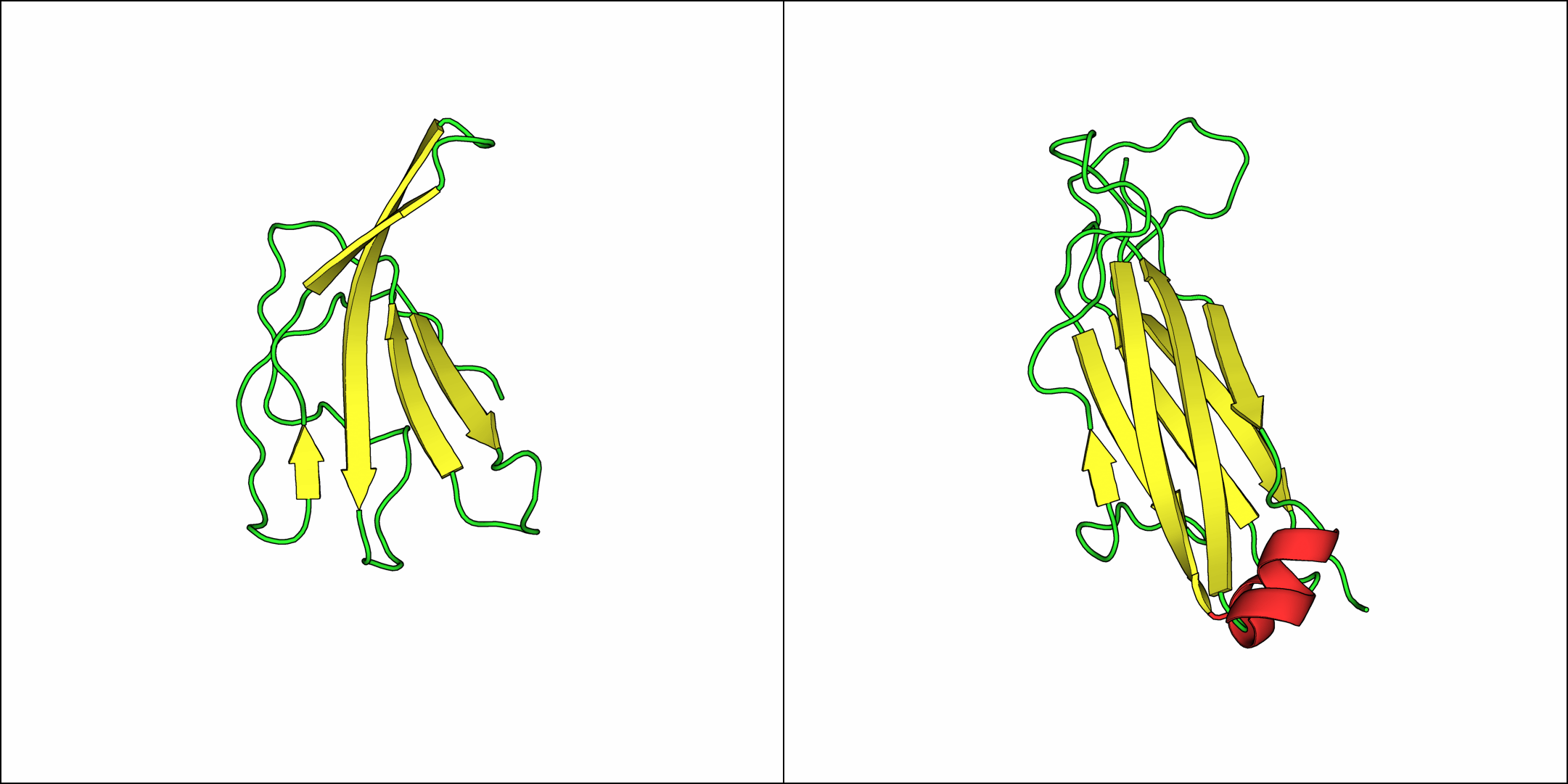 Рисунок 6. Домены TCR-α (слева) и TCR-β (справа).
Рисунок 6. Домены TCR-α (слева) и TCR-β (справа).
При помощи сервиса SheeP был построены карты β-листов. Они представлены на рисунках 6 и 7 в одинаковой ориентации.
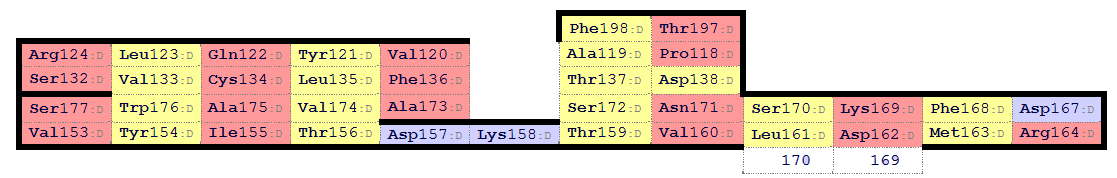 Рисунок 7. Карта β-листа для домена TCR-α (1oga, region d: 118-202)
Рисунок 7. Карта β-листа для домена TCR-α (1oga, region d: 118-202)
 Рисунок 8. Карта β-листа для домена TCR-β (1oga, region e: 119-245)
Рисунок 8. Карта β-листа для домена TCR-β (1oga, region e: 119-245)
При построении выравнивания последовательностей этих β-тяжей консервативные остатки цистеина в них задают выравнивание центрального тяжа. Остатки, спаренные с консервативным цистеином, задают выравнивание «соседних» тяжей. Это можно использовать для совмещения структур в PyMol:
pair_fit \
alpha and resi 134 and (name CA), beta and resi 145 and (name CA), \
alpha and resi 122 and (name CA), beta and resi 127 and (name CA), \
alpha and resi 175 and (name CA), beta and resi 192 and (name CA), \
alpha and resi 155 and (name CA), beta and resi 172 and (name CA), \
alpha and resi 133 and (name CA), beta and resi 144 and (name CA), \
alpha and resi 135 and (name CA), beta and resi 146 and (name CA)
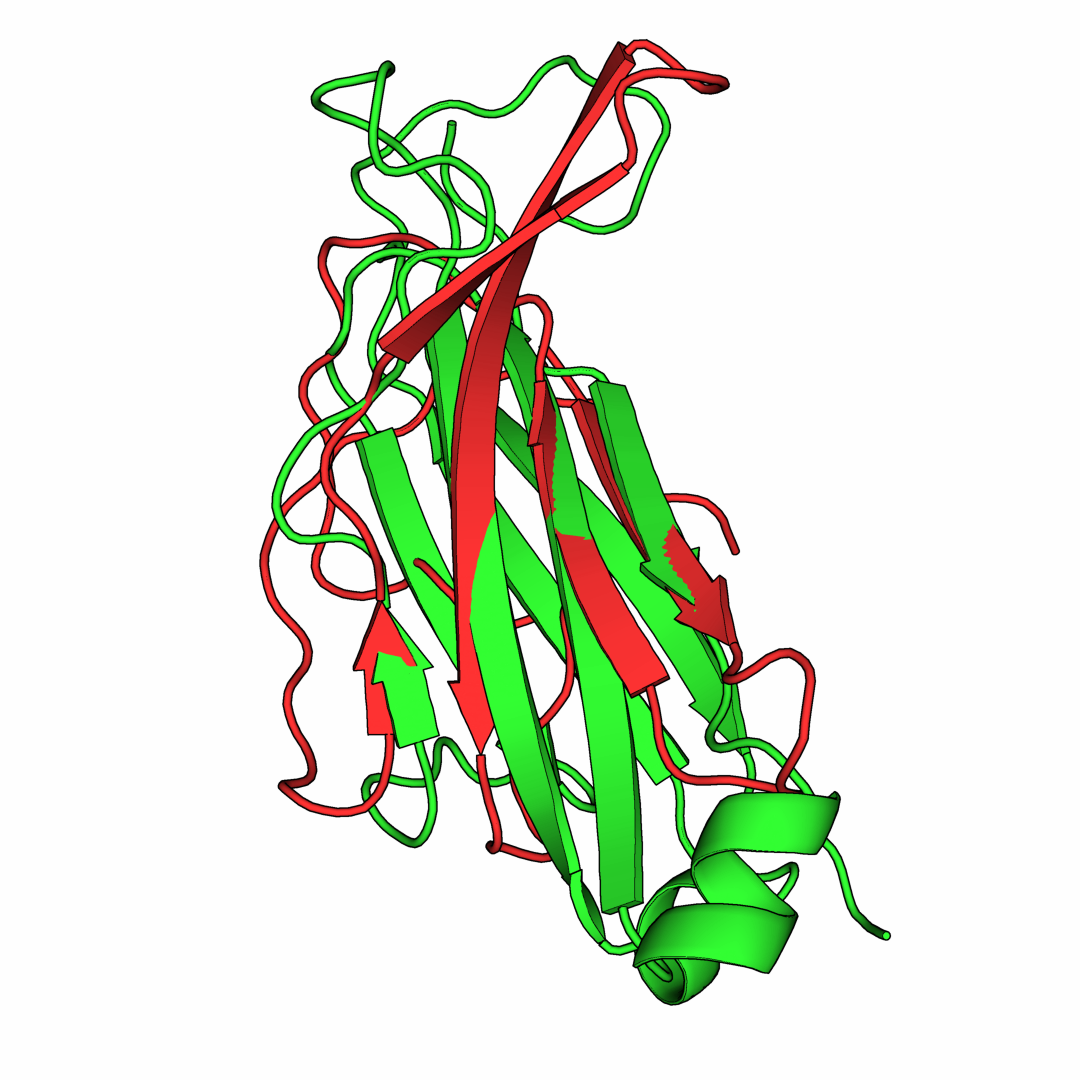 Рисунок 9. Совмещение доменов TCR-α
и TCR-β.
Рисунок 9. Совмещение доменов TCR-α
и TCR-β.
Из рисунка 9 видно, что топологии доменов хорошо совпадают.