| ФББ МГУ | Корень | Обо мне | Семестры |
Рентгеноструктурный анализ
Гидрофобные кластеры
Текст задания: гидрофобные кластеры.
Практикум выполнен с помощью сервиса CluD.
Поиск гидрофобных кластеров в структуре 2G9H
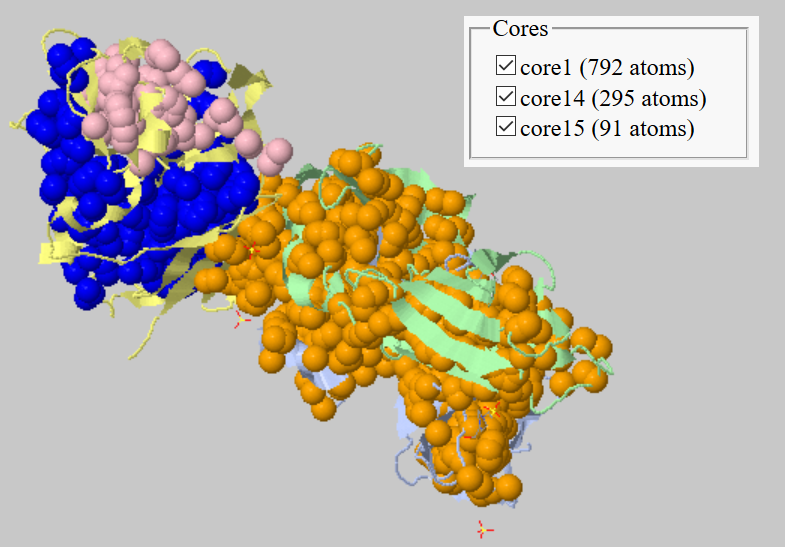
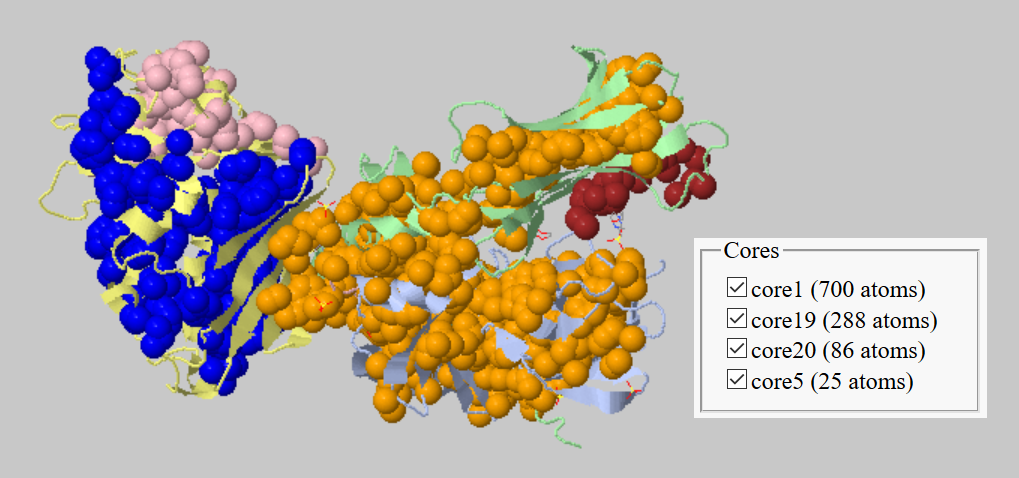
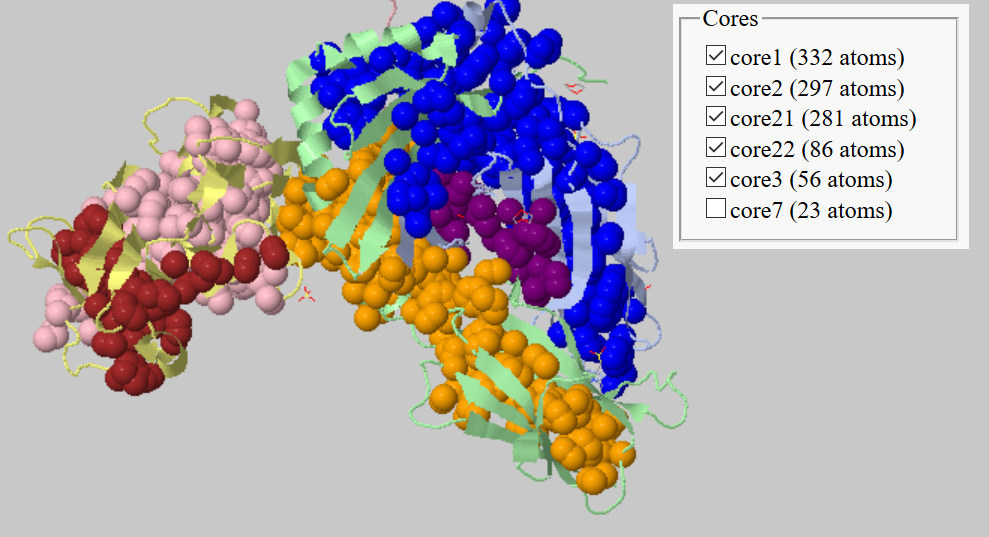
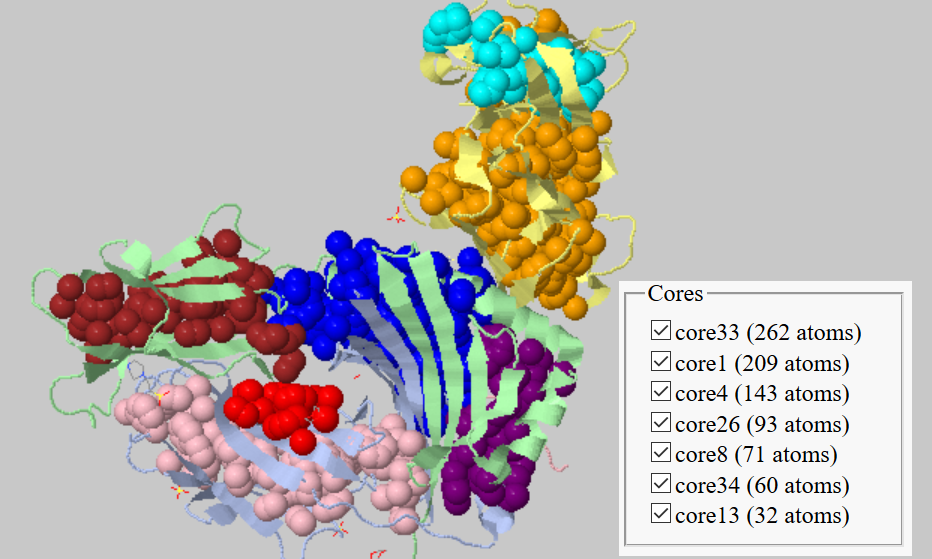
В данном задании я решила рассмотреть структуру комплекса человеческого MHC класса II и Staphylococcal Enterotoxin I (SEI) (PDB ID: 2G9H).
Для выполнения этого задания я поставила галочку напротив "whole structure" и варьировала порог расстояния и размера кластера для получения биологически интерпретируемых результатов. По иллюстрациям в данном разделе можно видеть, что оптимальный порог расстояния 4.55Å. Так гидрофобное ядро не слишком дробится, в отличие от ситуации при использовании порога 4.4Å, и гиброфобные ядра отдельных цепей (доменов) не сливаются в один огромный, как при пороге 4.8Å. Порог размера гиброфобных кластеров не очень важный параметр для работы программы, но он важен с точки зрения биологической интерпретации. В данном случае, мне кажется просто бессмысленным рассматривать кластеры, в которых содержится менее 20 атомов.
Я не уверена, что можно сказать, что гидрофобные кластеры, найденные с порогом расстояния 4.55Å, соответствуют доменам белка, тк домены здесь не образуют разделенных в пространстве структур. Но с точки зрения соответствия расположения кластеров вторичной и третичной структуре белка, наиболее адекватная картинка получается при указанного использовании порога.
 |
 |
|
|
 |
 |
|
|
Поиск гидрофобных кластеров на интерфейсе двух цепочек в структуре тетрамера 1HAB (гемоглобин)
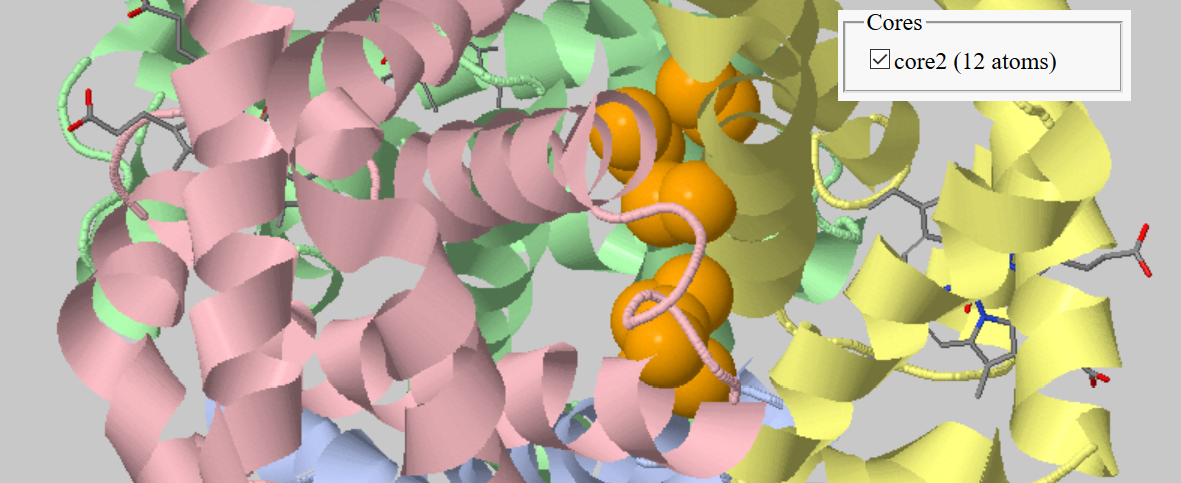
В этом задании я решила проанализировать CROSSLINKED HAEMOGLOBIN (PDB ID: 1HAB).
Сначала я попробовала поставить галочку напротив "whole structure" и поискать кластеры, находящиеся на стыке двух цепочек. Результат можно видеть ниже - был найден кластер размера 24 атома, находящийся на стыке цепочек A и D.
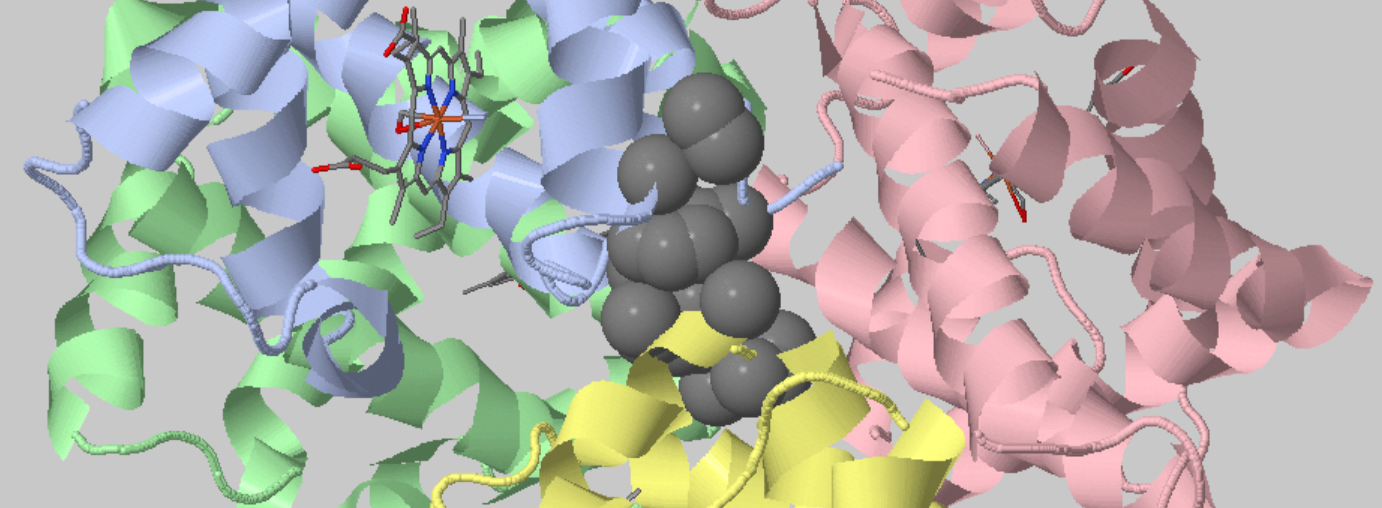
- Порог расстояния = 4.4
- Порог размера кластера = 20
- Найдено кластер размером 24 атома
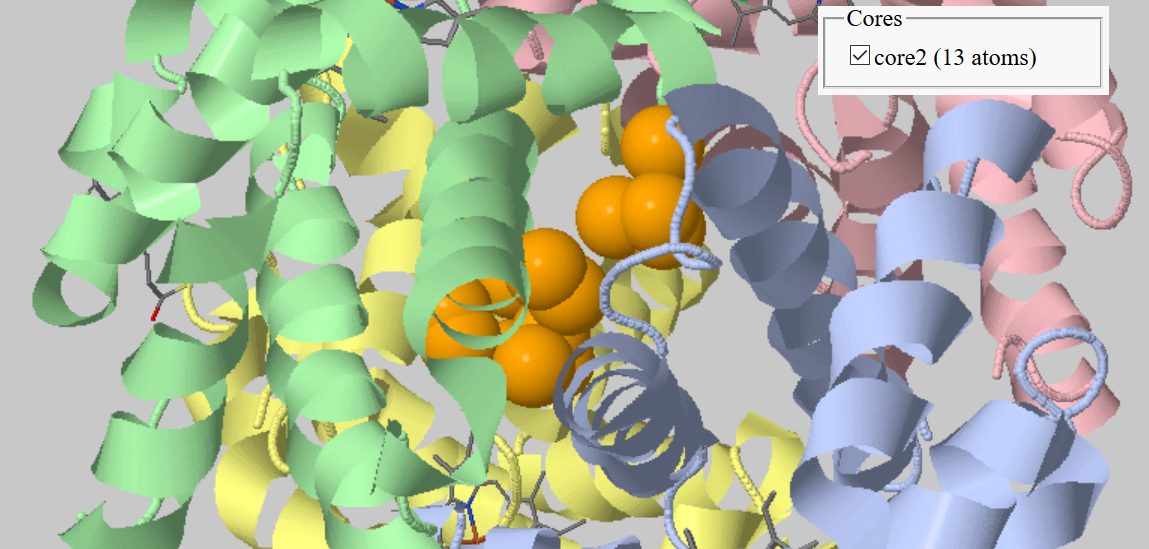
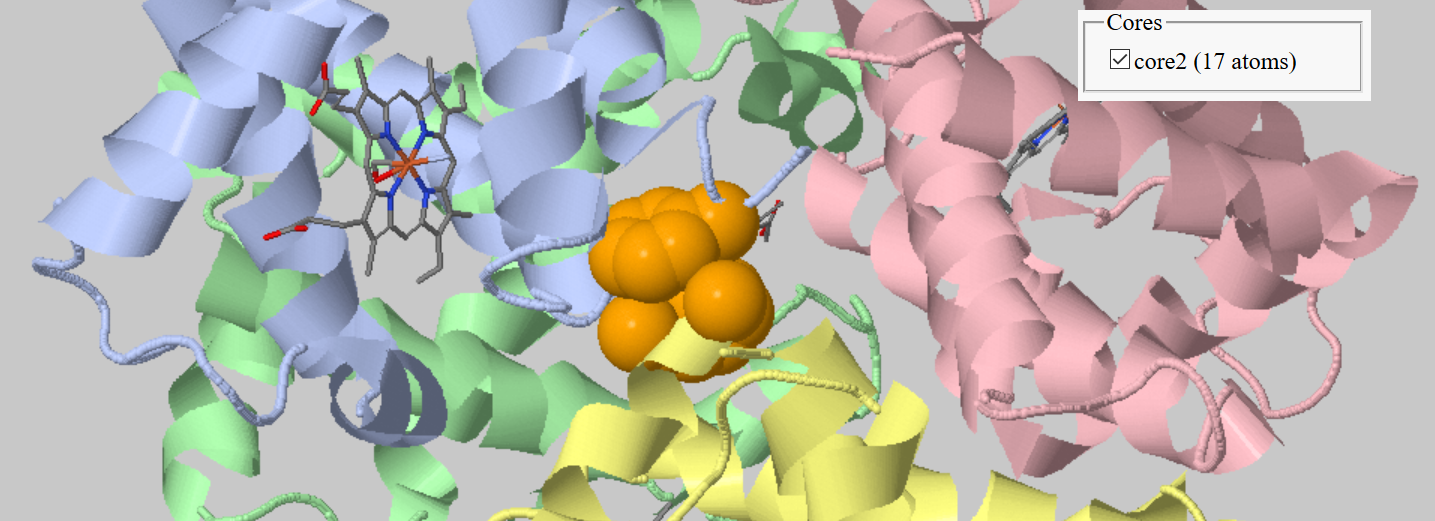
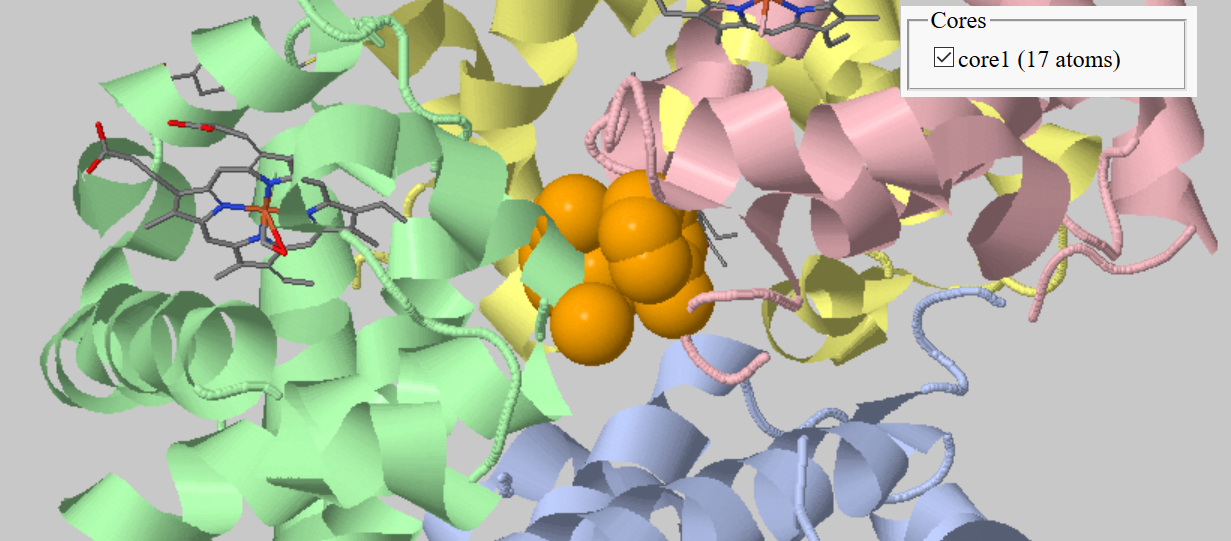
После этого я поставила галочку напротив "between chains" и выбирала разные цепочки гемоглобина (A, B, C, D) для проверки наличия гидрофобных кластеров на их интерфейсе. Результаты можно видеть на рисунках ниже. Ожидаемо, для того, чтобы найти гидрофобные кластеры, обеспечивающие взаимодействие разных цепочек белка, а не поддерживающие фолдинг одной цепи, пришлось уменьшить порог размера кластера до 10 атомов. Были обнаружены кластеры на интерфейсе цепочек A и B (размером 13 атомов), цепочек A и D (размером 17 атомов), цепочек B и C (размером 17 атомов), цепочек C и D (размером 12 атомов).
 |
 |
|
|
 |
 |
|
|
| Семестр 7 |
© Дарья Потанина, 2020