Хемоинформатика¶
Задание: https://kodomo.fbb.msu.ru/wiki/2016/8/MolSim/task3
Цель занятия используя пакет моудлей RDkit предложить аналог ибупрофена:
- на сайте PubChem найти все радикалы c азидом для Click Chemistry и скачать их SMILES нотации или используйте pubchempy
- Найти формулу ибупрофена и предложить способ изменения его SMILES для эмуляции продукта Click Chemistry
- Заменить в найденых радикалах азидную группу на модифцированный ибупрофен.
- Превратить новые SMILES в объекты-молекулы
Отобрать те молекулы, которые удовлетворяют правилу пяти Lepinsky
Установим зависимости:
conda install -c rdkit rdkit conda install -c jirinovo pubchempy conda install -c conda-forge matplotlib conda install -c anaconda numpy# c̶o̶n̶d̶a̶ ̶i̶n̶s̶t̶a̶l̶l̶ ̶-̶c̶ ̶o̶m̶n̶i̶a̶ ̶n̶g̶l̶v̶i̶e̶w conda install nglview -c conda-forge
import ipywidgets
import nglview as nv
from rdkit.Chem.Draw import SimilarityMaps
from tqdm import tqdm, trange
import pubchempy as pcp
from pathlib import Path
from IPython.display import display, Image
import os
import sys
import time
import shutil
import asyncio
import numpy as np
from io import StringIO
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit import RDConfig
from rdkit.Chem.Draw import IPythonConsole, SimilarityMaps
from rdkit.Chem import Draw
import rdkit.Chem.Lipinski as Lipinksy
Найдем в PubChem SMILES ибупрофена и выведем его изображение.
smiles_string = 'CC(C)CC1=CC=C(C=C1)C(C)C(=O)O'
ibu = Chem.MolFromSmiles(smiles_string)
AllChem.Compute2DCoords(ibu)
display(ibu)
Посчитаем параметры Lipinski's rule of five (если молекула не удовлетворяет больше, чем одному такому правилу, она скорее всего не подойдет для использования как лекарства). Правила:
- Не более 5 доноров водородной связи;
- Не более 10 акцепторов водородной связи;
- Молекулярная масса не более 500 Da;
- Иметь коэффициент распределения вещества на границе раздела вода-октанол (log P) менее 5. Удовлетворяет ли им ибупрофен?
print(Lipinksy.NumHDonors(ibu))
print(Lipinksy.NumHAcceptors(ibu))
print(Lipinksy.rdMolDescriptors.CalcExactMolWt(ibu))
print(Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(ibu)[0])
Модификация ибупрофена для Click Chemistry¶
Ибупрофен удовлетворяет правилу пяти Липински. Попробуем провести с ибупрофеном реакцию азид-алкиновоего циклоприсоединения и проверить правила на полученном сединении.
# Продукт реакции азид-алкиновоего циклоприсоединения
tr = 'N1N=NC(=C1)C1=CC(CC(C)C)=CC=C1(C(C)C(=O)O)'
mol = Chem.MolFromSmiles(tr)
AllChem.Compute2DCoords(mol)
display(mol)
Создадим функцию, которая будет проверять наши производные на удовлетворение правилу пяти.
def is_valid_lipinsky(molecule):
return bool((Lipinksy.NumHDonors(molecule) <= 5)
and (Lipinksy.NumHAcceptors(molecule) <= 10)
and (Lipinksy.rdMolDescriptors.CalcExactMolWt(molecule) < 500)
and(Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(molecule)[0]) <= 5)
is_valid_lipinsky(ibu), is_valid_lipinsky(mol)
Хорошо, и ибупрофен и такой продукт удовлетворяет правилу пяти.
Скачивание азидов из PubChem¶
Попытаемся загрузить азиды с помощью https://pubchempy.readthedocs.io/en/latest/
compounds = []
# p1 = pcp.get_properties('CanonicalSMILES', 'N=N=N',
# 'smiles', searchtype='superstructure')
# p2 = pcp.get_properties('CanonicalSMILES', 'NN#N',
# 'smiles', searchtype = 'superstructure')
# compounds.extend(p1)
# compounds.extend(p2)
len(compounds)
compounds
REST API PubChem отвечает очень долго, так дело не пойдет, попросим датасет у однокурсников.
strings = np.genfromtxt('./pr3_data/dataset.txt', dtype=np.str)
smiles = []
for line in strings:
if len(line[1]) < 30 and not '.' in line[1]:
smiles.append(line[1])
len(smiles)
Замена в найденых молекулах азидной группы на модифцированный ибупрофен¶
Chem.WrapLogs()
sio = sys.stderr = StringIO()
mols = []
for smi in smiles:
if 'N=[N+]=[N-]' in smi:
newsmi = smi.replace('N=[N+]=[N-]', smiles_string)
else:
continue
# Новую молекулу лучше создавать в try из-за битых Smiles
try:
newmol = Chem.MolFromSmiles(newsmi)
if is_valid_lipinsky(newmol): # Проверка на правило пяти
mols.append(newmol)
except:
pass
len(mols)
Получаем некоторые возможные аналоги ибупрофена из найденных 7552. Выведем несколько примеров.
display(Draw.MolsToGridImage(
mols,
maxMols=9,
molsPerRow=3,
subImgSize=(400, 400))
)
Построим карты схожестей пары полученных производных с ибупрофеном.
similarity_data = [
SimilarityMaps.GetSimilarityMapForFingerprint(
ibu,
mol,
SimilarityMaps.GetMorganFingerprint
) for mol in mols[:2]
]
list(zip(*similarity_data))[1]
Веса вот такие. Не знаю, много это или нет, но остаток ибупрофена покрашен зеленым, а неибупрофен красным, значит программа, скорее всего, правильно все распознала.
Продолжим анализ в 3D¶
Установим nglview (https://github.com/arose/nglview)
conda install nglview -c conda-forge
jupyter-nbextension enable nglview --py --sys-prefixm3d = Chem.AddHs(mols[0])
Chem.AllChem.EmbedMolecule(m3d)
AllChem.MMFFOptimizeMolecule(m3d, maxIters=500, nonBondedThresh=200)
display(m3d)
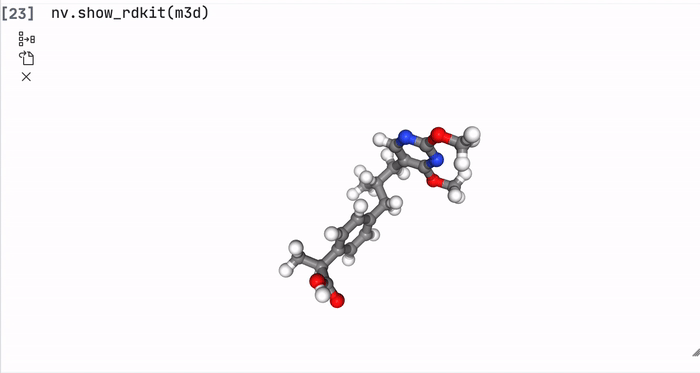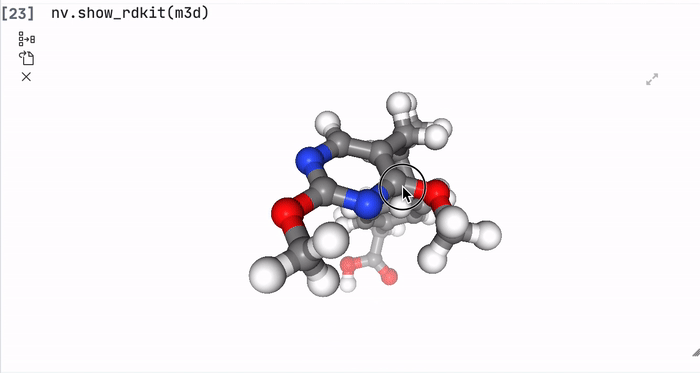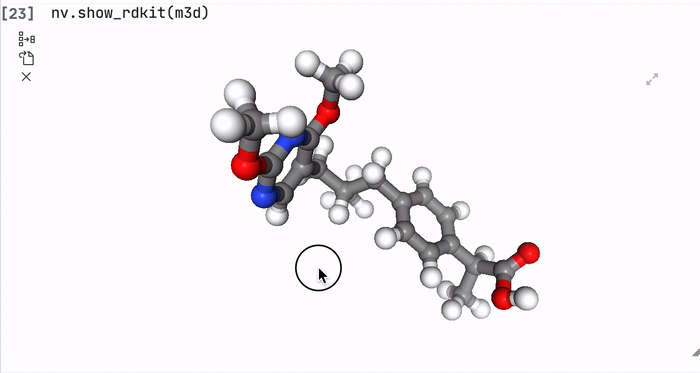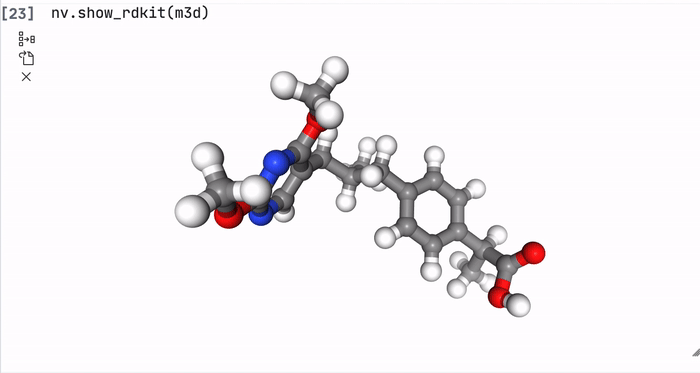
Например, вот такое производное в 3D выглядит так. Не знаю, зачем нужны два болтающихся кольца, так, на вид, оно не очень хорошо будет рабоать в организме. Попробуем это еще красивее и нагляднее изобразить.
nv.show_rdkit(m3d)
К сожаление в html этого не будет видно, поэтому запишем и вывесим gif.