Карта построена для последовательностей двух полипротеинов: вируса полиомиелита (P03301) и вируса ящура (P03311)
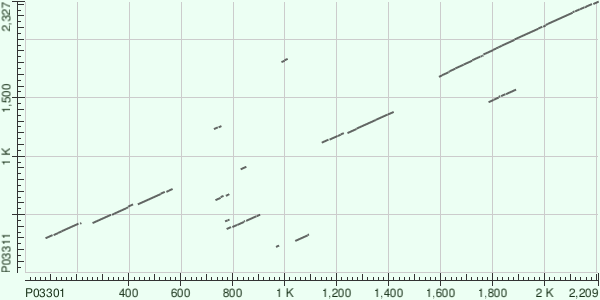
Лучшее выравнивание на карте - длинная полоса в правом верхнем углу, второе выравнивание - предыдущая полоса. Остальные "полосы" имеют меньший вес, хотя некоторые из них и более длинные.
| Identities | Positives | Length | Gaps | Score(bits) |
| 29% | 48% | P03301: 615(27.6%); P03311: 644(27.8%) | 59(8%) | 252 bits(643) |
| 37% | 52% | P03301: 277(12.5%); P03311: 263(11.3%) | 26(9%) | 159 bits(402) |
Зная координаты хорошо выравнившихся участков (в первых двух выравниваниях на обоих белках), можно узнать (на сайте UniProt в разделе Molecule processing для данных белков), в какие вирусные белки транслируются данные участки. В моем случае это Protein 3CD(P03301), RNA-directed RNA polymerase 3D-POL (P03311) для первого выравнивания и Protein 2C(P03301), Protein 2C(P03311) для второго.
В этом задании белок MotA_EColi выравнивается локально с одним гомологичным ему белком (MotA_Bacsu) и одним негомологичным (KAD_Bacsu), затем строятся "случайные выравнивания" - выравнивания тех же пар, где последовательность второго белка перемешана. Такое случайное выравнивание строится 100 раз, рассчитывается медианный score, score верхнего квартиля, битовый вес и p-value.
Так как у негомологичных белков битовый вес получился отрицательным, р-значение было посчитано не по формуле, а как доля выравниваний с весом больше данного среди 100 случайных выравниваний.
| пара белков | ID1, ID2 | вес выравнивания (S) | медиана весов случайных выравниваний (M) | верхний квартиль весов случайных выравниваний (Q1) | вес в битах (B) | p-value |
| гомологичные | MOTA_ECOLI, MOTA_BACSU | 240.5 | 47.0 | 52.5 | 36.2 | 1.2e-11 |
| негомологичные | MOTA_ECOLI, KAD_BACSU | 28.0 | 35.75 | 39.0 | -1.4 | 0.97 |
Первые два гомолога (две лучшие находки) белка AAN68968.1, найденные в Swiss-Prot:
| ID | AC | Organism | Identities | Positives | gaps | score | score(bits) | Expect | Coverage |
| PHYB_NOSS1 | Q9R6X3.1 | Nostoc sp. PCC 7120 | 35% | 54% | 18(3%) | 309 bits(792) | 309 bits(792) | 3e-92 | 61% |
| BPHY_PSEAE | Q9HWR3.1 | Pseudomonas aeruginosa PAO1 | 36% | 52% | 27(4%) | 289 bits(739) | 289 bits(739) | 8e-85 | 61% |
Вернуться на страницу семестра
© potapenko 2017-2018