Для выполнения практикума был выбран ген SSB, кодирующий белок Lupus La protein (не путать с SSBP -single strand binding protein). Одна из функций этого белка - защищать 3' конец продукта РНК-полимеразы III от экзонуклеазного расщепления. Предположительно играет роль в терминации трансляции, как-то влияет на IRES-опосредованную трансляцию в случае заражения вирусами, для которых характерны IRES (?). Название получил из-за своего взаимодействия с аутоантителами при синдроме Sjogren.
Ссылка на Lupus La protein в UniProt.
Таблица 1. Альтернативные транскрипты гена SSB.
| Gencode Transcript альтернативного продукта | координаты на хромосоме | экзонов |
| ENST00000260956.8 | hg38 chr2:169,798,812-169,812,064 | 12 (из них 11 кодирующих) |
| ENST00000409333.1 | hg38 chr2:169,799,279-169,812,064 | 12 (из них 11 кодирующих) |
С гена транскрибируются два альтернативных транскрипта, каждый содержит 12 экзонов, при этом первые экзоны у них разные, остальные 11 совпадают. При этом эти два транскрипта кодируют один и тот же белок, т.е. первый экзон у каждого транскрипта некодирующий.
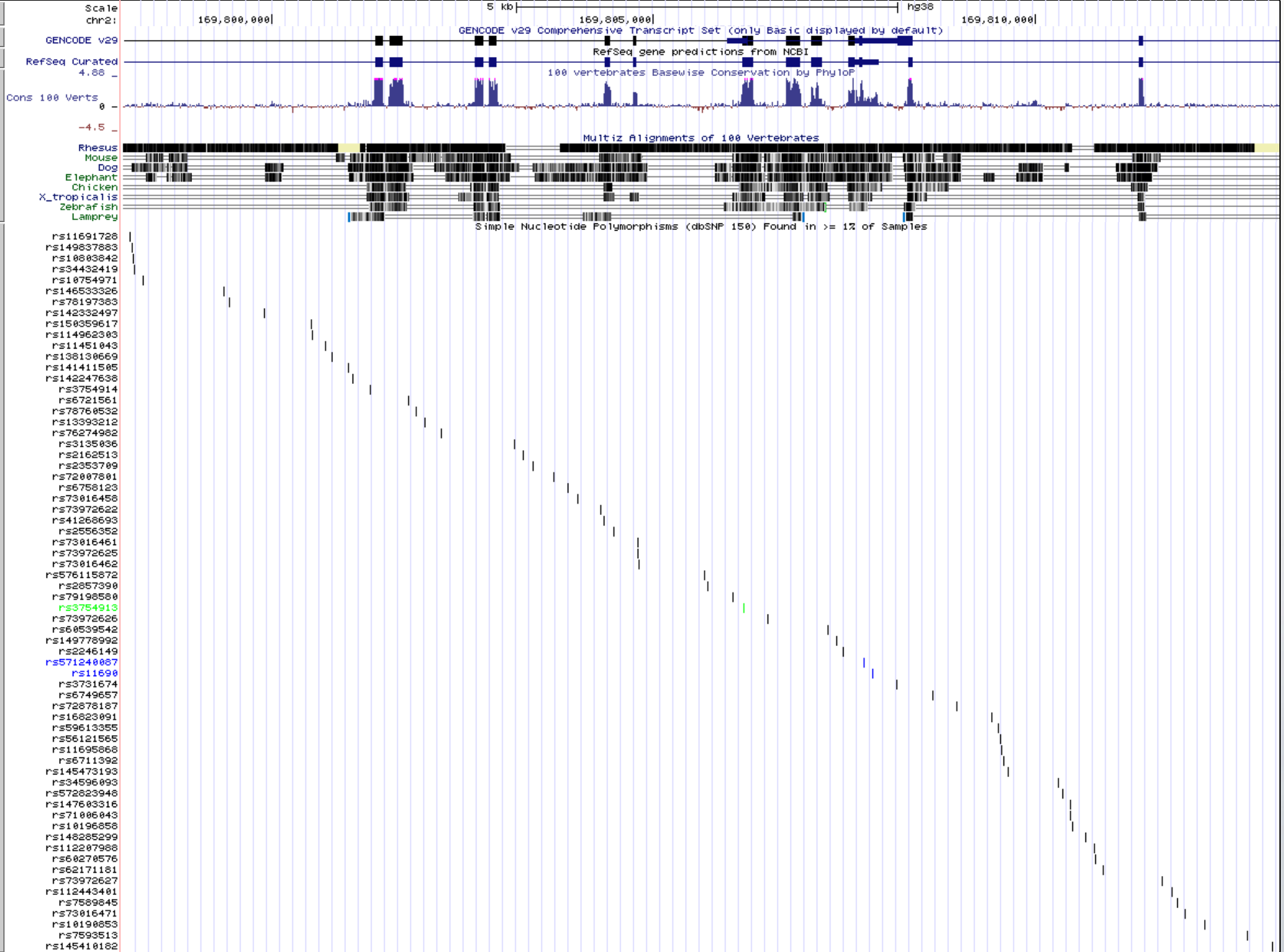
Рисунок 1. Окрестность гена SSB из UCSC genome browser
Update. В геномном браузере Ensembl нашлись еще транскрипты SSB, в том числе еще три белоккодирующих. Правда, их нет в RefSeq, а соответствующие белки в базе UniProt имеют статус Unreviewed.
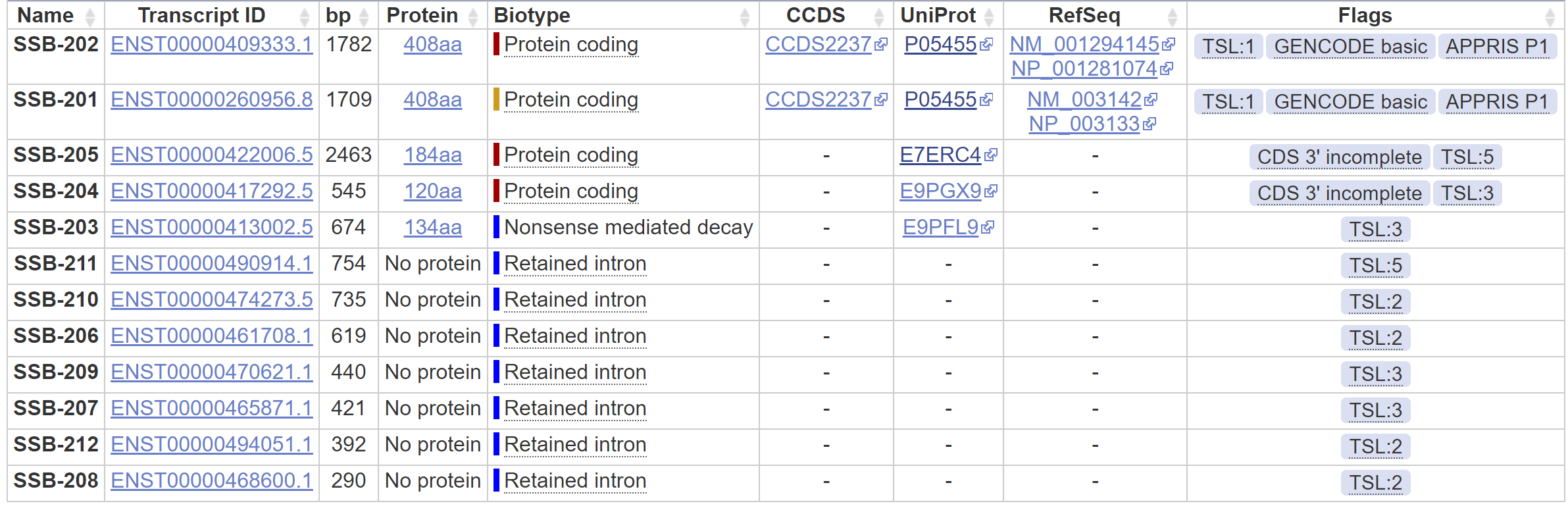
Рисунок 2. Транскрипты гена SSB, указанные в Ensembl.
Тот же ген SSB был найден в геномном браузере ENSEMBL. Там же было построено выравнивание гена SSB человека и шимпанзе. Выравнивание было скачано и с помощью команды distmat пакета EMBOSS было посчитано количество различий. Описание программы distmat указывает, что она строит матрицу различий между последовательностями (матрица квадратная, размером с количество последовательностей в выравнивании, с нулями на диагоналях (логично, что расстояние между последовательностью и ей же самой ноль)). В моем случае из файла меня интересует только одно число - расстояние между последовательностями гена SSB человека и шимпанзе. Это расстояние может быть посчитано разными способами, в простейшем случае - по такой формуле:
S = m/(npos + gaps*gap_penalty) m - score of matches (1 for an exact match, a fraction for partial matches and 0 for no match) npos - number of positions included in m gaps - number of gaps in the sequences gap_penalty - the score given to a gapped position D = uncorrected distance = p-distance = 1-S
Мне не совсем понятно, почему так можно, но в описании сказано, что расстояние между последовательностями можно понимать как количество замен на 100 нуклеотидов. В случае с геном SSB D = 0.78. Длина выравнивания 20198 нуклеотидов. Значит, всего замен 20198 * 0.78 / 100 = 158 штук.
По данным Variation table (Ensembl) в человеческом гене SSB всего 1351 SNP (именно замен, а не инсерций и делеций). Если рассматривать только полиморфизмы, встречающиеся с вероятность 0.015 - 0.5, их уже 512, то есть медианное значение вероятности замен меньше 0.015. Кажется, что это довольно много: так считать нельзя, но можно посчитать довольно грубую оценку сверху. Представим, что среднее значение не сильно отличается от медианы, и тогда у одного человека встречается 0.015*1351 = 20 замен. Большинство этих полиморфизмов находится в некодирующих участках гена.
В любом случае, в среднем между двумя людьми отличий в этом гене (как и в любом другом) будет меньше, чем между шимпанзе и человеком. Это не отменяет возможности подобрать такую пару человеческих последовательностей этого гена, что они будут отличаться между собой больше, чем одна из этих последовательностей от последовательности этого гена шимпанзе. Такую "человеческую" последовательность составить можно, но вероятность встретить человека с такой последоватльностью очень мала.
Вернуться на страницу семестра
© potapenko 2017-2018