Построение А-формы было выполнено так: fiber -a -seq='tcagtcagtcagtcagtcag' tcag-a.pdb. Построение В-формы было сделано аналогично. Для Z-формы программа fiber умеет строить только полимер из GC-повторов и полимер из d(As4T) (см. рисунок 1). Поэтому после параметра -z программа игнорирует параметр -seq и сторит GC-полимер.
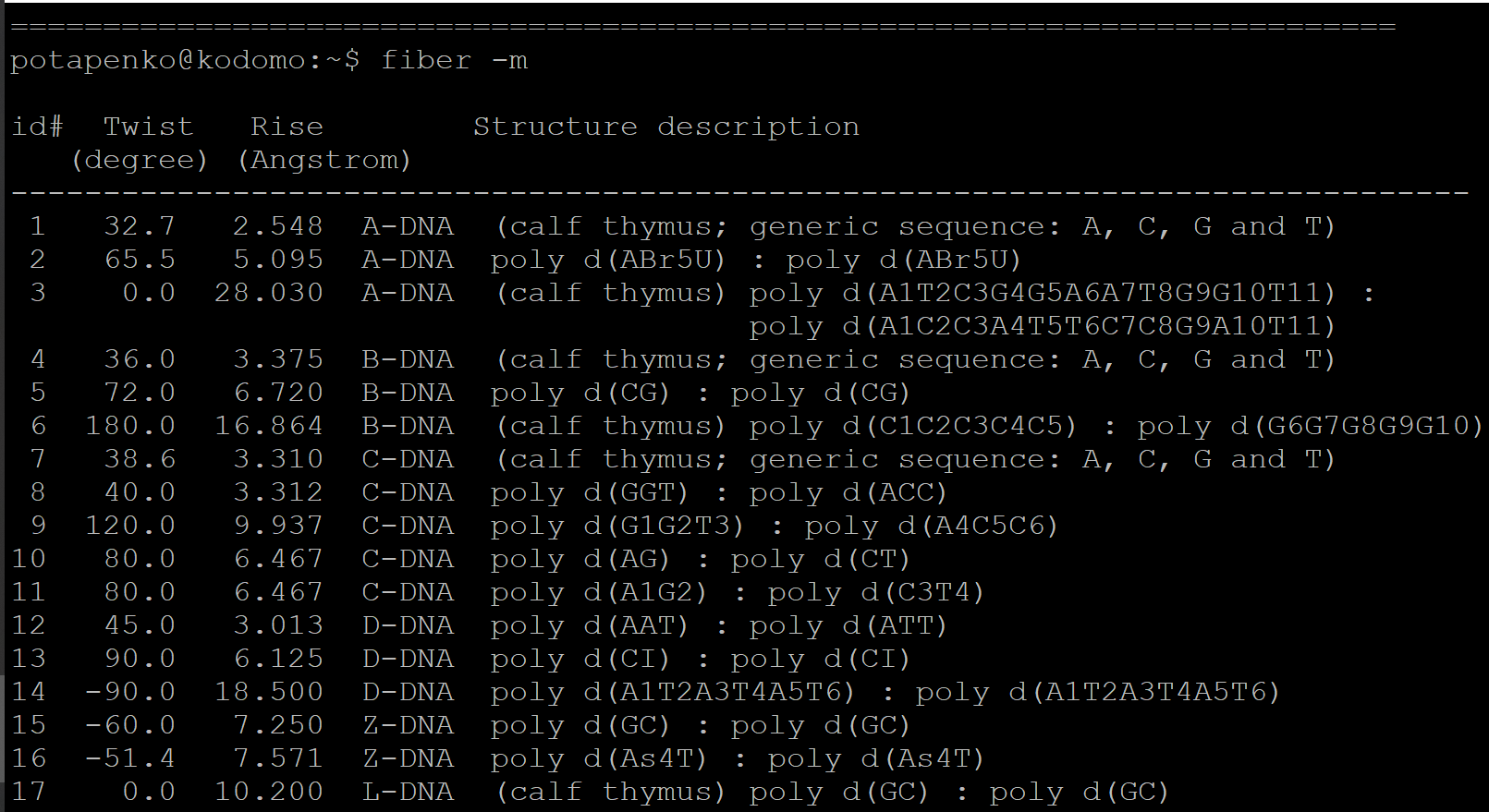
Рисунок 1: fiber может построить модели разных форм ДНК. Нас интересуют сроки 1, 4 и 15. Видно, что Z-формы всего две (строки 15 и 16), и обе они имеют заранее определенный состав.
[Правая спираль - это правый винт, т.е. если смотреть с любого торца спирали, идущие "на нас" крайние витки будут закручены в положительном направлении.]
["Ширина бороздки - это расстояние от атома фосфора данного нуклеотида до некоторого фосфора комплементарной цепи - такого, что расстояние до соседних фосфоров несколько больше"; т. е. если померить расстояния от выбранного фосфора до всех фосфоров в другой цепи, два локальных минимума и будут ширинами бороздок (?).]
В выбранном основании (тимин [DT]20:B в экспериментальной модели B-формы с pdb_id = 1bna):
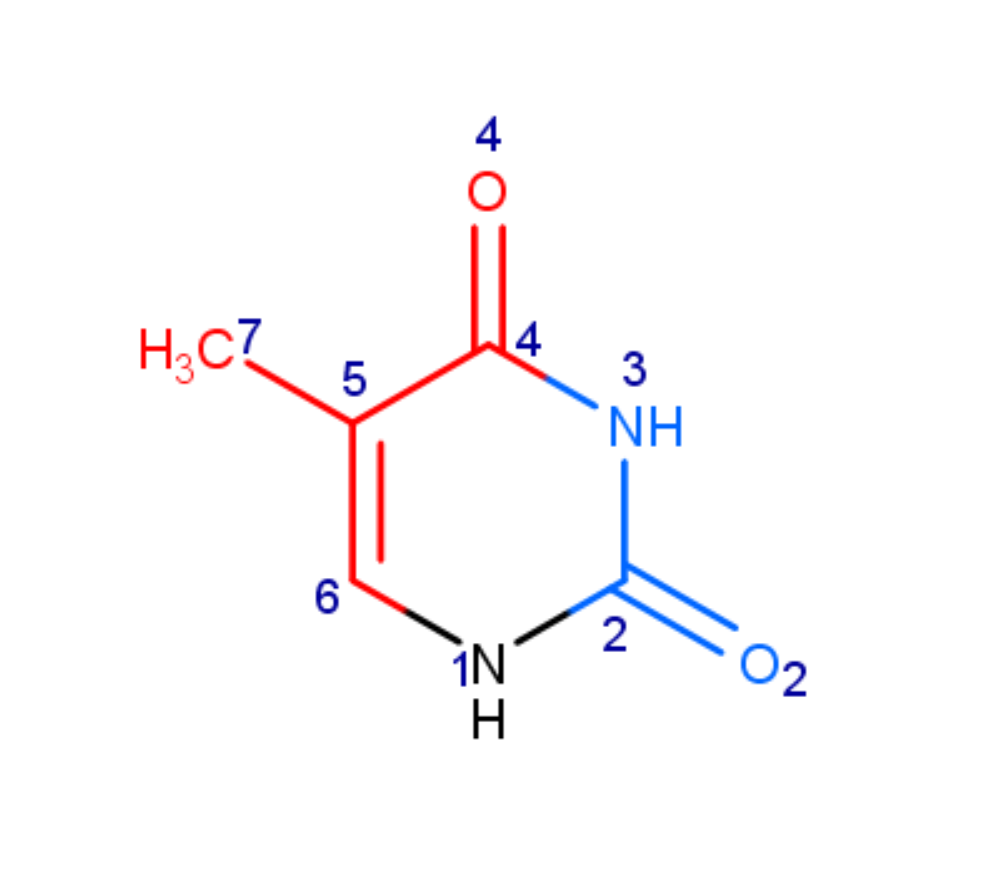
Рисунок 2. Тимин. Красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
Таблица 1. Формы ДНК.
| А-форма (3v9d) | В-форма (1bna) | Z-форма (1tne) | |
| тип спирали | правая | правая | левая |
| шаг спирали (A) | 29.17-30.89* | 30.08 | 43.50** |
| число оснований на виток | 11 | 10 | 12** |
| ширина большой бороздки (A) | 11.97 [DT]7:A.P - [DC]3:B.P | 18.10 [DT]7:A.P - [DG]16:B.P | 8.19 [8MG]4:A.P-[DG]12:B.P |
| ширина малой бороздки (A) | 15.74 [DT]7:A.P - [DC]10:B.P | 11.06 [DT]7:A.P - [DG]22:B.P | 15.72 [DC]3:A.P-[DC]9:B.P |
* Значение зависит от того, шаг между какими атомами измерять. Данный фрагмент содержит 13 атомов фосфора. Значит, шагом спирали будет как расстояние между фосфорами 1 и 12, так и между фосфорами 2 и 13. Расстояния равны 2.917 и 3.089, что не позволяет сравнить их с шагом спирали В-формы. Я связываю это с тем, что трехмерная структура была получена для небольшого фрагмента, а измерения проводятся по краям структуры. Данные презентации говорят, что шаг спирали у А-формы меньше, чем у В-формы.
** экспериментальный участок слишком короткий, чтобы провести такие измерения, поэтому цифры даны по измерениям сгенерированного участка.
В упражнении требовалось на основании значений торсионных углов тРНК (в моем случае с pdb_id = '1gts') определить, на какую из форм ДНК больше похожа тРНК. Для этого были усреднены значения всех торсионных углов для каждого нуклеотида в тРНК и в A- и В-формах ДНК (Сравнение с z-формой не производилось, потому что структура тРНК - правая спираль содержащая в том числе и А-Т пары, поэтому на Z-форму точно похожа меньше, чем на А или В.
Для каждого основания в цепи была получена большая таблица с торсионными углами с помощью конвейера из find_pair и analyze. После этого значения для каждого угла каждого нуклеотида в каждой из трех (1gts, 1bna, 3v9d) структур были усреднены. Потом была составлена таблица 2 (см. ниже) с абсолютными значениями разностей всех торсионных углов всех типов нуклеотидов из тРНК и В-формы ДНК (первые 4 строки) и из тРНК и А-формы ДНК (следующие 4 строки). Усреднение проводилось с помощью Excel, потом таблицы переводились на html с помощью питона, всовывающего тэги вместо пробелов.
Таблица 2. Сравнение торсионных углов. полная таблица на гугл-диске
| dfve | alpha | beta | gamma | delta | epsilon | zeta | chi |
| diff_1gtc_1bna_A | 2,8 | 11,1 | 16,6 | 34,7 | 264,4 | 27,3 | 30,0 |
| diff_1gtc_1bna_C | 19,1 | 3,6 | 21,6 | 36,2 | 15,1 | 47,5 | 38,2 |
| diff_1gtc_1bna_G | 54,7 | 49,8 | 22,1 | 39,8 | 140,6 | 45,2 | 5,6 |
| diff_1gtc_1bna_T | 11,6 | 14,3 | 14,5 | 27,6 | 312,6 | 3,5 | 0,6 |
| diff_1gtc_3v9d_A | 77,4 | 182,9 | 87,4 | 7,6 | 84,0 | 23,3 | 6,2 |
| diff_1gtc_3v9d_C | 37,1 | 8,5 | 6,4 | 2,5 | 59,5 | 17,0 | 0,5 |
| diff_1gtc_3v9d_G | 3,7 | 6,9 | 49,5 | 2,4 | 0,4 | 14,2 | 10,9 |
| diff_1gtc_3v9d_T | 24,1 | 63,3 | 5,4 | 2,1 | 33,4 | 16,7 | 33,2 |
Видно, что цифры в последних четырёх строках "в среднем" меньше (среднее значение всех чисел в верхних четырех строках - 46.8, в нижних - 30.9), поэтому я делаю вывод, что данная тРНК по вторичной структуре больше похожа на А-ДНК, если сравнение ведётся по всем торсионным углам.
Впрочем, различие не очень большое, a взятые участки нуклеиновых кислот не очень большие, содержат разные доли типов нуклеотидов, а значения торсионных углов сильно варьируют даже внутри одного типа нуклеотидов в одной структуре.
Из литературных источников (Нельсон, Кокс) известно, что преобладающая двунитевая структура РНК - это правозакрученная спираль в А-форме. В лабораторных условиях для двунитевой РНК была получена Z-форма, В-форма для РНК не найдена.
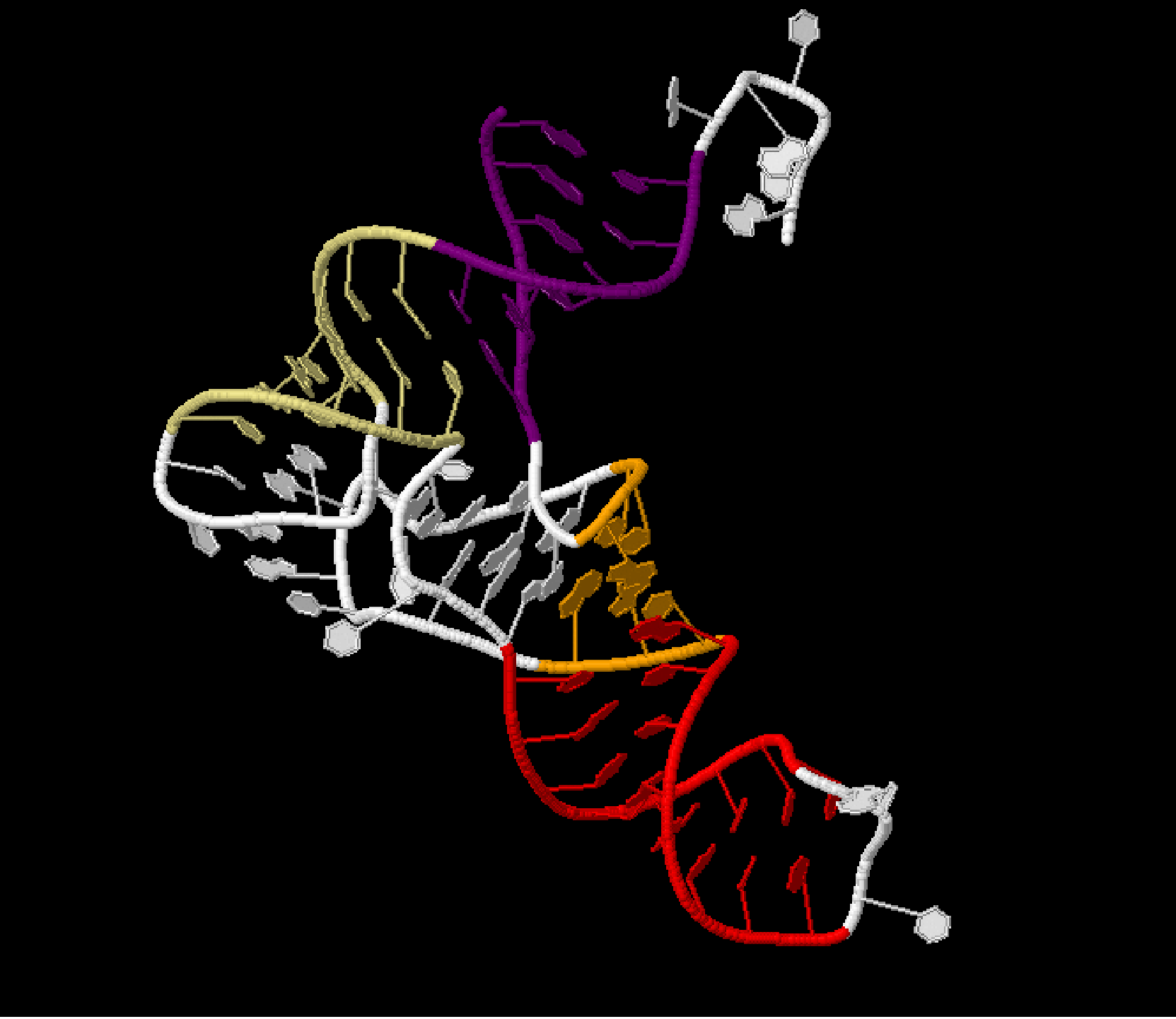
Транспортные РНК имеют специфическую структуру, в которую входят 4 плеча (аминокислотное, антикодоновое, D-плечо и риботимидиновое). Некоторые остатки содержат измененные основания и сахара. Определить координаты стеблей (в случае тРНК - плечей во вторичной структуре РНК) можно с помощью того же конвейера из find_pair и analyze. Программа выдает пары оснований, между которыми есть водородная связь. Среди этих пар нужно выбрать подряд идущие, это и есть стебли. Вот их координаты:
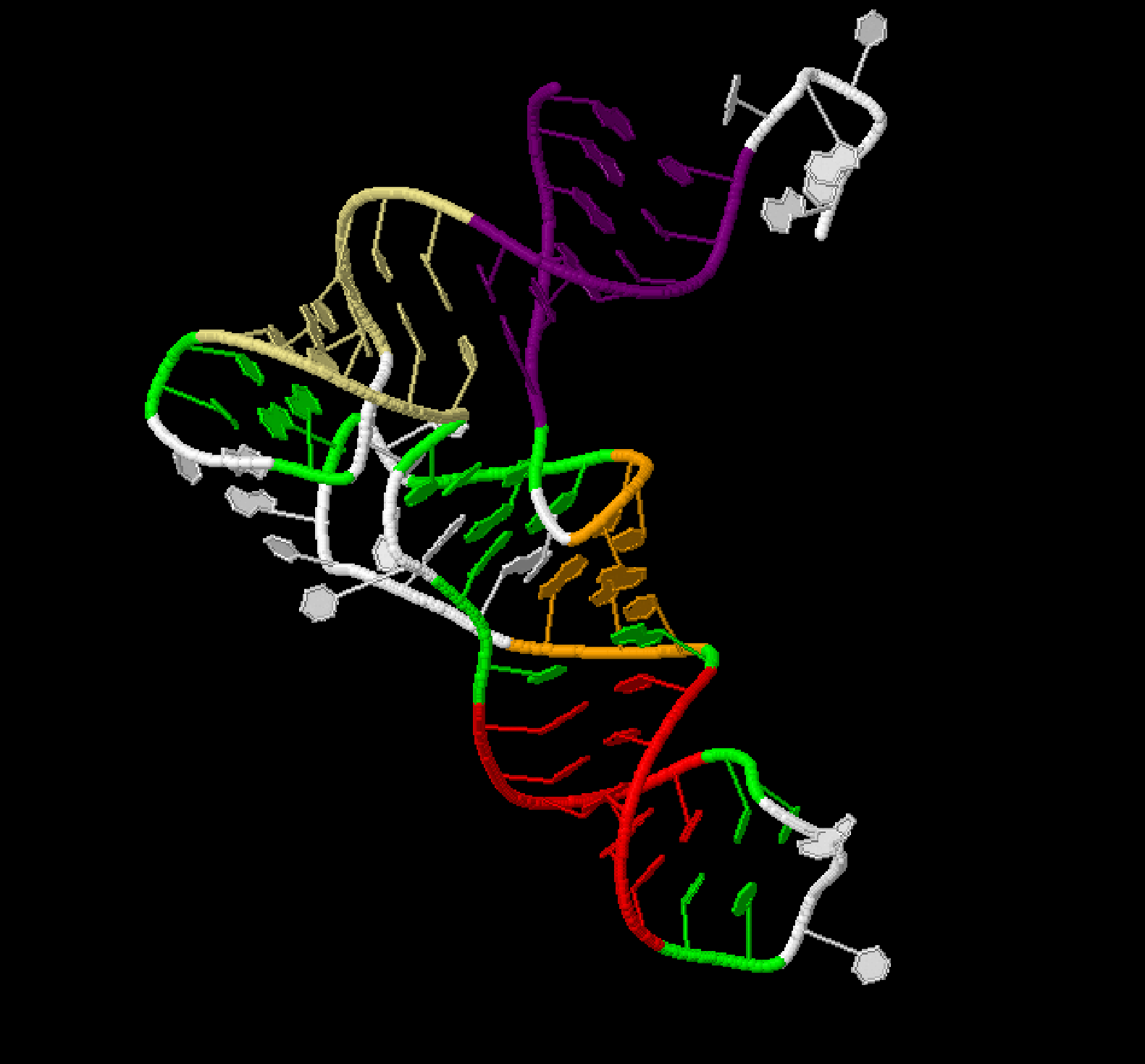
Всего в структуре найдено 28 водородных связей между основаниями, из них 8 - неканонические. Количество неканонических пар в структуре выдает программа analuze, искать эти пары нужно вручную, просматривая характеристики найденных водородных связей. На рисунке 4 неканонические пары отмечены ярко-зеленым.
 |  |
| Рисунок 3: стебли тРНК | Рисунок 4: неканонические пары в тРНК. |
Среди найденных водородных связей есть одиночные, не входящие в состав стеблей. Эти пары предположительно помогают поддерживать третичную структуру. Таких пар всего 5, они выделены темно-зеленым на рисунке 5.
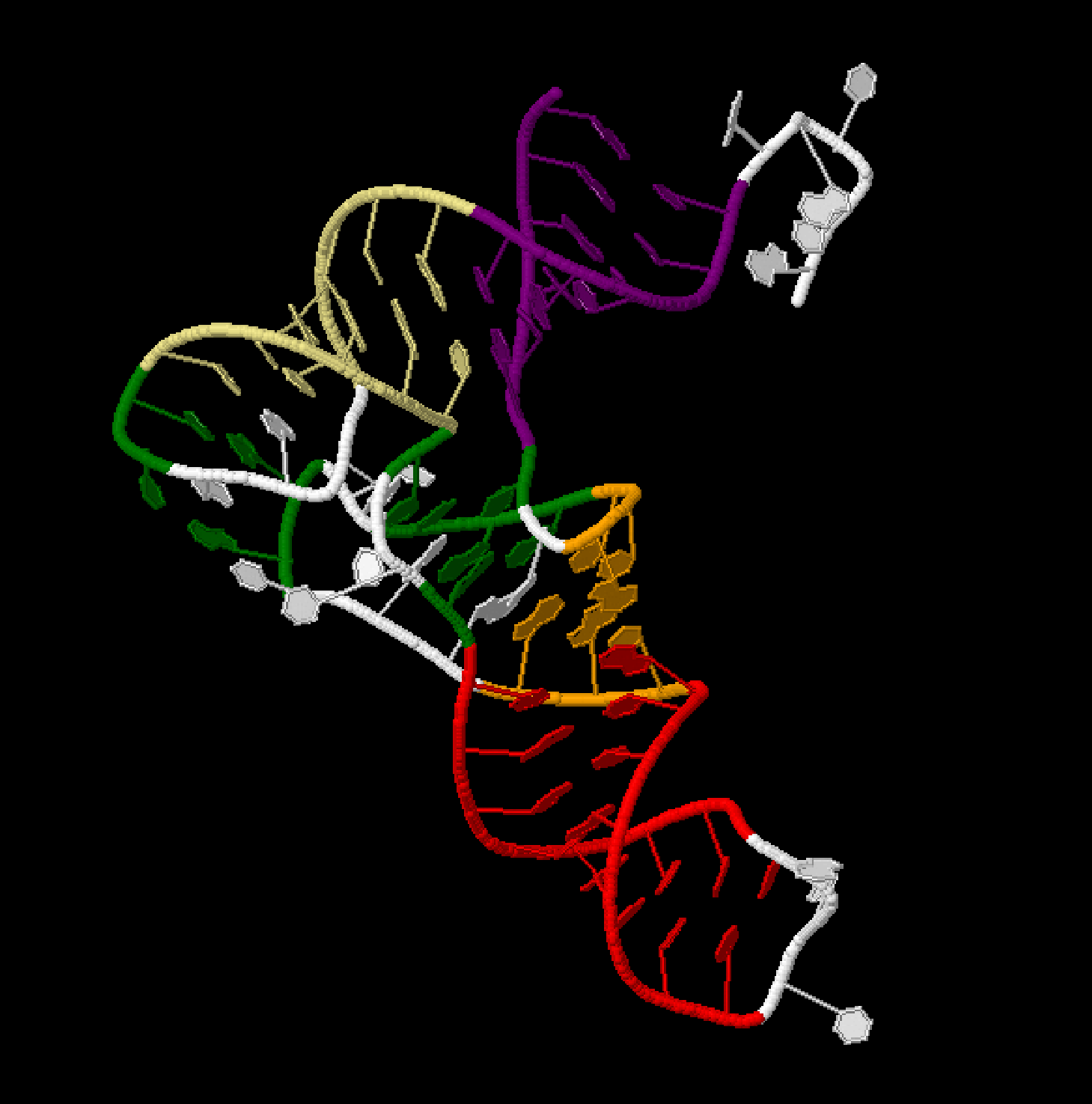
Рисунок 5. Пары, поддерживающие третичную структуру тРНК.
В файле , полученном программой analyze, можно также найти площади перекрывания соседних пар оснований (в квадратных ангстремах), наибольшее значение перекрывания у пары пар оснований G53~A58 и U54~C61. Интересно, что эти две пары не образуют общий стебель.
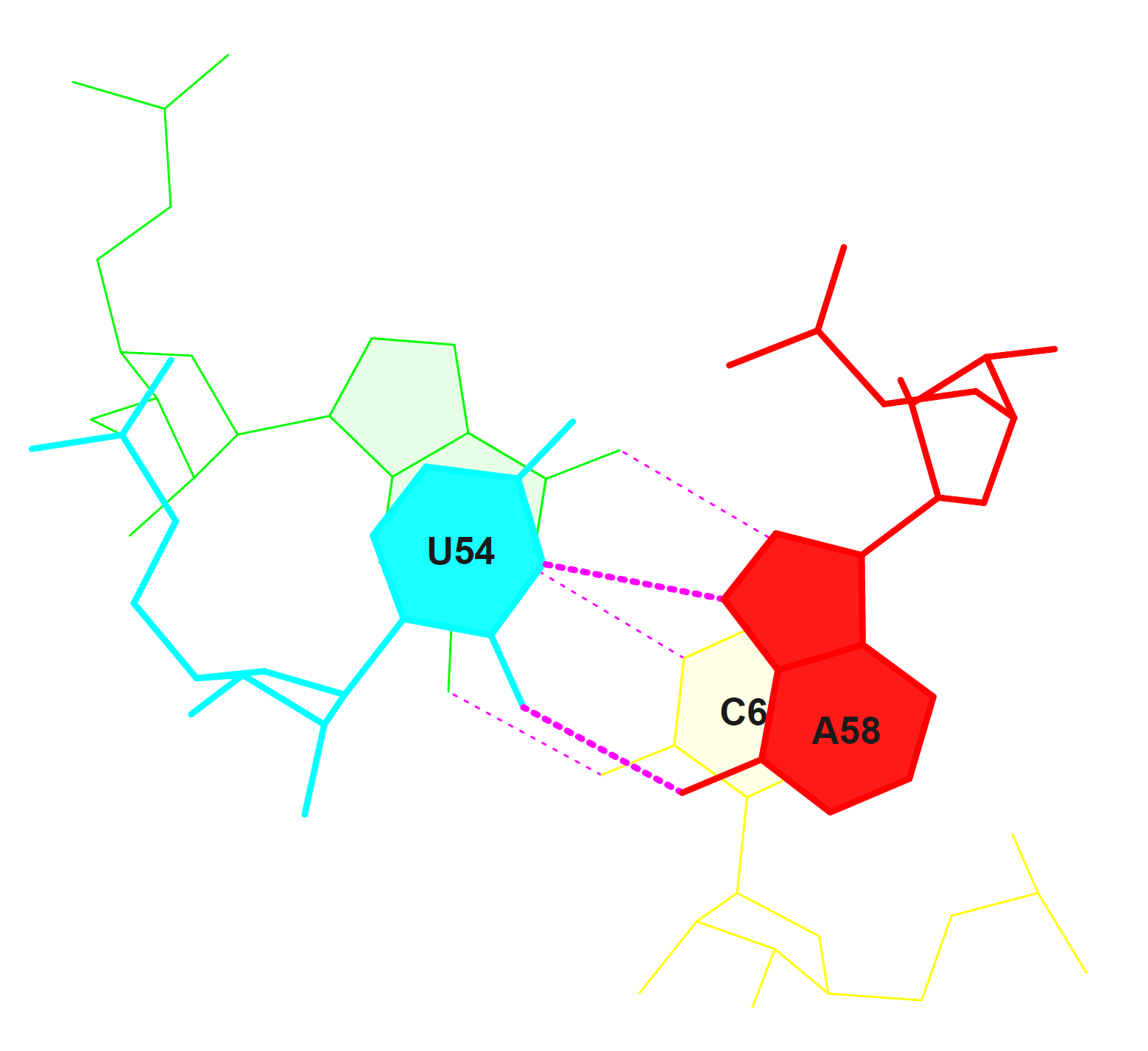
Рисунок 6. Пара пар с наибольщей площадью перекрывания (наиболее сильным стекингом), общепринятое изображение построено с помощью ps2img и переведено в удобный формат с помощью ps2pdf.
Вернуться на страницу семестра
© potapenko 2017-2019