Исходный файл с прямой последовательностью (название укорочено, оригинальное название WSWS2960_COI_F_G11_WSBS-Seq-1-08-15.ab1)
Исходный файл с обратной последовательностью (название укорочено, оригинальное название WSWS2960_COI_R_G12_WSBS-Seq-1-08-15.ab1)
Характеристика хроматограммы прямой цепи.
При просмотре в Chromas (Lite) начало выделено желтым (т. е. признано программой ненадежным) до 25 нуклеотида, я удалила эту часть последовательности и еще несколько нуклеотидов - до 28го включительно. После выравнивания с обратной цепью стало ясно, что в принципе можно было оставить в начале чуть больше нуклеотидов: пусть они не так хорошо читаются по прямой цепи, зато отлично считываются на обратной цепи. Удалено 31 нуклеотидов, из которых 11 были как-то распознаны программой. Конец последовательности выделен желтым с 650го нуклеотида (в новой нумерации после удаления начала), я удалила с 647, т. к. позиция 647 стояло N, "на глаз" по хроматограмме нельзя было однозначно определить нуклеотид, а комплементарного участка обратной цепи на это место нет. Удален 41 нуклеотид, из которых 29 были как-то распознаны программой. Конечно, уже начиная с ~550 нуклеотида хроматограмма не такая "хорошая" (уровень шума выше, здесь и находится большая часть проблемных нуклеотидов), как в начале и середине, но с помощью обратной цепи последовательность читается.
Чтобы оценить соотношение сигнала и шума, удобнее сжать хроматограмму по оси абсцисс. У меня плохой глазомер, так что я указывала мышкой на пики шума, смотрела ординату и сравнивала ее с ординатой сигнала. Отношение сигнал % шум получается 6-8 (к концу последовательности уменьшается).
Характеристика хроматограммы обратной цепи.
Координаты обратной цепи удобнее указывать по прямой цепи, сразу выполним переход к комплементарной ей цепи. Начало (в новой системе отсчета) желтое до 5 нуклеотида, я удалила до 13 включительно, среди 13 удаленных нуклеотидов 11 были как-то распознаны программой. Конец желтый с 663 нуклеотида, так и удаляю 44 нуклеотида, из которых 13 были распознаны автоматически. Можно заметить, что для обратной цепи ее конец (с точки зрения секвенирования и исходного файла - начало) имеет заметно более длинный ненадежный участок, чем у прямой цепи.
На обратной цепи шум в среднем ниже (если исключить концевые участки), но несколько больше выбивающихся высоких пиков. В среднем пики примерно той же высоты.
Редактирование последовательностей.
После того, как плохо читаемые концы были удалены, каждая хроматограмма была просмосмотрена, были внесены изменения там, где это было возможно без сравнения со второй цепью. После этого последовательности прямой и обратной цепи были выровнены (needle) и были внесены изменения, требущие сравнения со второй цепью, и проверены изменения, внесенные до выравнивания.
Всего было внесено 21 изменение: исправлены на 6a, 4t, 4g, 4c, 2 нуклеотида был удалены, один нуклеотид заменен на код полиморфизма r (это все исправления, не считая обрезания концов). Изменения вносились маленькими буквами, число маленьких букв в последовательности легко узнать в текстовом редакторе или с помощью python.
В обозначенных пределах мне попались проблемные нуклеотиды, которые нельзя было бы проверить по второй цепи. Они относятся к концу прямой цепи и началу обратной; в нескольких таких позициях я заменила N на нуклеотид, пик которого заметно выше на хроматограмме, а в позиции 25 обратной цепи оставила букву R, т.к. в этой позиции пики аденина и гуанина примерно одинаковые, невысокие, но выше уровня шума. Без обратной последовательности нельзя точно сказать, полиморфизм это или сигналом является только один из пиков, а второй - шум.
Результаты.
Консенсусная последовательность , полученная по выравниванию программой consambig пакета EMBOSS.
Отредактированные последовательности на хроматограммах: прямая и обратная
Отредактированные последовательности в fasta формате: прямая и обратная
JalView-проект с выравниванием прямого и обратного прочтений
Примеры исправлений.
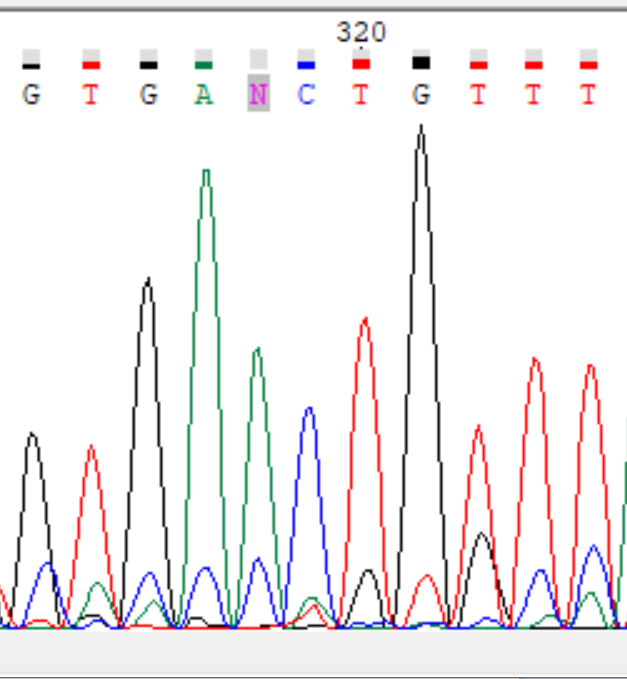
 | Прямая цепь, позиция 318. На хроматограмме достаточно явный пик с невысоким уровнем шума цитозина, стоит N. Исправлено на аденин, проверено по обратной цепи. |
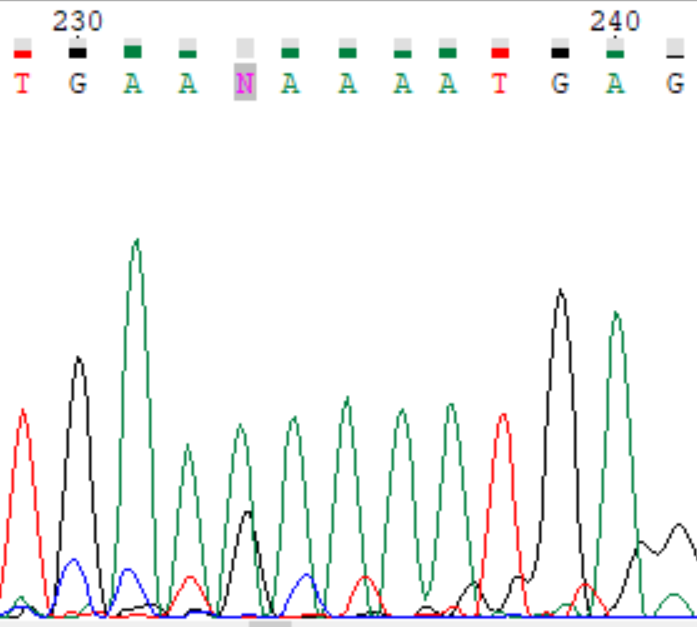
 | Прямая цепь, позиция 233. Пик аденина выше пика гуанина, но это может быть обусловлено тем, что соседние 6 нуклеотидов тоже аденины. На обратной цепи здесь аденин, исправляем на А. |
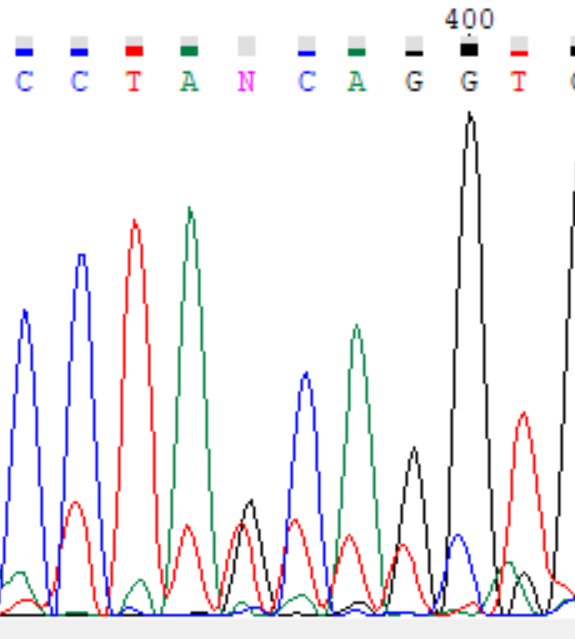
 | Прямая цепь, позиция 396. Пики тимина и гуанина примерно одинаковой высоты, оба довольно низкие. Может быть, это полиморфизм? Но нет, сравниваем с обратной цепью, исправляем на гуанин. |
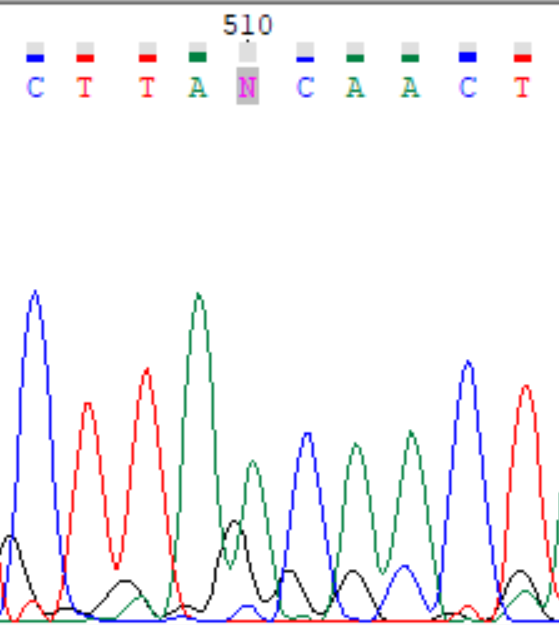
 | Прямая цепь, позиция 510. На пик аденина "наползает" сравнимый по высоте пик гуанина. На обратной цепи хорошо читается аденин, исправляем. |
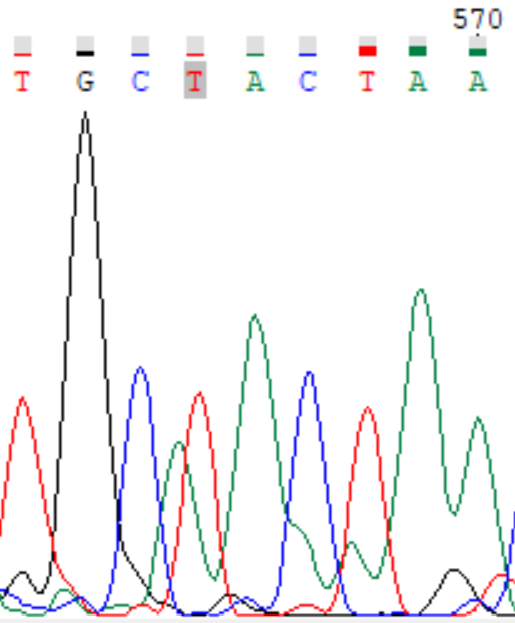
 | Прямая цепь, позиция 565. Между соседними невысокими пиками цитозина и тимина вклинивается пик аденина сравнимой высоты. На всякий случай проверяем по обратной цепи, там идут подряд цитозин и тимин, добавочного нуклеотида нет, исправления не требуются. |
Пример нечитаемого фрагмента хроматограммы.
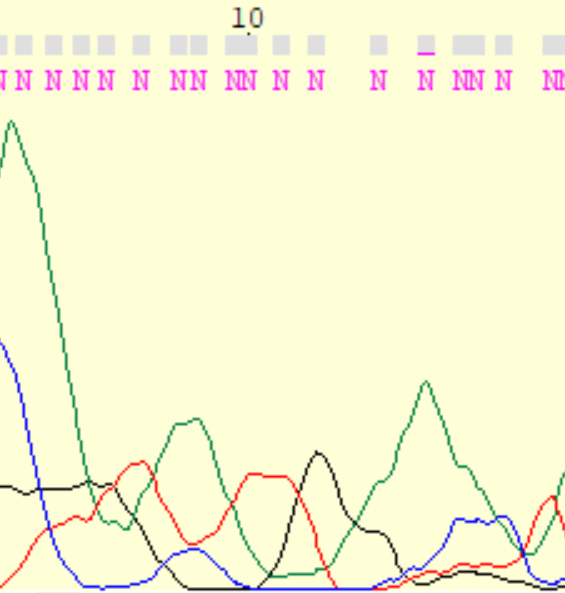 | Пример 1: это самое начало хроматограммы прямой последовательности. Пики очень широкие, шириной в несколько нуклеотидов, и нехарактерной формы. Скорее всего, это пятна краски (характерно для самого начала и самого конца хроматограмм). |
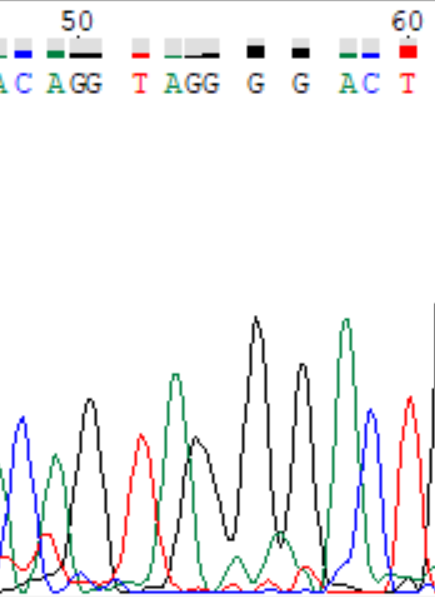 | Пример 2: участок хроматограммы прямой последовательности. Шум низкий, но расстояния между пиками неравные, нельзя сказать даже, сколько в этом участке нуклеотидов. Последовательность этого участка была восстановлена с использованием обратной цепи. |
Вернуться на страницу семестра
© potapenko 2017-2018