Примечание: я выполняла задания прошлого года по той же теме. Ссылка на страницу с условиями заданий.
1. PSI-BLAST.
Для работы была выбран идентификатор AC = P17265. Он соответствует белку Ribosome hibernation promotion factor, кодируемому геном hpf, из организма Rhizobium meliloti (альфапротеобактерия). Этот белок вместе с белком ribosome modulation factor (RMF) задействован в димеризации 70S-рибосом в 100S-рибосомы в течение стационарной фазы; Димеры рибосом неактивны с точки зрения трансляции, но их численность иногда растёт экспоненциально; видимо, димеризация используется для регуляции численности активных рибосом в клетке (?, не разобралась).
Для составления семейства гомологов данного белка был использован blast по алгоритму psi-blast (Position-Specific Iterative BLAST). Этот алгоритм способен находить дальних родственников заданного белка благодаря использованию PSSM. Процесс происходит итеративно, и уже после первой итерации для поиска вместо BLOSUM используется PSSM (сделанная по выравниванию находок от первой отерации). Кажется, что если бы банк последовательностей был устроен случайно, "сходила бы лавина" и количество находок после каждой итерации росло и в конце концов собрался бы весь банк, но пространство последовательностей в банке устроено разумно, и через сколько-то итераций поиск заканчивается.
Все параметры были оставлены по умолчанию, порог (treshold) = 0.005. Результаты каждой итерации записаны в таблице 1.
Таблица 1. Итерации psi-blast.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1. | 17 | P0A147.1 | 7e-04 | P26983.1 | 0.027 |
| 2. | 27 | P33987.1 | 1e-08 | P9WMA8.1 | 0.015 |
| 3. | 28 | P9WMA8.1 | 0.002 | Q6P9R4.3 | 0.013 |
| 4. | 28 | P24694.1 | 3e-18 | P27321.3 | 0.22 |
| 5. | 28 | P24694.1 | 2e-18 | P27321.3 | 0.19 |
Во второй итерации добавилось 10 новых белков, после третьей - только один. В четвёртой итерации новых белков не добавилось, но (из-за того, что E-value считался по немного другой матрице) порядок белков в конце списка изменился, и увеличился разрыв E-value между первой находкой после порога и последней находкой до порога, что также явзяется признаком выделения семейства гомологичных белков. Т.к. матрица, используемая в пятой итерации, была построена по тем же 28 последовательностям, что и матрица для четвёртой итерации, логично, что список находок не изменился.
Из найденных 28 белков подавляющее большинство (24) имеет то же название (HPF), что и исходный белок, но принадлежат другим организмам. Организмы очень разные, большинство - бактерии, но нашлись HFP из дрожжей и серой крысы (!). Другие 4 белка - хлоропластный Ribosome-binding factor PSRP1 (ингибирует трансляцию), Dormancy associated translation inhibitor (тоже ингибирует трансляцию), и Ribosome-associated factor Y (дважды, из описания его функции: "During stationary phase prevents 70S dimer formation"). Видно, что эти белки функционально похожи на HPF.
Поиск паттерна.
Во втором практикуме я работала с выравниванием последовательностей фермента енолазы (мнемоника ENO) из семи видов протеобактерий. В этом задании нужно было уточнить паттерн, присутствующий в генах енолазы, взятый из базы Prosite . Уточнить нужно для протеобактерий, но, так как эти семь бактерий относятся к разным группам протеобактерий (два представителся гаммапротеобактерий, два - бетапротеобактерий и три - альфапротеобактерий), использоваться будут последовательности енолазы только этих семи организмов.
Сам паттерн (который потом нужно будет уточнять) был найден на сайте Prosite сканированием последовательности енолазы из E.coli (которая тоже является протеобактерией, но это не имеет значения, ведь Prosite ищет неспецифичные для таксонов паттерны). Единственный найденный паттерн имеет идентификатор PS00164 и название Enolase signature. Паттерн - это выражение (запись) мотива, где в каждой позиции перечислены возможные нуклеотиды. Найденный паттерн имеет вид:
[LIVTMS]-[LIVP]-[LIV]-[KQ]-x-[ND]-Q-[INV]-[GA]-[ST]- [LIVM]-[STL]-[DERKAQG]-[STA]
В последовательности ENO_ECOLI сигнал имеет вид ILIKfNQIGSLTET, соответствует координатам 339 - 352. Стоит заметить, что пятая аминокислота в этом сигнале обозначена маленькой буквой - это означает, что эта позиция не является частью сигнала, в паттерне это место обозначено знаком "х", на это месте может быть любая аминокислота. По координатам можно найти сигналы в выравнивании семи последовательностей, открытом в JalView:
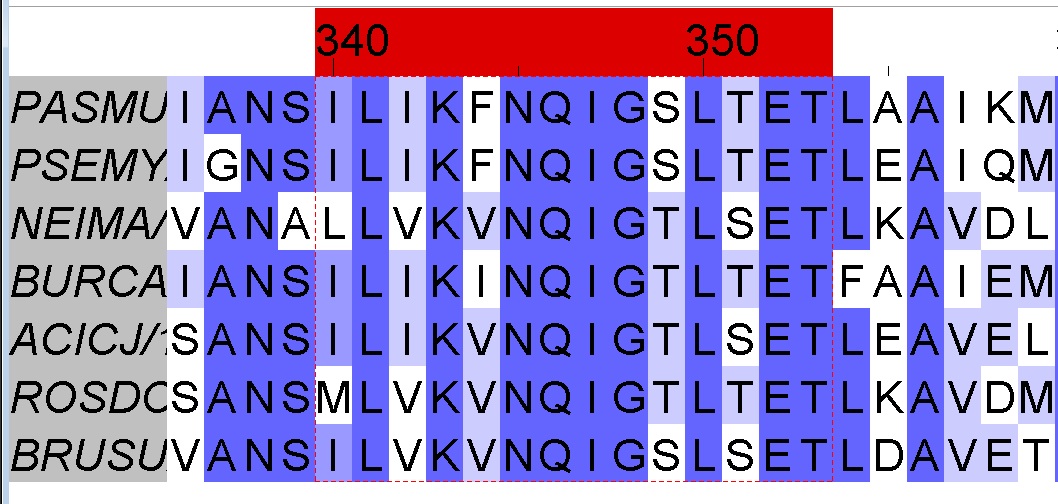
Рисунок 1. Выравнивание семи протеобактерий (мнемоники видов слева на сером фоне), открытое в JalView. Раскраска "по % идентичности", красным выделена область, в которой находятся сигналы.
Уточнение паттерна.
На рисунке 1 видно, что часть колонок достаточно консервативна. Интересно, что позиция, обозначенная в паттерне буквой "х", менее консервативна, чем остальные: в семи последовательностях есть три варианта аминокислоты на этой позиции: F, I, V. Мне также кажется осмысленным добавить к паттерну одну колонку справа и две слева, т.к. эти позиции не менее консервативны, чем внутренние.
Отредактированный (более строгий) паттерн имеет вид:
[ILM]-L-[IV]-K-[VFI]-N-Q-I-G-[TS]-L-T-E-T
Или, с тремя прибавленными колонками:
N-[SA]-[ILM]-L-[IV]-K-[VFI]-N-Q-I-G-[TS]-L-T-E-T-[LF]
Поиск соответствий уточнённому паттерну в банке Swiss-Prot.
С помощью ScanProsite (Option 2 - Submit MOTIFS to scan them against a PROTEIN sequence database) были найдены 450 белков из Swiss-Prot, содержащие мотивы, подходящие под данный паттерн. Список идентификаторов этих белков был сохранён в файл.
Теоретически этот список должен быть близок к "правильному списку" - всем енолазам протеобактерий. Такой "правильный" список был получен поиском в Uniprot командой:
mnemonic:eno_* taxonomy:"Proteobacteria [1224]"
Результаты такого поиска были сохранены в файл. (только список находок) - всего 396 находок. С помощью скрипта были найдены следующие характеристики:
Из чисел видно, что многие "правильные" белки были не найдены по паттерну, а многие найденные оказались "неправильными". Это можно объяснить несколькими способами. Во-первых, паттерн уточнялся по семи последовательностям, которые пусть и относятся к разным группам внутри таксона, но всё-таки не передают всего разнообразия реальных сигналов, имеющихся у протеобактерий. Во-вторых, возможно, я зря добавила три колонки, показавшиеся мне консервативными, к паттерну. Возможно, что в сигнал они не входят (т.е. не участвуют во взаимодействии при узнавании), а у этих семи случайно оказались чуть консервативнее, чем последовательность вокруг. В-третьих, я не знаю, что это за паттерн: в базе он называется "енолазный паттерн", но, возможно, не у всех енолаз он должен быть, или он есть у некоторых представителей не_енолаз, связывающихся с тем же белком, или что-нибудь ещё в том же роде - чтобы разобраться, нужно искать, какой белок узнаёт сигнал, что этот белок делает, что общего у белков из списка FP и т.д.
Вернуться на страницу семестра
© potapenko 2017-2019