
Примечание: я выполняла задания "Варианта 2". Ссылка на страницу с условиями заданий.
1. Выбор семейства доменов.
На сайте Pfam можно увидеть список всех имеющихся в базе семейств доменов:
Для выполнения задания было выбрано семейство доменов c ID = ADAM-CR (расшифровывается как A Disintegrin And Metalloproteinase cysteine-rich), AC = PF08516. ADAM - заякоренные в мембране белки, которые протеолитически изменяет поверхность клетки и внеклеточного матрикса, тем самым влияя на поведение клетки, ADAM-CR - один из доменов этих белков, регулирующий их активность.
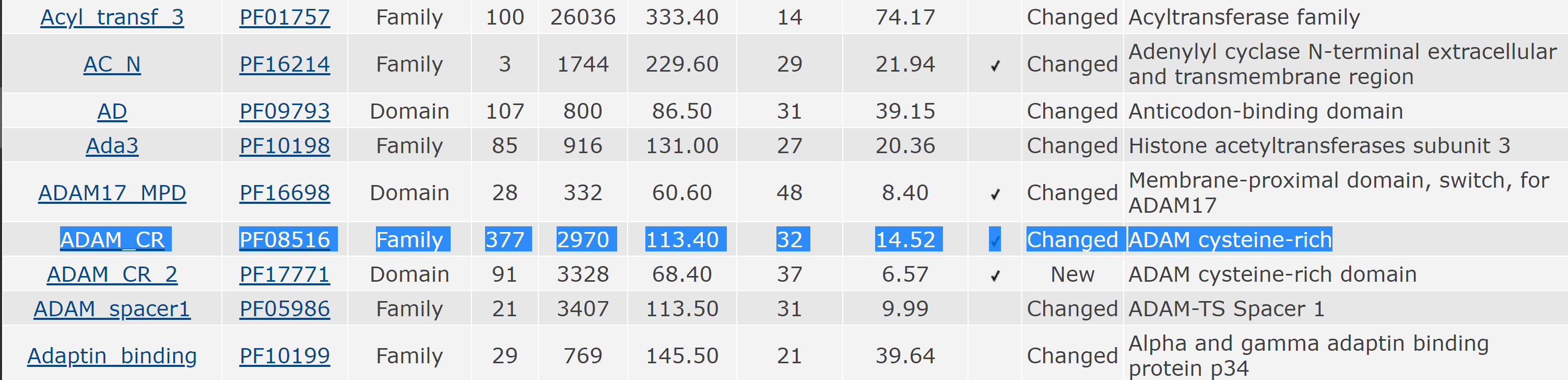
На сентябрь 2019 к этому семейству доменов относится 2970 последовательностей средней длиной 113 аминокислот, галочка стоит в колонке "наличие 3D структуры" (наличие структуры PDB в базе, так-то каждый белок имеет 3D структуру). На странице самого семейства указано, что к нему относятся 83 архитектуры, 27 структур, а уже упомянуты е 2970 последовательностей относятся к 311 видам, и все эти виды эукариотические.
Выравнивание всех последовательностей, открытое в JalView, выглядит характерно: довольно много очень консервативных столбцов, а между ними длинные индели за счёт того, что у какого-то белка из трёх тысяч в этом месте вставка. За счёт этого выравнивание получается в 4 раза длиннее заявленных средних 113 аминокислот.
Выравнивание было раскрашено по консервативности, к нему было добавлена структура, соответствующая последовательности ADA22_HUMAN с pdb_id = 5Н2Z.
2. Выбор архитектур.
Первая из двух выбранных архитектур (изображение на левом рисунке ниже) - Disintegrin, ADAM_CR. Такой архитектурой обладают 74 последовательности. Как следует из названия, в ней содержатся 2 домена: первый - это ADAM_CR, второй - Disintegrin (это семейство коротких белков - ингибиторов интергин-зависимой клеточной адгезии, входит в состав некоторых ядов гадюк).
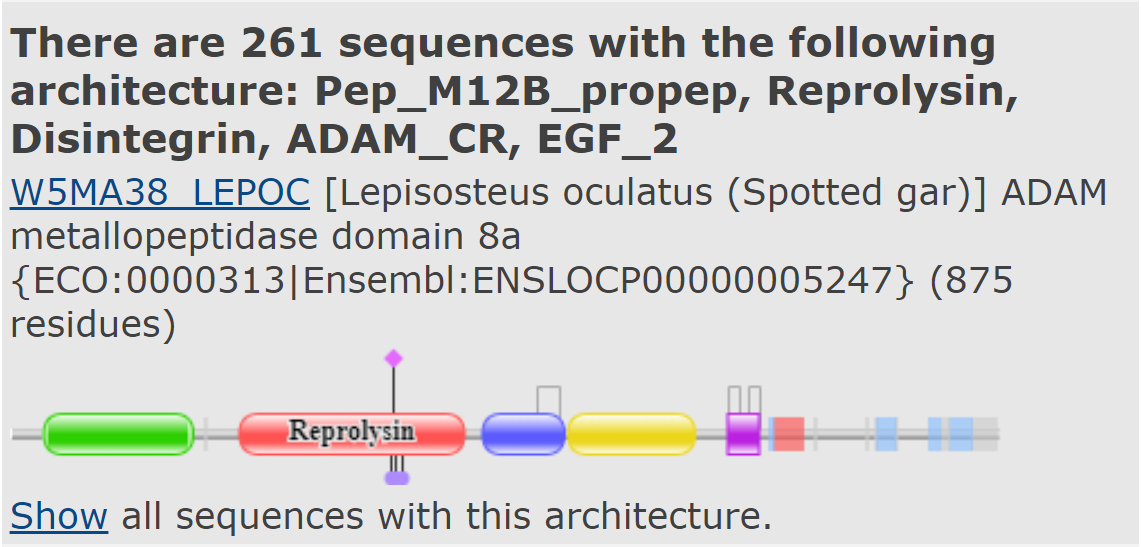
Вторая выбранная архитектура (правый рисунок ниже) - уже пятидоменная, называется Pep_M12B_propep, Reprolysin, Disintegrin, ADAM_CR, EGF_2. В неё входят те же два домена и три новых. Такой архитектуре соответствуют 264 последовательности. Reprolysin - семейство цинк-зависимых металлопептидаз, входит в состав некоторых ядов гадюк, но встречается и у млекопитающих, в том числе у человека это белок, предположительно вовлечённый в выход лейкоцитов в ткань из капилляров.
"Pep_M12B_propep" расшифровывается как Reprolysin family propeptide, т.е. содержит пропептиды (предшественники) доменов из предыдущего семейства репролизинов. EGF-like domain очень консервативный, встречается в очень разных белках, объединяет их то, что они заякорены в клеточной мембране, а этот домен находится с внешней стороны.
 |
 |
| Рис.2. Первая доменная архитектура. | Рис.3. Вторая доменная архитектура. |
С помощью скрипта (все скрипты даны на странице и никак не модифицировались) была получена таблица с последовательностями домена PF08516, чтобы далее из таблицы можно было выбрать строки, соответствующие двум выбранным архитектурам. Были выбраны следующие параметры: -z означает, что входные данные подаются в сжатом виде (файл swisspfam.gz), параметр -р указывает, последовательности какого домена нужны, параметр -о задает имя таблицы с результатами. Запущенная команда:
python swisspfam-to-x/swisspfam.gz -p PF08516 -o PF08516.xlsx
Ссылка на полученную таблицу.
По полученной таблице была построена сводная таблица (строки – AC последовательностей, столбцы – домены Pfam). После чего по сводной таблице были выбраны последовательности, соответствующие выбранным архитектурам и записаны их АС Фильтрация проводилось инструментами LibreOffice.
3. Выбор таксона и подтаксонов.
Для полуучение информации о таксономической принадлежности последовательностей сначала были скачаны полные записи всех последовательностей. Для этого список выбранных ранее АС бы загружен в Uniprot - Retrieve и сохренен в текстовый файл, содержащий полные записи нужных последовательностей (в формате Uniprot).
Далее был использован скрипт, добавляющий информацию про такнсномию из этого файла в экселевскую таблицу. Запущен строкой:
python uniprot_to_taxonomy.py -i uniprot_retrieve.txt -o taxon.txt
Полученные данные (полученная таблица) были добавлены к основной таблице с помощью функиции VLOOKUP (из пакета LibreOffice).
В качестве подтаксона таксонов Metazoa были выбраны типов Ecdysozoa и Chordata, от каждого домена было взято по примерно 15 белков внтури каждого из двух подтаксонов.
Итоговый файл-таблицу можно посмотртеть тут.
Полученные последовательности были переименованы , X - первая архитектура (двухдоменная), Y - вторая (пятидоменная); после буквы через черту идёт скоращение от таксона - Ecdys или Chord. и загружены в Jalview. В полученном выравнивании были удалены пустые столбцы.
4. Построение филогенетического дерева домена.
Дерево строилось по итоговому выравниванию (ссылка на проект проект (.jvp)). Для построения дерева в программе MEGA-X выравнивание было экспортировано в формат fasta. Дерево было построено алгоритмом Maximum Likelihood с бутстреп-поддержкой в 1000 реплик.
Скобочную формулу дерева (в MEGA export > newick) можно посмотреть здесь, на ветвях оставлены цифры - бутстреп-поддержка, т.к. часть ветвей получилась ненадёжной.
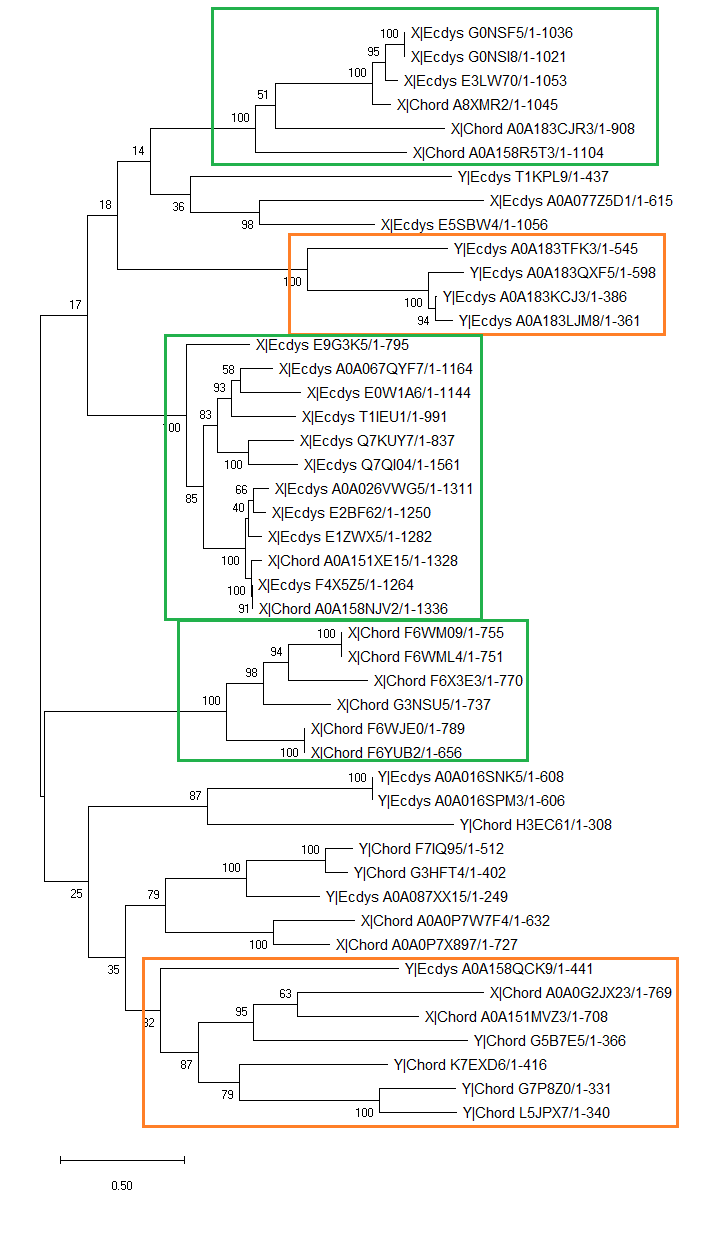
Рисунок 4. Филогенетическое дерево, построенное в программе MEGA.
Интерпретация дерева. По дереву, построенному по одному домену, присутствующему в нескольких (в данном случае двух) доменных архитектурах, можно сделать вывод о том, какое разделение произошло раньше: разделение на таксоны (линяющие и хордовые) или разделение на доменных архитектуры. На данном дереве видны несколько крупных клад, в которых встречается почти только одинаковые доменные архитектуры (клады, содеражащие в основном домены из архитектуры "Х", заключены в зелёные прямоугольники, клады, содержащие в основном домены из архитектуры "Y" - в оранжевые). Ветви, где это не так (т.е. рядом расположены домены из разных архитектур), часто имеют низкую бутстреп-поддержку.
При этом часто встречаются на близких ветвях встречаются домены из разных таксонов. Из-за низкой поддержки некоторых даже крупных клад нельзя определить точно, но я бы предположила, что сначала произошло деление на доменные архитектуры (у общего предка двух подтаксонов), а потом - разделение на линяющих и хордовых. Хотя этому выводу противоречит то, что большие клады со второй доменной архитектурой (два оранжевых прямоугольника) встречаются в разных "концах" дерева, как будто разделение на домены происходило дважды...
Вернуться на страницу семестра
© potapenko 2017-2019