В этом задании нужно было построить дерево по белкам семи бактерий (выбранных в практикуме 1), гомологичных заданному белку с мнемоникой CLPX из бактерии E.coli (которая, кстати, относится к гаммапротеобактериям, то есть не является аутгруппой для построенного дерева, а входит в одну из ветвей).
Полные протеомы семи бактерий были скопированы из папки /P/y17/term4/Proteomes и объединены в один файл командой:
cat *.fasta >> 7proteoms.fasta
Из этого общего файла была создана база данных для локального blast командой:
makeblastdb -in 7proteoms.fasta -dbtype prot
Из файла с полным протеомом E. coli была вырезана последовательность белка CLPX и сохранена в отдельный файл CLPX_ECOLI.fasta. Лучшие находки blast были выписаны в отдельный файл:
blastp -query ECOLI/CLPX_ECOLI.fasta -db 7proteoms.fasta -out homologs.fasta -evalue 0.001
Из файла с результатами были взяты мнемоники находок, записаны через точку с запятой, и в JalView через опцию "fetch sequences" последовательности были загружены, а потом выровнены с помощью Muscle. (Хотя файл с результатами содержит последовательности находок и можно было вырезать их оттуда, а не скачивать ещё раз).
Выравнивание "выглядело" не очень хорошо, поэтому я попробовала выровнять последовательности другим алгоритмом (MAFFT), сильно лучше не стало, поэтому дерево, реконструированное алгоритмом Maximum Likelihood, было сделано с бутсреп-поддержкой в 100 репликах, чтобы знать, каким ветвям "можно доверять". Я специально не стала укорачивать названия последовательностей перед тем, как строить дерево, так как часть последовательностей имеет плохо читаемые мнемоники, имеющие разный вид у белков с одинаковыми названиями.
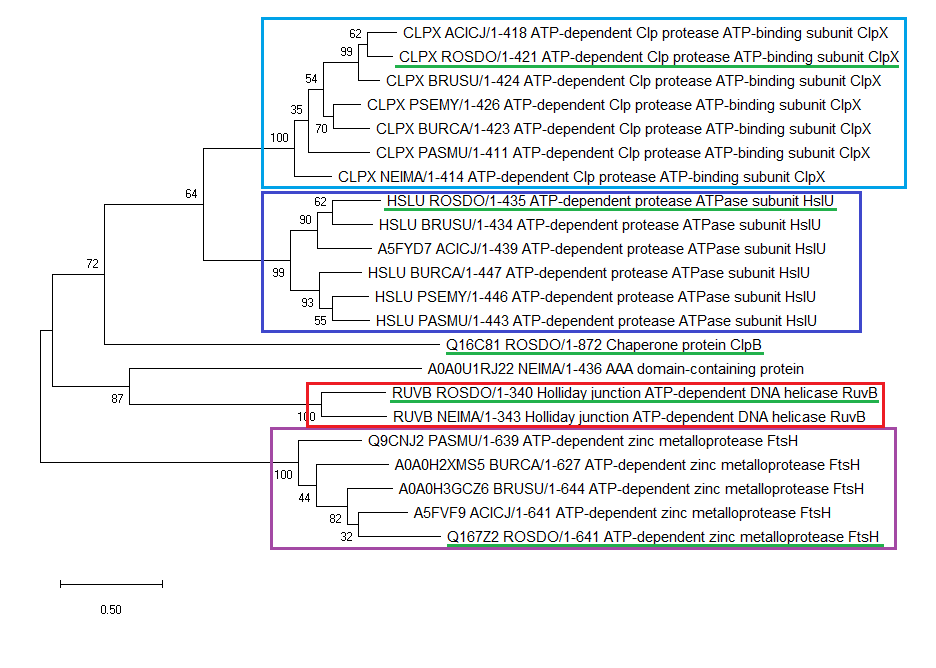
Рисунок 1. Дерево гомологов CLPX_ECOLI среди выбранных семи бактерий.
В прямоугольники голубого, синего, фиолетового и красного цветов заключены группы белков, которые внутри группы являются ортологами (т.к. имеют одинаковую мнемонику функции, но относятся к разным видам). Узлы дерева, ближайшие в прямоугольникам, соответствуют дупликациям. Так, хороший пример дупликации - разделение на ClpX и HslU. Эта дупликация, видимо, произошла довольно рано, так как у шести бактерий из семи есть оба белка. Белка HslU нет только у вида с мнемоникой NEIMA. Т.к. по "правильному" (данному в условии) дереву этот вид принадлежит внутренней ветви, можно предположить, что белок HslU был у этого вида (иначе его не было бы у сестринского вида с мнемоникой BURCA).
Пары белков из разных прямоугольников, пренадлежащие одному виду, являются паралогами (т.к. имеют разные мнемоники функций и принадлежат одному виду, т.е. разошлись в результате дупликации). Узлы дерева внутри прямоугольников соответствуют видообразованию. Например, зёленым подчеркнуты паралоги, принадлежащие виду с мнемоникой ROSDO. Один из этих паралогов (шаперон ClpB) не имеет ортологов в других организмах на этом дереве, что может быть связано либо с потерей этого белка у других видов, либо тому, что ортологичные белки других видов были чуть меньше похожи на CLPX_ECOLI и не попали в выдачу blast (эта версия кажется правдоподобной, т.к. находка этого белка имеет E-value = 7e-04).
Вернуться на страницу семестра
© potapenko 2017-2018