Для выполенния заданий был выбран белок с мнемоникой ENO_BACSU (енолаза (фермент одной из стадий гликолиза) из бактерии Bacillus subtilis). Аминокислотная последовательность был найдена в базе Uniprot.
Для начала в окно сервиса CDD Conserved Domain Database была введена аминокислотная fasta-последовательность белка. Затем в окне результатов в поле (верхний правый угол) View -> Full Results.
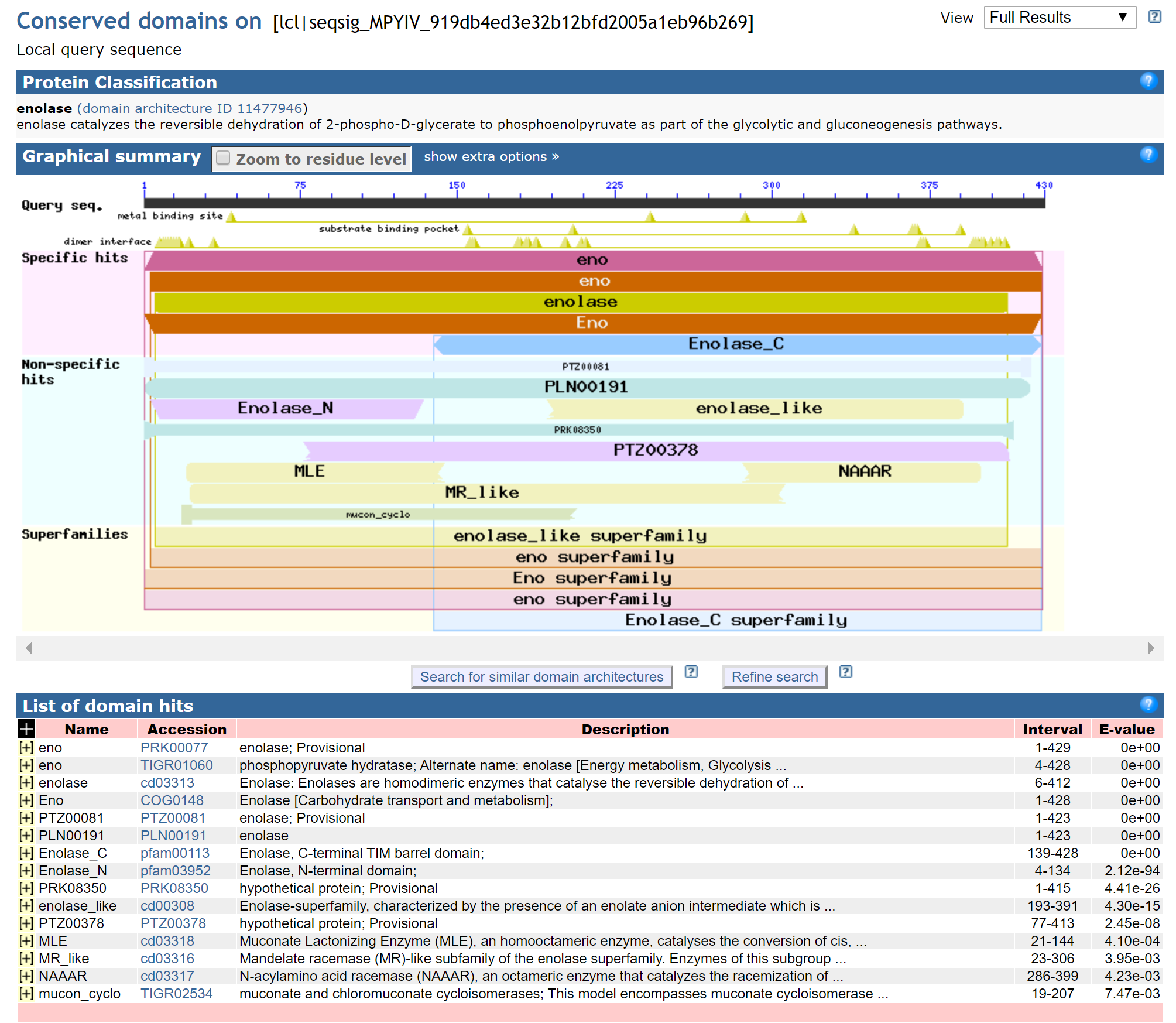
Рисунок 1. Результаты поиска в СDD.
На странице появился список хитов, среди которых - искомый КОГ. Информация о КОГе:
В COGNAT был запущен поиск геномного окружения данного КОГа с параметрами:

Рисунок 2. Геномное окружение при пороге встречаемости 20%. Енолаза обозначена бордовой стрелкой, COG0504 - коричневатой, COG2919 - жёлто-зелёная. Показано геномное окружение для части протеобактерий, у которых данные КОГи часто встречаются в геномном окружении КОГа 0148.
Как уже было сказано, для порогового значения встречаемости в 20% находятся всего два контига из ближайшего окружения. Белки этих КОГов не связаны прямо функционально с COG0148: они относятся к другим функциональным категориям. Один из них - COG0504, в основном представленный CTF-синтазой, относится к функциональной категории F (транспорт и метаболизм нуклеотдов). Расположен у большинства организмов (из тех, у кого она вообще рядом) раньше гена енолазы на один или два гена, всегда расположен на той же цепи, что и енолаза.
Другой часто встречающийся в окружении COG2919 представлен очень короткими белками, относится к функциональной категории D (контроль клеточного цикла и клеточное деление). Представлен белком клеточного деления FtsB и белком Septum formation initiator, белками с похожими названиями, в т.ч. предсказанными. Этот КОГ расположен после КОГа, к которому принадлежит енолаза, и всегда на той же цепи. Может встречаться как сразу после КОГа енолазы, так и через два-три гена после неё.

Рисунок 3. Геномное окружение при пороге встречаемости 10%. Енолаза обозначена бордовой стрелкой, COG0504 - коричневатой, COG2919 - жёлто-зелёная, COG2877 - синевато-зелёная, COG0149 - чёрная. Показано окружение для группы протеобактерий, у которых оно достаточно консервативно.
Если опустить пороговое значение встречаемости до 10%, будут показаны ещё два КОГа. Это COG2877 (синтазы кислоты KDO, присутствующей в клеточной стенке?, функциональная категория М) и COG0149 (изомераза триозофосфата), который имеет соседний номер с COG0148 (к которому относится енолаза), и принадлежит той же функциональной категории G.
В целом можно сказать, что геномное окружение у КОГа 0148 не консервативное: даже те КОГи, которые встречаются у хотя бы 10% организмов, могут быть расположены через разное число генов от гена КОГа 0148, часто располагаются по разные стороны от него. Однако, как видно на рисунке 3, у отдельных групп организмов окружение может быть достаточно консервативно.
Поиск наиболее похожего белка должен был производиться с помощью инструмента AmiGO поиском blast.
Однако, когда я зашла на соответсвующий сайт, я не обнаружила там blast (возможно, он теперь расположен где-то в другой части сайта и я его просто не нашла). Как я понимаю, далеко не все белки есть в базе данных GO. Поэтому blast нужен для того, чтобы найти белки, похожие на "мой" белок, и посмотреть термины GO для них, а потом сказать, что у очень похожих (по последовательности) белков термины GO должны быть одинаковые.
Поэтому я решила попробовать поискать похожие белки "обычным" blast на сайте NCBI. К сожалению, там нет возможности ограничить поиск теми белками, которые есть в GO, и я собиралась ограничить поиск теми белками, которые есть в Uniprot. Таким образом нашлись енолазы из каких-то родственных бактерий, и я перешла на их страницы в Uniprot, чтобы взять их идентификатор из Uniprot и по нему искать в БД GO.
В результате мне это не понадобилось, т.к. в Uniprot на странице ENO_BACSU была ссылка "Complete GO annotation on QuickGO" , ведущая на сайт EMBL, откуда и взята дальнейшая информация.
Таблица 1. Отнесение белка к терминам GO.
| тип отношения | Идентификатор GO | Название термина | Перевод названия | Код типа достоверности |
| part_of | GO:0000015 | phosphopyruvate hydratase complex | фосфопируватгидрогеназный комплекс | IEA |
| enables | GO:0000287 | magnesium ion binding | взаимодействие с ионом магния | IEA |
| involved_in | GO:0006096 | glycolytic process | гликолиз | IEA |
| enables | GO:0005515 | protein binding | взаимодействие с белком | IPI |
Всего для данного белка была 21 аннотация к терминам GO, но только одна из них имела код типа достоверности какой-то кроме IEA, то есть реально подтвержденных данных очень мало.
Таблица 2. Описание кодов достоверности, использованных в Таблице 1.
| Код типа достоверности | Расшифровка кода | Объяснение |
| IEA | Inferred from Electronic Annotation | Предположила на основе биоинформатической аннотации, экспериментами не подтверждено. |
| IPI | Inferred from Physical Interaction | Предположили на основе физического взаимодействия. |
Вернуться на страницу семестра
© potapenko 2017-2018