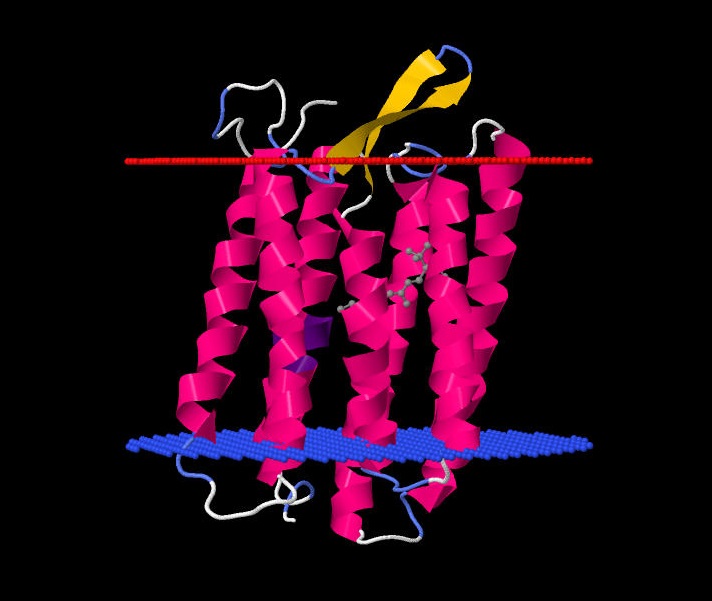
Для выполенния заданий был выбран белок из базы Orientation of Proteins in the Membrane OPM > Transmembrane > Alpha-helical polytopic > Rhodopsin-like receptors and pumps > Microbial and algal rhodopsins > Archaerhodopsin-1 (1uaz). Этот белок является протонной помпой, активирующейся при воздействии светом. Если открыть его структуру в JMol, видно, как между альфа-спиралей скоординирован ретиналь.
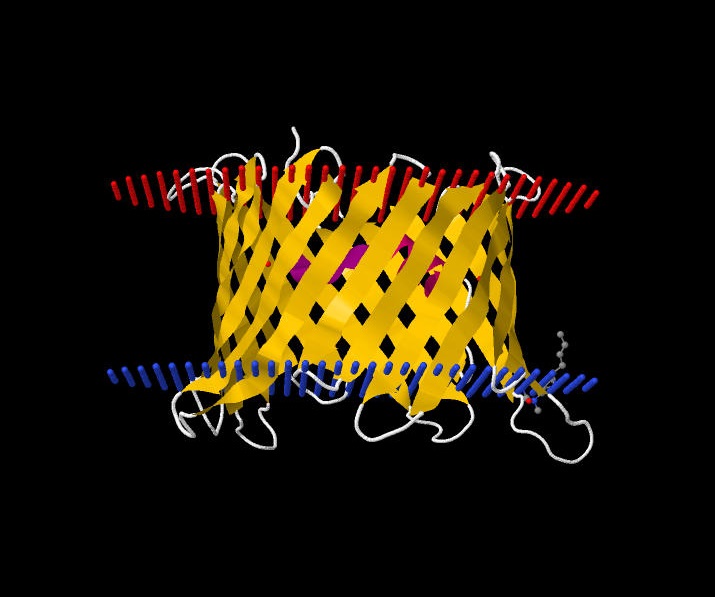
Аналогчно (спускаясь вниз по группам белков) был найден второй белок, он в трансмембранной части содержит бета-слой: Transmembrane > Beta-barrel transmembrane > Mitochondrial and plastid porins > Voltage-dependent anion channel (VDAC) porin > VDAC-2 channel. Это белок с названием VDAC-2 channel и идентификатором 4bum. Он является порой между митохондрией и цитозолем, через которую могут проходить маленькие гидрофильные молекулы.
Таблица 1. Основные характеристики выбранных белков. Комментарии см. ниже.
| PDB_ID | 1UAZ | 4BUM |
| название | Archaerhodopsin-1 | VDAC-2 channel |
| толщина гидрофобной части | 31.8 ± 1.3 A | 23.4 ± 0.9 |
| трансмембранные вторичные структуры | 7 альфа-спиралей | 19 элементов вторичной структуры образуют бета-бочонок + одна альфа-спираль внутри (малиновая на рисунке) |
| координаты трансмембранных участков | 1(15-38),2(48-68),3(86-104),4(111-133),5(138-160),6(179-197),7(207-230) | 1( 14- 21), 2( 28- 33), 3( 40- 46), 4( 55- 62), 5( 70- 74), 6( 81- 87), 7( 95- 102), 8( 111- 119), 9( 123- 130),10( 137- 145), 11( 149- 156),12( 166- 173),13( 180- 184),14( 191- 197),15( 204- 209),16( 218- 226),17( 243- 249),18( 257- 263), 19( 274- 281) |
| среднее число остатков в спирали \ тяже | 18 | 6 |
| локализация | клеточная мембрана Halobacterium sp. | внешняя мембрана митохондрий Danio rerio |
| изображение белка (p-сторона вверх) |  |

|
Данные в таблице, говорящие о толщине гидрофобной части, были взяты со страниц этих белков в базе OPM. Кроме того, я открыла pdb-структуры этих белокв в программе JMol и сама измерила толщину трансмембранной части белков, значения получились похожие, но измерения были заметно менее точными: сложно расположить "линейку" строго перпендикулярно поверхности мембраны и результат немного зависит от того, от какого до какого аминокислотных остатков измерять. Однозначно можно утверждать, что толщина гидрофобной части у данного белка с альфа-спиралями почти в полтора раза больше, чем толщина гидрофобной части данного белка с бета-бочонком.
Файл с pdb-структурой лучше скачивать из БД OPM, тогда открывается сразу раскрашенная по вторичной структуре модель, атомами почему-то азота и кислорода показано положение двух мембран. Сторона мембраны, обращенная в цитозоль, обозначена атомами азота (синие), обращенная наружу или в межмембранное пространство, обозначена красными атомами кислорода.
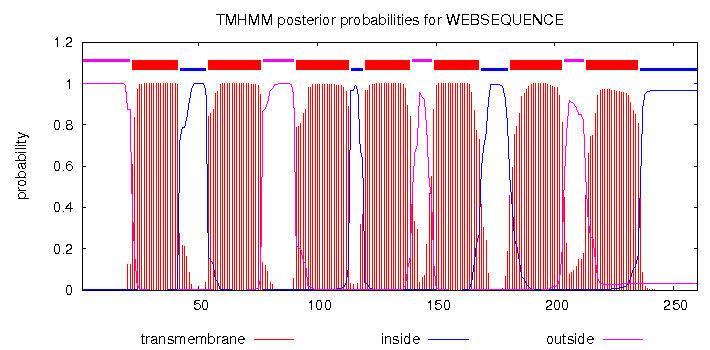
Программы, предсказывающие трансмембранные спирали ( TMHMM и Phobius ), принимают на вход белковую последовательность, поэтому последовательность белка Archaerhodopsin-1 была скачана из Uniprot в fasta-формате.
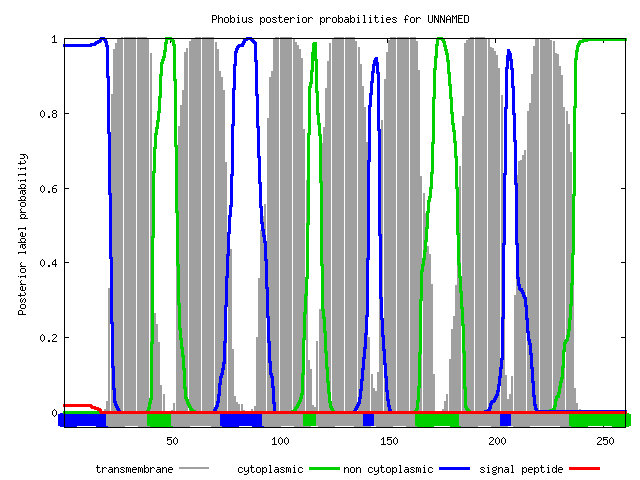
Как видно из графической выдачи программ (рисунки 3 и 4), обе программы программы верно предсказали количество и примерные координаты трансмембранных участков: на обоих графиках по оси х указаны координаты (в аминокислотных остатках), по оси у - степень уверенности в том, что этот остаток относится к одной из частей белка (разными цветами показаны уверенности для внутренних участков, внешних участков и трансмембранных участков).
 |
|
| Рис.3. Графическая выдача программы TMHMM. | Рис.4. Графическая выдача программы Phobius. |
Более подробную информацию можно увидеть в текстовой выдаче. Я свела в одну таблицу результаты работы двух программ (только координаты трансмембранных участков), а в последней колонке указала координаты трансмембранных участков, указанные в OPM. Видно, что, во-первых, результаты предсказаний двумя программами расходятся очень слабо (максимум - на пять остатков, только в одном предсказании), но отличаются от информации, указанной в ОРМ. При этом координаты из OPM стабильно отличаются на 5-7 остатков, поэтому я предполагаю, что такой систематический сдвиг может быть результатом ошибки (разной нумерации аминокислотных остатков, ведь файл с последовательностью взят из другой базы данных). Хотя, возможно, это особенность этих алгоритмов.
tmhmm 22 41 phobius 23 41 OPM 15-38 tmhmm 54 76 phobius 53 75 OPM 48-68 tmhmm 91 113 phobius 95 113 OPM 86-104 tmhmm 120 139 phobius 120 141 OPM 111-133 tmhmm 149 168 phobius 147 165 OPM 138-160 tmhmm 181 203 phobius 186 204 OPM 179-197 tmhmm 213 235 phobius 210 236 OPM 207-230
Я решила проверить предположение про разную нумерацию, предполагая, что нумерация, используящаяся на сайте ОРМ, такая же, как в pdb-файле, представленном на этом сайте, и в JMol посмотрела в этом файле, какими являются первые шесть и последние шесть остатков в цепи, а потом нашла эти участки в fasta-файле, с которым работали программы предсказания. Вот что получилось:
>sp|P69052|BACR1_HALSS Archaerhodopsin-1 OS=Halobacterium sp. (strain SG1) OX=33006 GN=bop PE=1 SV=1 MDPIALTAAVGADLLGDGRPETLWLGIGTLLMLIGTFYFIVKGWGVTDKEAREYYSITIL VPGIASAAYLSMFFGIGLTEVQVGSEMLDIYYARYADWLFTTPLLLLDLALLAKVDRVSI GTLVGVDALMIVTGLVGALSHTPLARYTWWLFSTICMIVVLYFLATSLRAAAKERGPEVA STFNTLTALVLVLWTAYPILWIIGTEGAGVVGLGIETLLFMVLDVTAKVGFGFILLRSRA ILGDTEAPEPSAGAEASAAD
Жирыным шрифтом обозначены остатки, которые есть в структуре в pdb-файле, курсивом - те остатки по краям последовательности, которых в структуре из ОРМ нет. И действительно, первых шести остатков в структуре нет, поэтому нумерация должна быть сдвинута в какую-нибудь сторону на 6 позиций. Вот какие расстояния получаются, если везде нумеровать так, как остатки идут в fasta-файле:
tmhmm 22 41 phobius 23 41 OPM 21-44 tmhmm 54 76 phobius 53 75 OPM 54-74 tmhmm 91 113 phobius 95 113 OPM 92-110 tmhmm 120 139 phobius 120 141 OPM 117-140 tmhmm 149 168 phobius 147 165 OPM 144-166 tmhmm 181 203 phobius 186 204 OPM 186-203 tmhmm 213 235 phobius 210 236 OPM 213-236
Видно, что тогда в среднем отличия предсказанной структуры от реальной меньше (хотя у TMHMM всё еще есть отличие в координатах в пять аминокислотных остатков), в среднем около двух остатков для обеих программ.
База данных TCDB (Transporter classification DataBase) содержит далеко не все белки даже среди транспортёров, так что поиск по pdb_id и по AC (взятому, опять же, из Uniprot), не дал результатов, хотя вообще по идентификаторам искать можно. Потом я попробовала искать по названию "VDAC-2" и нашла два белка, один человеческий, второй дрожжевой. Видимо, этого белка из Halobacterium в базе нет.
Для белков в этой БД указан код ТС (Transport Classification):
На самом деле, в БД намного больше белков VDAC-2, их можно найти, если искать по полному названию, или названию без дефиса, и т.д. Или же можно посмотреть все белки из данного семейства, их всего 10 штук в этой базе.
Про первый белок (Archaerhodopsin-1) в ОРМ указано, что он относится к семейству 3.E.1 The Ion-translocating Microbial Rhodopsin (MR) Family. Однако, в списке белков этого семейства его нет, есть только Archaerhodopsin-2 и Archaerhodopsin-3, имеющие коды:
Я также воспользовалась blast на сайте ОРМ (со стандартными параметрами); оказалось, что первые две находки и есть эти архейные родопсины 2 и 3. Четвертая находка - первый, но бактериальный родопсин. Все они относятся к семейству 3.Е.1.1.
Вернуться на страницу семестра
© potapenko 2017-2018