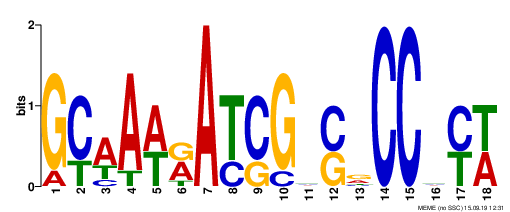


Т.к. сайт бактериальной базы мотивов RegPrecise не работал, я выполняла задания прошлого года по той же теме. Ссылка на страницу с условиями заданий.
1. Отбор данных.
Для работы была взята бактерия Xanthomonas campestris. Среди бактерий этого вида нужно было выбрать штамм с как минимум восемью проверенными аннотированными записями с ключевым словом Purine biosynthesis [KW-0658]. Поиск проводился в Uniprot по запросу:
keyword:"Purine biosynthesis [KW-0658]" organism:"xanthomonas campestris"
Белки со статусом "Reviewed" были у четырёх штаммов, только у одного из них было больше 8 (9) белков. Uniprot-vнемоника этого штамма - XANCP, полное название - Xanthomonas campestris pv. campestris (strain ATCC 33913 / DSM 3586 / NCPPB 528 / LMG 568 / P 25). Все найденные белки приведены в таблице 1. Все они соответствуют запросу:
keyword:"Purine biosynthesis [KW-0658]" organism:"xanthomonas campestris" AND reviewed:yes AND organism:"Xanthomonas campestris pv. campestris (strain ATCC 33913 / DSM 3586 / NCPPB 528 / LMG 568 / P 25) [190485]"
Таблица 1. Отобранные белки.
| Идентификатор | Мнемоника | Название белка | Название гена и локуса |
| Q8PBI2 | PURT_XANCP | Formate-dependent phosphoribosylglycinamide formyltransferase | purT XCC1137 |
| Q8PBR6 | PURA_XANCP | Adenylosuccinate synthetase | purA XCC1053 |
| Q8PCQ7 | PUR4_XANCP | Phosphoribosylformylglycinamidine synthase | purL XCC0656 |
| Q8P8Q4 | FOLD_XANCP | Bifunctional protein FolD | folD XCC2185 |
| Q8P8Q6 | GUAA_XANCP | GMP synthase | guaA XCC2183 |
| Q8PD48 | PUR2_XANCP | Phosphoribosylamine--glycine ligase | purD XCC0497 |
| Q8PD47 | PUR9_XANCP | Bifunctional purine biosynthesis protein PurH | purH XCC0498 |
| Q8P725 | PUR5_XANCP | Phosphoribosylformylglycinamidine cyclo-ligase | purM XCC2789 |
| Q8PD87 | PUR7_XANCP | Phosphoribosylaminoimidazole-succinocarboxamide synthase | purC XCC0453 |
Был найден полный геном бактерии в EMBL-EBI (AC записи - AE008922.1) и скачан в формате txt. В нём по названию нужных локусов были найдены координаты нужных девяти генов, по ним были посчитаны координаты upstream-регионов длиной 100 нуклеотидов (участков с с 5'-стороны). Для области перед геном, лежащим на прямой цепи - это 100 нуклеотидов перед его первой координатой, а для области перед геном с обратной цепи - это 100 нуклеотидов после его бОльшей координаты. Получившиеся координаты представлены в таблице 2.
Таблица 2. Координаты upstream-областей.
| Название локуса | Координаты гена в геноме | Координаты upstream-области |
| purT | 1323492..1324694 | 1323392..1323491 |
| purA | 1212078..1213370 | 1211978..1212077 |
| purL | 784352..788398 | 784252..784351 |
| folD | complement(2577356..2578240) | complement(2578241..2578340) |
| guaA | complement(2759384..2760139) | complement(2760140..2760239) |
| purD | complement(597809..599104) | complement(599105..599204) |
| purH | complement(599816..601399) | complement(601400..601499) |
| purM | 3298408..3299433 | 3298308..3298407 |
| purC | complement(551876..552808) | complement(552809..552908) |
Из полного генома по полученным координатам были вырезаны последовательности. Пример команды:
descseq -seq embl::AE008922.txt:1323392:1323491 -name "purT" -description " " -out purT.fasta
В случае, если ген лежит на обратной цепи, бралась обратная комплементарная последовательность, например:
descseq -seq embl::AE008922.txt:2760140:2760239:r -name "guaA" -description " " -out guaA.fasta
Потом все девять последовательностей были собраны в единый файл:
cat ????.fasta >> 9upstreams.fasta
Ссылка на итоговый файл 9upstreams.fasta
2. Поиск мотивов с помощью программы МЕМЕ
Поиск мотива производился с помощью программы MEME, установленной на kodomo. Сначала командой ememe -help были выяснены возможные параметры. Были выбраны следующие:
Общий вид команды:
ememe -dataset 9upstreams.fasta -outdir meme -nmotifs 3 -revcomp
В результате работы программы была создана новая директория meme, в которой собраны результаты. В частности, в ней есть изображения всех трёх мативов в разных форматах (в виде LOGO, т.е. высота букв отражает информационное содержание). Основная информация содержится в файле meme.html. Всю ту же (?) информацию, в том числе и составленные PWM, можно посмотреть в файле meme.txt.
 |
 |
 |
| Рис.1. LOGO первого мотива. | Рис.2. LOGO второго мотива. | Рис.3. LOGO третьего мотива. |
Первый мотив (мотивы отсортированы в порядке увеличения их E-value) имеет длину 18 нуклеотидов, встречается в 7 последовательностях из 9, его E-value = 1.4e+001, что, вообще говоря, очень много (т.е. очень плохо и очень недостоверно), и информационное содержание 19 бит.
LOGO второго и третьего мотива более короткие, но "смотрятся" лучше (буквы выше), но это только за счёт того, что каждый из них найден только у двух последовательностей (пары последовательностей разные), и их E-value соответственно равны 2.3e+003 и 2.0e+004, информационное содержание 18 и 20 бит (слабо отличается от содержания первого мотива).
Таким образов, все три найденные мотива не вызывают доверия.
Вернуться на страницу семестра
© potapenko 2017-2019