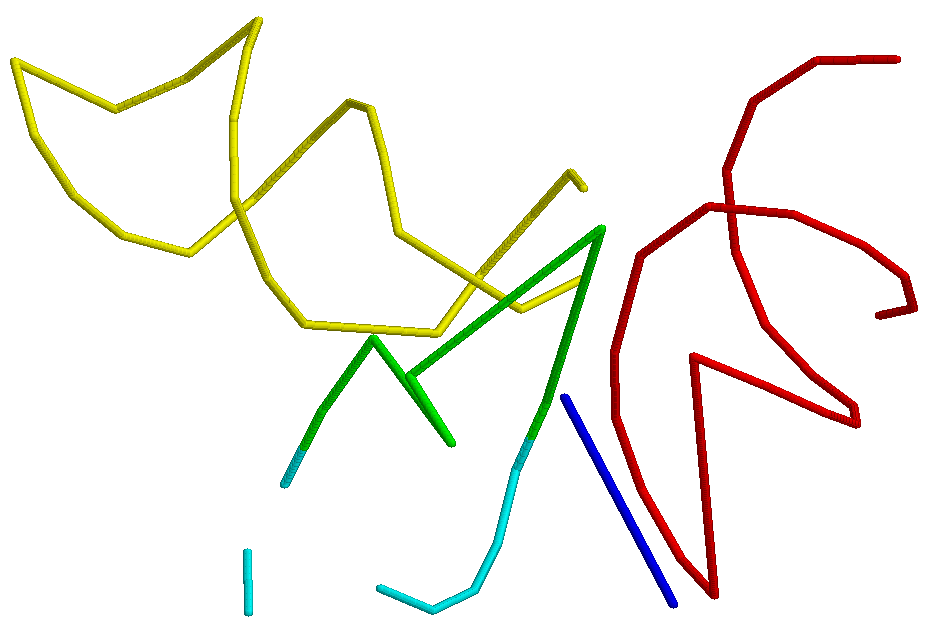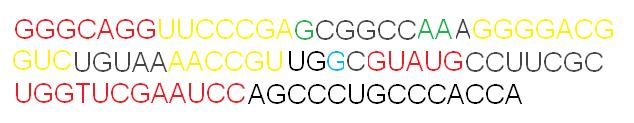На главную
Term3
Т
Р
Н
К
Задание №1. Укажите в отчете PDB-код, полученный Вами и опишите особенности структуры.цепи) и в дальнейшем работайте только с ней.
PDB-код, полученный мною - 1h3e
PDB-файл pdb 1h3е.pdb содержит информацию о структуре комплекса ДНК-белок (т.е. комплекс нуклеин-белковый).
Этот комплекс состоит, соответственно, из двух частей. - нуклеиновой и белковой.
Нуклеиновая часть - Тирозил-тРНК(нуклеиновая кислота - состоит из одной цепи В.
Белковая часть - тирозил-тРНК синтетаза (белок). Так же как и тирозил-тРНК, он состоит из одной цепи.
Эта синтетаза получена из организма THERMUS THERMOPHILUS, а вот откуда получена нуклеиновая кислота, к сожалению, не написано.
Можно сделаьт ошибочный вывод, что этот белок и нуклеиновую кислоту моно получить только из одного организма, поэтому название организма не писали второй раз просто за ненадобностью.
Однако, изучив несколько других pdb-файлов, понимаем, что это не так - нуклеиновую и белковую часть комплекса могут получать из разных организмов.
Более того, они могут даже принадлежать к разным таксонам - прокариоты и эукариоты.
Однако о данном комплексе тирозил-тРНК - тирозил-тРНК синтетаза ничего этого нельзя сказать за неимением организма из которого получили тирозил-тРНК.
Вся эта информация была взята из файла 1h3е.pdb, поля COMPND и SOURSE.
Содержание этих полей:
COMPND 1 MOL_ID: 1;
COMPND 2 MOLECULE: WILD-TYPE TRNATYR(GUA);
COMPND 3 CHAIN: B:
COMPND 4 ENGINEERED: YES;
COMPND 5 MOL_ID: 2;
COMPND 6 MOLECULE: TYROSYL-TRNA SYNTHETASE;
COMPND 7 CHAIN: A;
COMPND 8 SYNONYM: TYROSINE--TRNA LIGASE;
COMPND 9 EC: 6.1.1.1;
SOURCE 1 MOL_ID: 1;
SOURCE 2 SYNTHETIC: YES;
SOURCE 3 MOL_ID: 2;
SOURCE 4 ORGANISM_SCIENTIFIC: THERMUS THERMOPHILUS;
SOURCE 5 STRAIN: HB27;
SOURCE 6 EXPRESSION_SYSTEM: ESCHERICHIA COLI;
SOURCE 7 EXPRESSION_SYSTEM_STRAIN: BL21(DE)PLYSS;
SOURCE 8 EXPRESSION_SYSTEM_PLASMID: PET29B
На рисунке изображен этот комплекс, модель Spacefill, синим показана тРНК, голубым - белок, раскраска "chain" (изображение построено с помощью программы RasMol)

На этом рисунке изображена только РНК, без белка (модель Spacefill, синим показана тРНК):

Задание №2. Изучить, входят ли в структуру тРНК какие-либо особенности - модифицированные основания, пропуски в нумерации и/или вставки ("insertion codes"), с какого атома начинается нумерация тРНК.
GGGCAGGUUCCCGAGCGGCCAAAGGGGACGGUCUGUAAAACCGUUGGCGUAUGCCUUCGCUGGTUCGAAUCCAGCCCUGCCCACCA
Заданная тРНК состоит из 86 остатков (выше приведена нукл. последовательность). 4 остатка из 86 содержат являются модифицированными. Т.е. мутации в исследуемой тРНК составляют около 4,5 %. Среди них:
PSU Псевдоуридин-5'-монофосфат
5MU 5-метилуридин-5'монофосфат
MAD 6-гидро-1-метиладенозин-5'-монофосфат
Для изучения нумерации остатков тРНК использовали команду
'ATOM.........P.......B' 1h3e.pdb > res.txt
Т.е. остались только строки, описывающие фосфор.
Получив этот файл и изучив его, можно сделать некоторые утверждения:
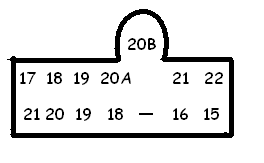
нумерация тРНК начинается с 3405 и заканчивается 4827 номером;
имеются пропуски 17;35;54;58
имеются две вставки ("insertion codes") 20A,20B и 47A-47I
Почему это так?
Первое. Нумерация атомов тРНК начинается не с первого, а аж с 3405 атома! Скорее всего, это связано с тем, что в PDB файле нумерация не начинается каждый раз по новой с
началом новой субъединицы. Она одна для белка, нуклеиновой кислоты, АТФ. тРНК - цепь В, а белок - цепь А. Естественно, что сначала пронумеровали белок, а нумерация тРНК началась
с 3405 атома.
Второе (и сразу третье) - почему имеются вставки и пропуски? Скорее всего, это объясняется тем, что в одной из цепей выпетливание. Нкулеотиды, участвующие в нем, нумеруются
как раз №а, №b, а номера атомов, которые находятся напротив выпетливания, пропадают, чтобы не нарушать последовательную нумерацию в спиральном участке. А все обычные, стандартные
пары оснований пронумерованы как обычно. Схематично это выглядит примерно так:
Задание №3, 4. Проанализируйте структуру программой find_pair. Кратко опишите полученный результат.
С помощью программы RasMol был создан файл, содержащий только нужную цепь (команда "save 1H3E1.pdb"). В этом файле все строчки HETATM (которые программа не воспринимает)
юыли заменены на АТОМ(Ctrl+H). С помощью программы find_pair полученная структура была изучена. Был проаналиирован файл
1H3E1.out (а точнее эта часть).
Возникла проблема с определением цепей - мы должны были получить 4 цепи (т.к. у нас тРНК), а получали больше.
Эта проблема была решена с помощью скрипта col_helices.txt, данного нам в выдаче после запуска
команды find_pair, было построено изображение спиралей данной нам тРНК:
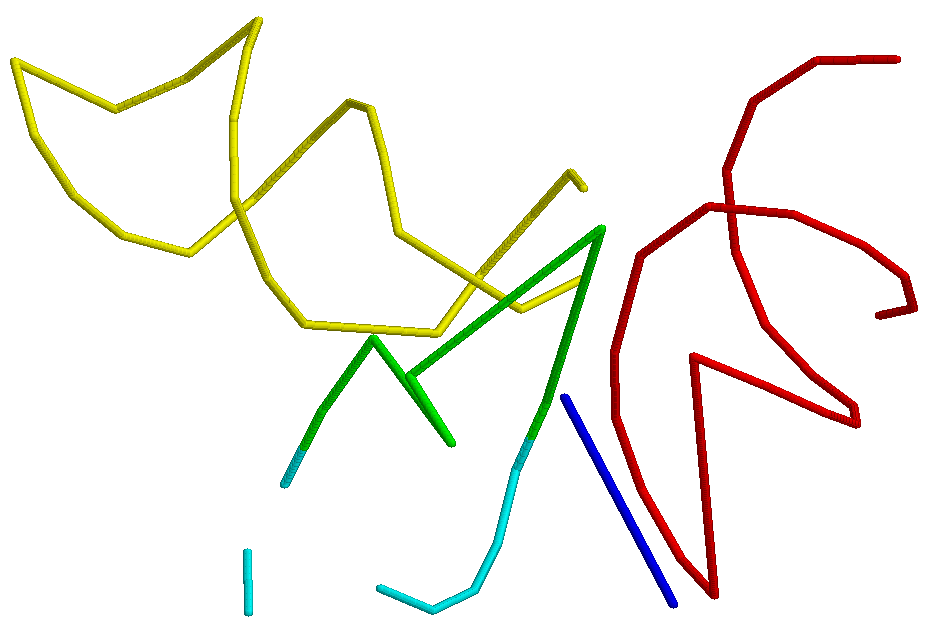
На этой картинке:
- первая спираль окрашена в желтый цвет: U8-U9; G23-U33; A36-A37; A39-U44; C10-A14.
- вторая спираль окрашена в красный цвет: G1-G7; G49-G53; C61-C72.
- третья спираль окрашена в зеленый цвет: G46-G45; A20-A21; G15; C40.
- четвертая спираль окрашена в голубой цвет: C47. find_pair нашел этот остаток почему-то не как один остатков, а как U47; U47; C47; C47; G47; U47; G47; C47. То есть как целую
последовательность из из 8ми нуклеотидных оснований, из которых некоторые повторяются - С47, С47, С47 ... Все вопросы снял pdb-файл. Дело в том, что в этом файле под номерами
47, 47А, 47 В и т.д. лежат как раз 8 разых оснований. Скорее всего это связано с выпетливанием в этом месте (см. выше). То есть мы получаем не ошибку программы, а простое
объяснением.
Вообще не очень стройную картинку можно объяснить тем, что файл очень длинный и программа просто не смогла по-нормальному его прочитать.Да и на общем изображении тРНК видно,
что структура не совсем обычная... Возможно, такое построение последней, четвертой спирали вызвано и этим тоже.
Также достаточно проблематично как-то раскрасить структуру по спиралям, потому что почти все спирали на картинке получились "разорваны".
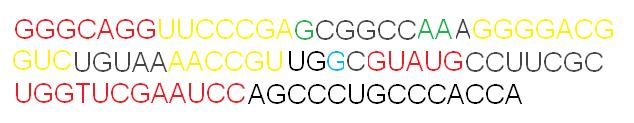
Задание №5. Пользуясь средствами RasMol, проанализируйте структуру. Приведите в отчёте пример внеспирального стекинг-взаимодействия между основаниями; пример водородных связей между основаниями, не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований.
Аргументированно объясните, на какую из форм ДНК похожи спирали РНК.
Потенциально возможные типы взаимодействий между белками и нуклеиновыми кислотами (случай комплекса 1h3e.pdb):
1. взаимодействие между фосфатами и положительно заряженными группами аминокислот ;
2. водородные связи между фосфатами, сахарами, основаниями нуклеиновых кислот и пептидными группами или гидрофильными боковыми цепями аминокислотных остатков;
3. стекинг-взаимодействия между боковыми группами ароматических аминокислот (Trp, Tyr, Phe, His) и основаниями.;
4. гидрофобные взаимодействия между основаниями и боковыми группами неполярных аминокислот.
Это основные нуклеотид-белковые взаимодействия. Рассмотрим два из них (как требется в задании).
Внеспиральные стекинг-взаимодействия
Данное изображение иллюстрирует стекинг-взаимодействия между 2-72, 18-19-57 основаниями. Энергия стекинг взаимодействий ~ 3 -15 ккал/моль.
Список команд:
background white
select *
wireframe 50
restrict rna and not backbone
select (oxygen,nitrogen) and rna and not backbone
spacefill 200
select oxygen
color red
select nitrogen
color blue
zoom 140
select * and not (18:B,19:B,57:B,2:B,72:B)
spacefill off
Водородные связи
На этой картинке изображении водородные связи между основаниями, не сводящиеся к Уотсон-Криковскому спариванию комплементарных оснований 13-9:B, 15-48:B, 20-47:B, 33-36:B, 37-32:B, 44-26:B, 47-47:B

Список команд:
background white
select *
wireframe 40
define helix1 1-7:B,49-53:B,55:B,18:B,61-72:B
select helix1
color green
define helix2 8-15:B,23-33:B,36-44:B,48:B
select helix2
color purple
define helix3 19:B,56:B
select helix3
color black
define helix4 20:B,47:B
select helix4
color blue
define helix5 46:B,47:B,47:B,47:B
select helix5
color orange
define helix6 47:B,47:B
select helix6
color cyan
restrict rna and not backbone
select (oxygen,nitrogen) and rna and not backbone
spacefill 120
select * and not (33:B,36:B,37:B,32:B,44:B,26:B,13:B,9:B,15:B,48:B,20:B,47:B)
spacefill off
define hh 33:B,36:B,37:B,32:B,44:B,26:B,13:B,9:B,15:B,48:B,20:B,47:B
select (oxygen) and hh
color red
select (nitrogen) and hh
color blue
zoom 150
Анализ формы спирали
На данный момент мне известны два типа определения формы спирали (А или В форма). Первый - с помощью программы RasMol, опираясь на визуальное впечатление и небольшие механические
(сделанные вручную) вычисления. Второй - с помощью программы analyse.
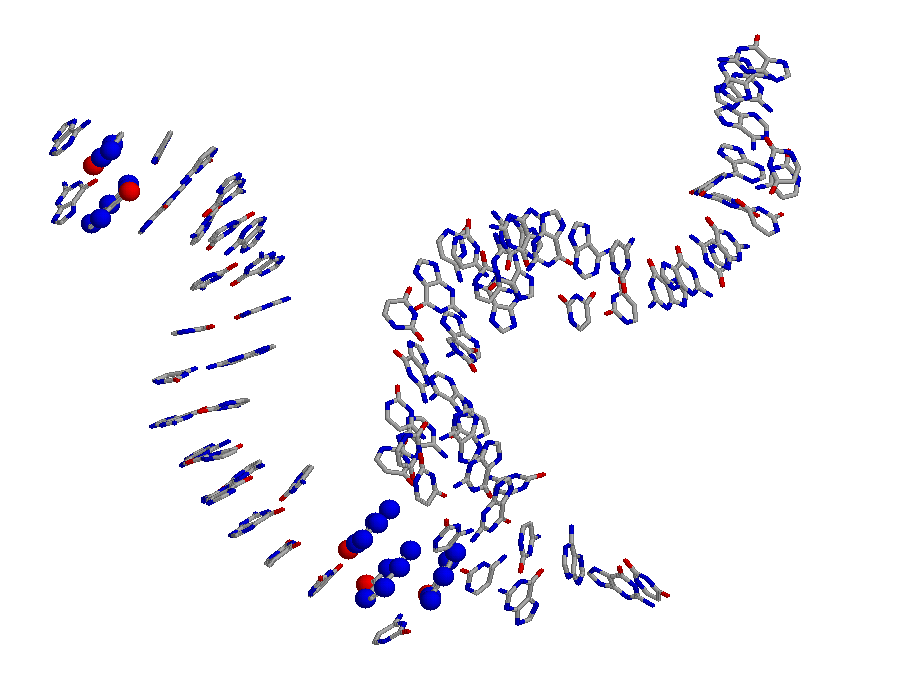
Ниже приведена картинка, построенная с помощью програмы Pymol. Здесь изображена тРНК нукле-белкового комплекса из pdb-файла 1h3e.pdb, в модели cartoons, раскраска chain.
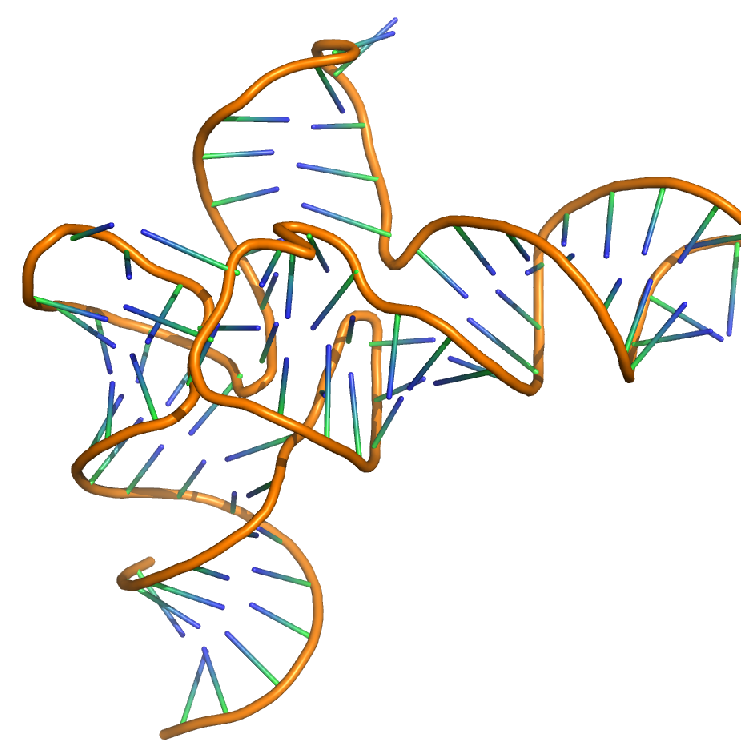
Если рассмотреть эту структуру тРНК, нельзя сделать какого-то однозначного вывода о форме ее спиралей, т.к. эти спирали достаточно сложно выделить, две спирали не очень длинные,
а последняя вообще состоит из одного остатка. Конечно, можно было воспользоваться командой find_pair, но мне хотелось всё же попытаться сделать это каким-то образом, опираясь
исключительно на визуальную оценку.
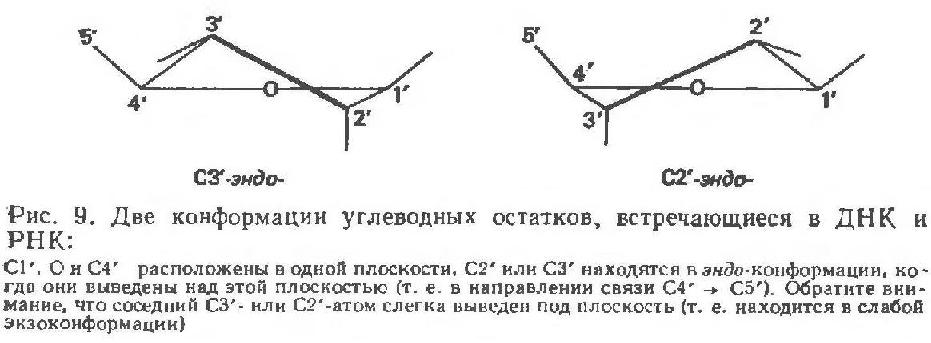
Как известно, одним из основных отличий А- и В-форм является конформация, которую приминает второй углерод рибозы.Если он принимает C2-эндо конмормацию, то это А-форма, а
если С3-эндо - то это В-форма спирали (А.С. Спирин. Структура и биосинтез нуклеиновых кислот, глава "Макромолекулярная структура нуклеиновой кислоты", стр. 23). На картинке ниже как
раз изоюражены две такие конформации. Рисунок взят из того же учебника (А.С. Спирин. Структура и биосинтез нуклеиновых кислот):
Эта картинка уже построена вручную и иллюстрирует конкретную тРНК, исследуемую нами. Здесь изображен произвольно взятый остаток из области тРНК, более или менее напоминающий
спираль. Остаток брался и из первой, и из второй спирали абсолютно произвольно. Остаток изображен в модели sticks, раскраска по атомам (с помощью программы Pymol):
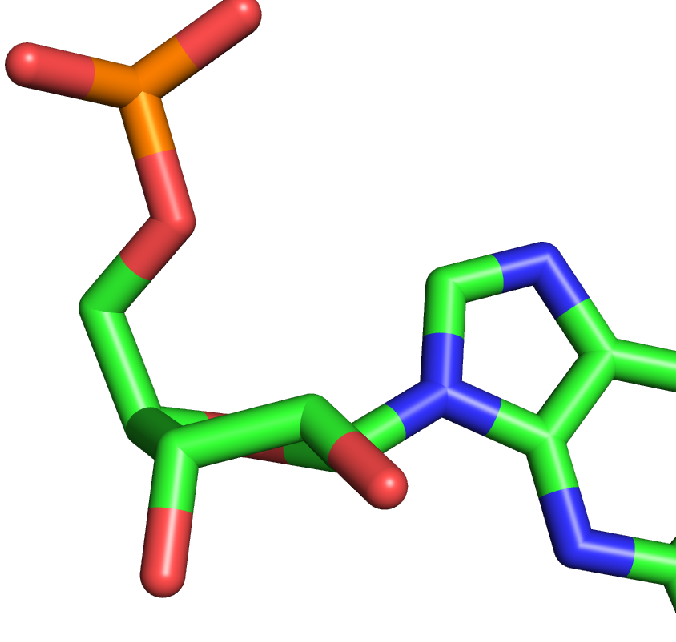
Видно, что углерод находится в C2-эндо конмормации. Делаем вывод - эти спирали больше напоминают А-форму.
Теперь возьмем произвольный остаток из петли, а не из более или менее напоминающего спираль участка. Понятно, что он не может быть похож на ту или иную форму спирали, т.к. петли
исследуемой тРНК сами по себе не похожи на спирали. Однако интересно, в какой конформации находится углерод на этом участке.
Остаток (опять же взяли несколько произвольно выбранных остатков и сравнили - получили один результат) изображен в модели sticks, раскраска по атомам (с помощью программы Pymol):
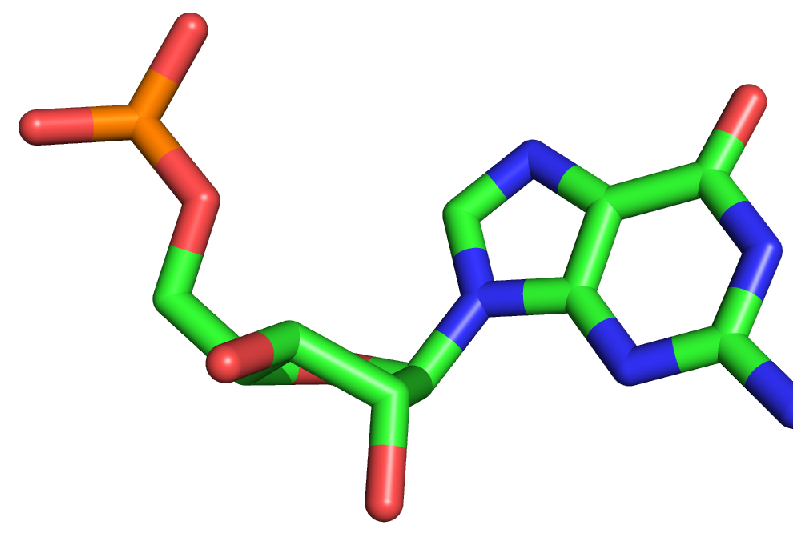
А этот углерод уже принимает C3-эндо конмормацию. Конечно, из этого нельзя делать вывод, что петля похожа на А-форму спирали. Однако вполне логично, что углерод принимает
другую конформацию, потому что если бы конформация не менялась, спираль скорее всего продолжалась бы и не удалось бы образовать такие причудливые петли.
Итак, делаем вывод, что спирали исследуемой тРНК больше напоминают А-форму.
Задание №5. Найти возможные двуспиральные участки в последовательности исследуемой РНК с помощью программы einverted из пакета EMBOSS.
Для выполнения команды с помощью программы einverted был использован файл trna.fasta, в котором модифицированные основания были заменены на N, что значит "любой".
При запуске программы строка запроса в Unix следующая:
einverted
т.е. пишем только название команды, всё остальное она спросит сама. Она спрашивает, например, имя файла, с которым надо работать, другие параметры : gap penalty (оставляем по
умолчанию 5), match score (оставляем по умолчанию 5), mismatch score (оставляем по умолчанию -4) и minimum score threshold (меняем только его - с 50 до 20. Иначе никаких
результатов не было получено). Вот что выдала программа einverted. Причем если проводить работу с параметром
minimum score threshold от 50 до 25, то на выходе получаем пустой файл. А если значение этого параметра - от 20 до 5, то информация в выходном файле одинаковая (см. выше).
Это связано с тем, что инвертированные повторы считаются за выравнивания, соответственно вес зависит от количества комплементарных пар. Т.к. участки, подаваемые на вход,
достаточно малы, приходится понижать порог.
Итак, программа einverted нашла один достаточно короткий участок. А вот программа find_pair нашал их больше, что подтверждается визуальным анализом с помощью
RasMol. Возможно, это связано с тем, что einverted учитывает не учитывает неканонические взаимодействия, а они на самом деле очень важны при формировании вторичной
структуры тРНК. Т.к. они не посчитаны, получается, что комплиментарных канонических взаимодействий достаточно мало - поэтому и такое короткий кусочек, причем только один. Это
лишний раз доказывает предположение, что неканонические взаимодействия чрезвычайно важны и про них ни в коем случае нельзя забывать.
Задание №6. Проанализировать последовательность тРНК программой mfold .
Для создания файлов с помощью программы mfold был использован файл tRNA_1.fasta, в котором все
модифицированные основания были заменены на близкий канонический нуклеотид (PSU, 5MU->U; MAD->A). Также была создана отдельная папка, потому что программа выдает очень много
разных файлов (mfold - по названию программы), в которой и проводилась работа. Для работы использовалась команда mfold . Строка запроса в Unix:
mfold SEQ=tRNA.fasta P=X
- значение параметра, которое изменялось от 10 до 20% - на сколько процентов энергия выдаваемой структуры может отличаться от оптимальной.
Было произведено несколько запросов - про возможном отклонении энергии на 10%, 15% и 20%. В выдаче ОЧЕНЬ много различны файлов, среди них есть *.GIF, в которых содержатся
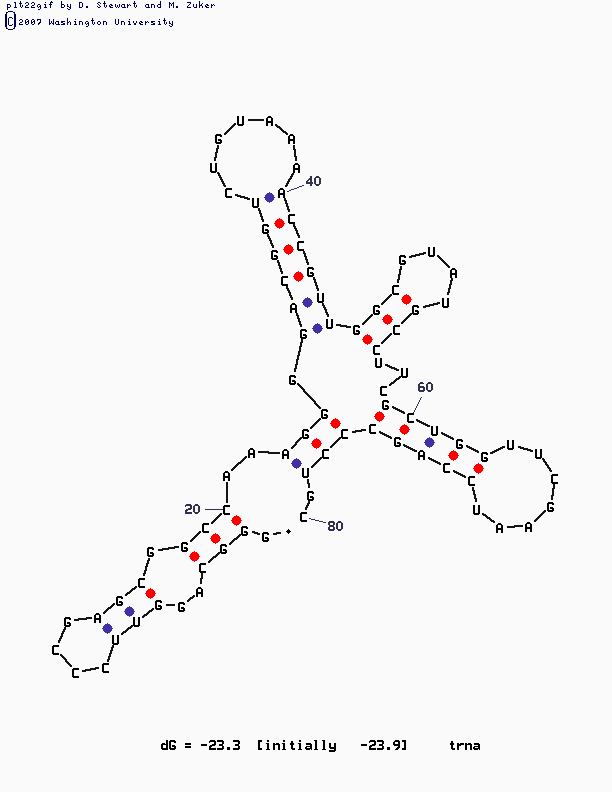
различные изображения тРНК. Надо было выбрать картинку, на которой структура тРНК наиболее похожа на классическую - в форме кленового листа:
Однако из-за объясненных выше особенностей данного для исследования файла со структурой тРНК изображения получились не очень хорошие, т.е. не стопроцентно удовлетворяющие
предъявляемым требованиям. Скорее всего из-за большой длины последовательности mfold просто не до конца справился со своей задачей, и поэтому получились не очень хорошие
рисунки. Из того, что получилось, был выбран наиболее похожий на клеверный листок:
Отчет в формате *.doc можно посмотреть здесь
© Pouliakhina
All rights reserved