Главная
Практикум №10: "Выравнивание геномов"
Задание 1. Построение карты сходства хромосом двух родственных бактерий.
Были выбраны следующие бактерии: Rickettsia rickettsii и Rickettsia prowazekii. Данные бактерии содержат
одну хромосому.
C помощью blast2seq было постороено выравивание данных геномов, а так же карта сходства. Рис.1 подтверждает, что
геномы данных видов одного рода иногда имеют приемлемое сходство - 92% идентичных позиций.
 Рис. 1. Некоторые характеристики полученного выравнивания.
Рис. 1. Некоторые характеристики полученного выравнивания.
На Рис.2 представлена карта сходства хромосом данных видов.
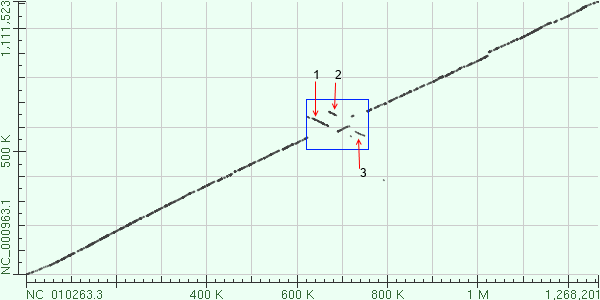 Рис. 2. Карта сходства хромосом. Координаты генома Rickettsia rickettsii
(NC_010263.3) откладываются по оси X,
Рис. 2. Карта сходства хромосом. Координаты генома Rickettsia rickettsii
(NC_010263.3) откладываются по оси X,
а Rickettsia prowazekii (NC_000963.1) - по оси Y.
Значительные эволюционные события произошли в месте, выделенном на Рис.2 синим цветом.
Данный участок имееет координаты приблизительно 630-760K (по хромосоме Rickettsia rickettsii). Скорее всего здесь произошла
транслокация - поменялись местами первый и второй фрагменты хромосомы с третьим фрагментом (Рис.2). Так как между данными областями присутствует
неизменённый участок, то можно предположить, что транслокация произошла даже между фрагментами хромосомы, находящимися
на некотором расстоянии. Кроме инверсии, данные участки генома, очевидно, претерпели ещё и инверсию.
Задание 2. Сходство и различие геномов близкородственных бактерий.
С помощью пакета NPG-explorer был построен нуклеотидный пангеном (NPG). В качестве материала использовались 4 штамма бактерии Rickettsia
rickettsii, каждый из которых имеет одну хромосому.
Файл с информацией о последовательности геномных ДНК и аннотации генов Здесь.
Протокол выполнения создания нуклеотидного пангенома Здесь.
Описание синтеничных участков - g-блоков - и их перестановок.
Информация о блоках была получена из файла blocks.gbi. Было выяснено, что в
хромосоме каждого штамма присутствует лишь один глобальный блок: g4x1269809. Данный блок состоит из 4 фрагментов и содержит в выравнивании
1269809 позиций.
Далее, используя информацию из файла blocks.blocks было выяснен порядок локализации
g-блоков в хромомоме. Но так как в хромосоме лишь один глобальный блок, то единственная полезная информация заключается в том, удалось выяснить,
на какой цепи расположен данный блок. В хромосоме каждого штамма он расположен на прямой цепи (">")
chr1 +Arizona&chr1&c g4x1269809 >
chr1 +Iowa&chr1&c g4x1269809 >
chr1 +Morgan&chr1&c g4x1269809 >
chr1 +R&chr1&c g4x1269809 >
Фрагмент выравнивания данных блоков в хромосомах четырёх штаммов представлен на Рис.3.
 Рис. 3.
Рис. 3.
Описание ядра геномов - s-блоков.
Информация о стабильных блоках была получена из файла pangenome.info
Число s-блоков - 108. Каждый s-блок состоит из 4 фрагментов.
Суммарная длина и процент от длины генома в среднем: 1240151 (98.39%),
Процент консервативных позиций в объединенном выравнивании s-блоков: 0.999001.
Описание блоков с повторами (r-блоки).
Использовался файл pangenome.gi. Два наиболее частовстречающихся блока:
r22x159. Имеет 22 повтора в рассматриваемых геномах, длина: 159. 2 раза встречается в хромосоме штамма Arizona, 6 раз в штамме Iowa,
по 7 раз в штаммах Morgan и R.
r16x225. Имеет 16 повторов, длина: 225. В хромосоме штамма Arizona присутствует 1 раз, Iowa - 3, в штаммах Morgan и R - по 6.
Описание крупных делеций на примерах h-блоков (полустабильных блоков).
На Рис.4 представлена информация о всех h-блоках, присутствующих в хромосомах штаммов.
 Рис. 4.
Рис. 4.
Видно, что у штамма R есть лишь однин полустабильный блок - h3x115, у штаммов Iowa и Morgan присутствуют все 7 возможных h-блоков,
у Arizona - 6 (нет h3x115).
Рассмотрим участок h3x115. Данный участок длиной 115 содержится во всех штаммах, кроме Arizona. Следовательно, можно предположить, что у данного
штамма произошла делеция данного участка хромосомы. Всё это подтверждается визуализатор qnpge (Рис.5 и Рис.6)
 Рис. 5.
Рис. 5.
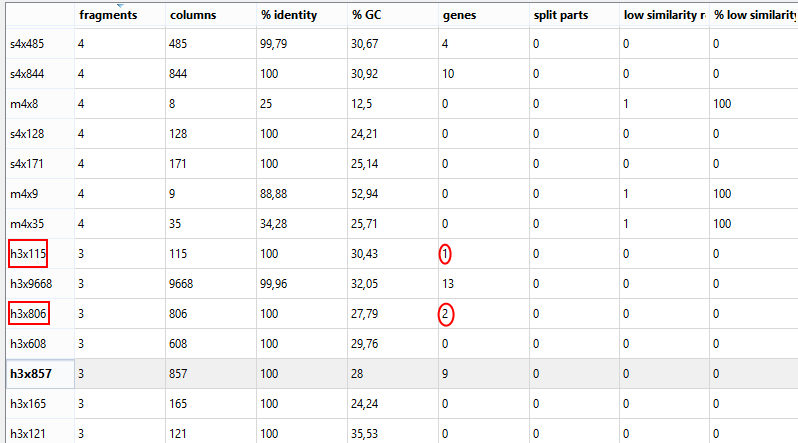 Рис. 6.
Рис. 6.
На данном участке располагается фрагмент одного ген (в штамме Iowa) - ген гипотетического белка (прямая цепь).
Рассмотрим участок h3x806. Данный участок длиной 806 содержится во всех штаммах,кроме R. Следовательно, можно предположить, что у данного
штамма произошла делеция данного участка хромосомы. Всё это подтверждается визуализатор qnpge (Рис.7a и Рис.6)
 Рис. 7a.
Рис. 7a.
На данном участке кодируется 2 гена. Один (в штамме Arizona) - ген 3-гидроксиацил-КоА дегидрогиназы (обратная цепь, Рис.7a).
 Рис. 7b.
Рис. 7b.
Другой - ген гипотетического белка (в штамме Iowa, Рис.7b) - так же на обратной цепи. Что интрерсно, фрагменты обоих этих данных генов, лежащих на данных
учасках имеют одинаковую длину (231 нуклеотид) и цепь (обратную). Можно предположить исходя из этого, что гипотетический белкок (в штамме Iowa) как раз и
является белком 3-гидроксиацил-КоА дегидрогиназой. Так же интересно отметить, что очень похожая последовательность в штамме Morgan на данном участке
является некодиующей согласно визуализатору qnpge.
Уникальных последовательностей, не имеющих гомологов среди всех геномов, кроме себя, найдено не было. Это, вероятно, можно объяснить тем, что
все четыре генома имеют очень хначительное сходство и просто не имеют таких u-блоков.
Примеры расхождений между аннотациями генов из одного блока.
Одно из таких расхождений уже было описано для блока h3x806. В нём в штамме Arizona аннотирован ген 3-гидроксиацил-КоА дегидрогиназы,
ген гипотетического белка аннотирован в штамме Iowa, а в штамме Morgan данный участок не признаётся за ген.
Ещё одно подобное расхождение присутствует в блоке s4x477. Здесь во всех штаммах, кроме Arizona, аннотирован ген NTPaзы длиной на данном блоке
90 нуклеотидов, располагающийся на обратной цепи (Рис.8). Однако для штамма Arizona ген не аннотирован.
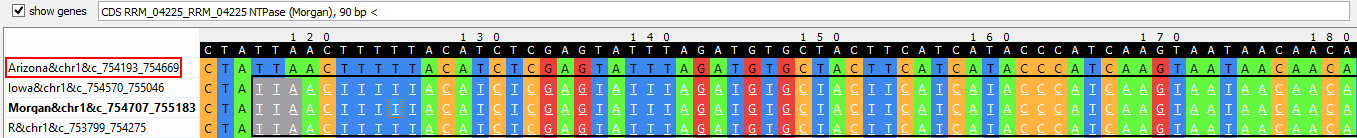 Рис. 8. Штаммы с аннотированным геном взяты в чёрный прямоугольник.
Рис. 8. Штаммы с аннотированным геном взяты в чёрный прямоугольник.
Штамм, в котором ген не аннотирован выделен красным цветом. Нуклеотиды кодирующей области подчёркнуты.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ









