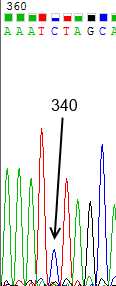
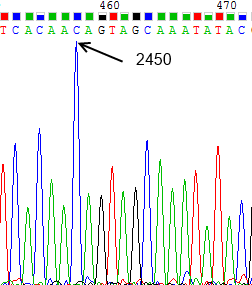
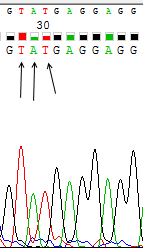
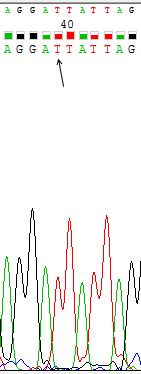
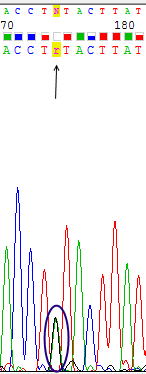
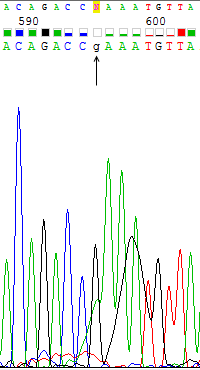
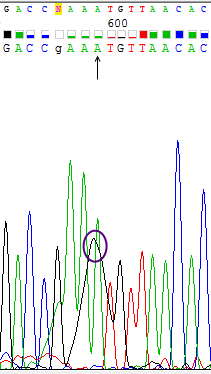
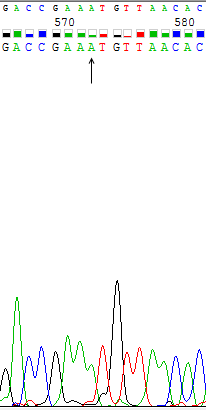
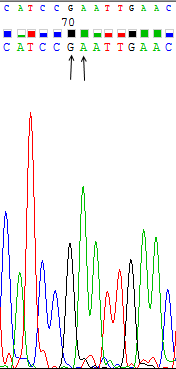
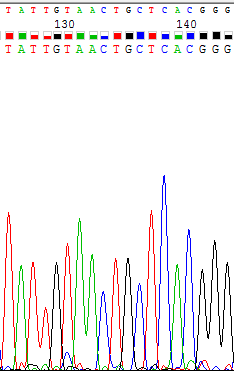
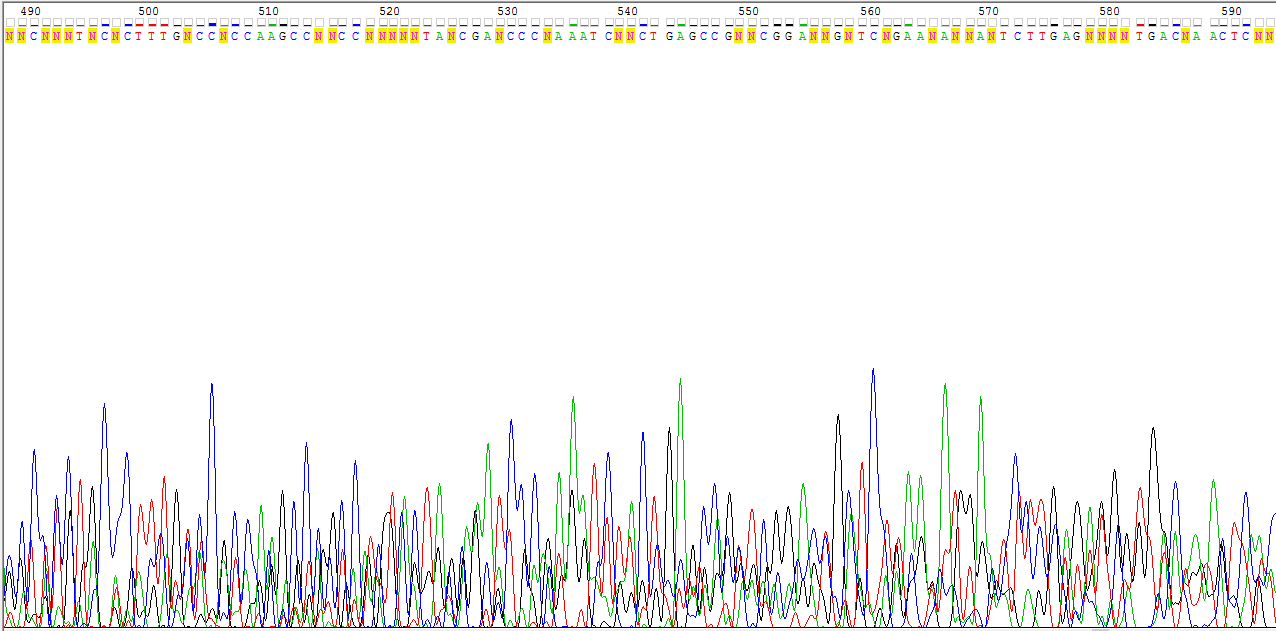
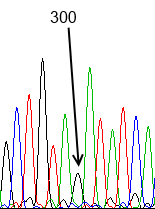
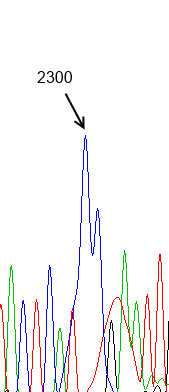
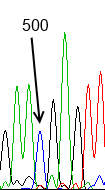
Сила сигнала не зависит от нуклеотида, так как, например, для цитозина есть сила сигнала, равная 2300, но для других цитозинов можно найти и довольно слабые сигналы (около 500, Рис.3), и, наоборот, на Рис.1 представлен пик для гуанина с очень слабой силой сигнала (300), но на Рис.4 представлен пик для другого гуанина с силой сигнала около 2200.
 |
 |
 |
 |
Характеристика хроматограммы обратной цепи: