Главная
Практикум №8: "Поиск по сходству (blast)"
Задание №1
Определение таксономии и функции нуклеотидной последовательности.
Была взята последовательность, полученная в практикуме №6: последовательность.
С помощью BLASTN было установлено к какому гену принадлежит эта последовательность, а так же определена таксономия организма.
Blast нашёл для данной последовательности несколько достоверных находок, из которых были выбраны 3 лучшие: Query cover 100%, E-value
равно 0 и процент идентичных позиций превышает 90% (Рис.1).
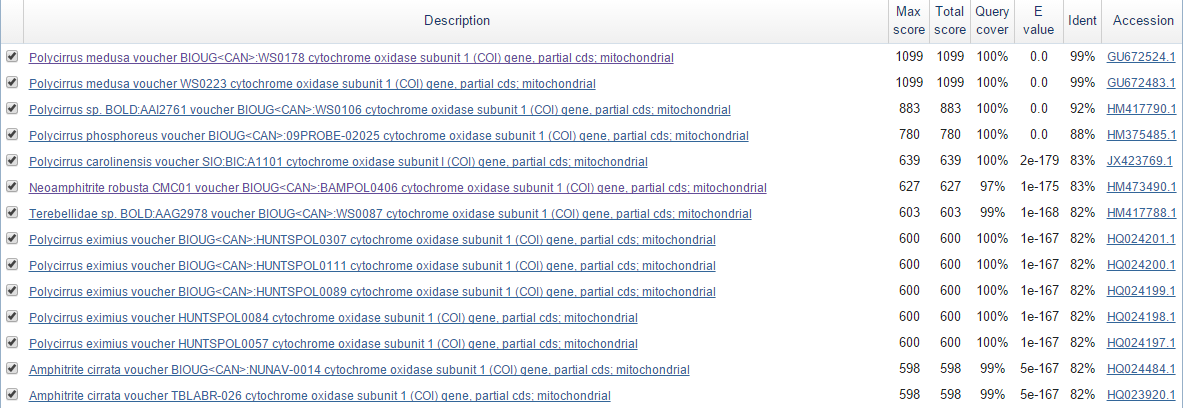 Рис. 1.
Рис. 1.
Последовательности во всех трёх находках представляют собой один и тот же ген, кодирующий субъединицу 1 митохондриального фермента
цитохромоксидазу (COI) (Рис.2). Первые две находки - из организма Polycirrus medusa (на Рис.2), третья находка -
из организма Polycirrus sp. BOLD:AAI2761 (Рис.3).
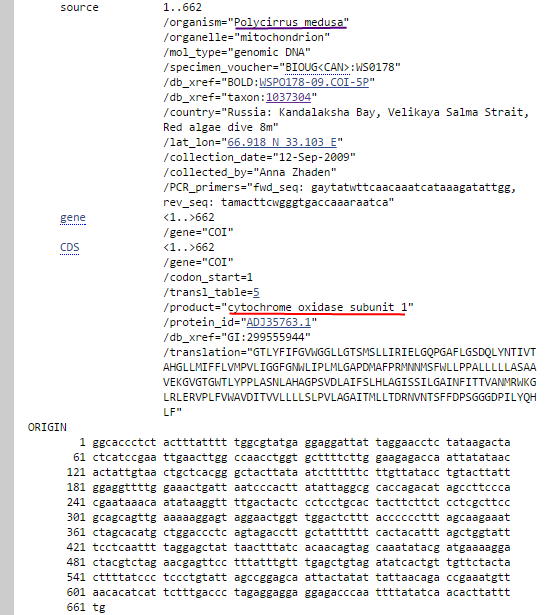 Рис. 2. Красным подчёркнуто название продукта гена, фиолетовым - название организма.
Рис. 2. Красным подчёркнуто название продукта гена, фиолетовым - название организма.
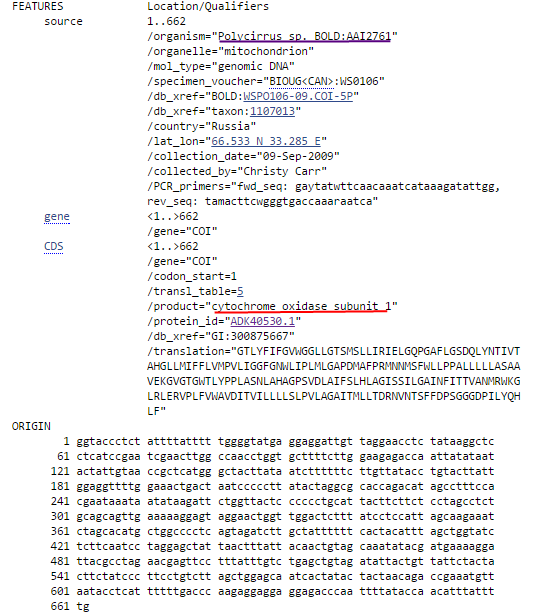 Рис. 3. Красным подчёркнуто название продукта гена, фиолетовым - название организма.
Рис. 3. Красным подчёркнуто название продукта гена, фиолетовым - название организма.
Оба организма принадлежат к одному роду многощетинковых кольчатых червей - Polycirrus. Таксономия (взята в прямоугольник) представлена на Рис.4.
 Рис. 4.
Рис. 4.
Далее были построены выравнивания входной последовательности с находками:
Выравнивание с первой находкой
Выравнивание со второй находкой
Выравнивание с третьей находкой
Таким образом, можно сделать вывод, что данная изучаемая последовательность так же является геном, который кодирует
первую субъединицу цитохромоксидазы полихет из рода Polycirrus.
В лучшей находке всего 1 замена на 611 пар оснований (Рис.6), то есть приблизительно 0,16 замен на 100 пар оснований.
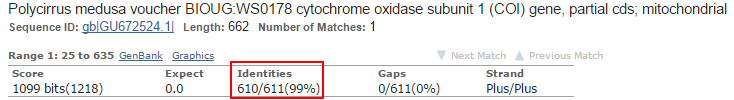 Рис. 6.
Рис. 6.
Лучшая находка из другого рода (Рис.7) Neoamphitrite (семейство то же Terebellidae) имеет уже 99 замен на 594 пар оснований (Рис.8), тогда
на 100 п.о. замен примерно 16,87, что в сто раз больше, чем в лучшей находке!
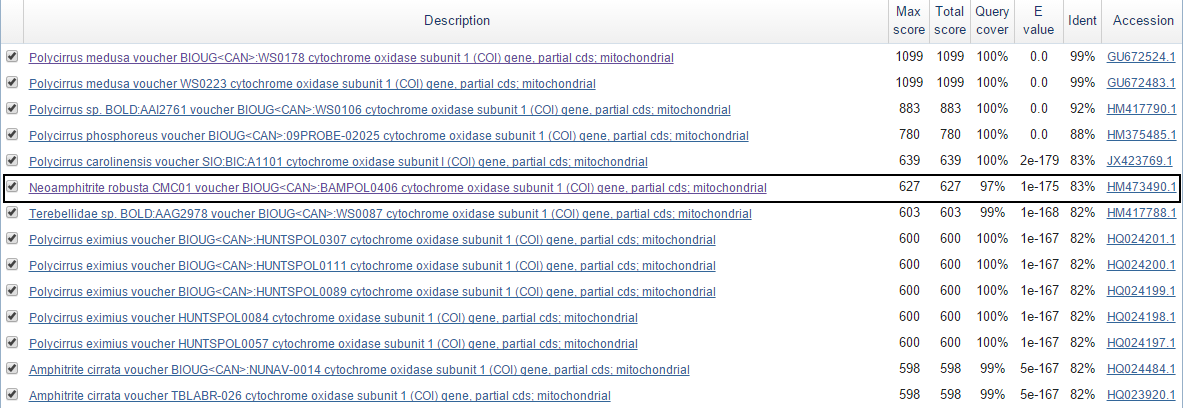 Рис. 7. Лучшая находка другого рода взята в прямоугольник.
Рис. 7. Лучшая находка другого рода взята в прямоугольник.
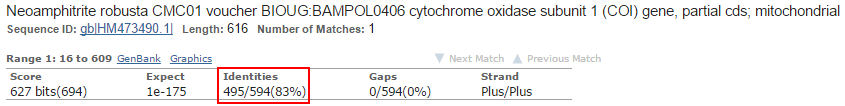 Рис. 8.
Рис. 8.
Задание №2
Сравнение списков находок нуклеотидной последовательности 3-я разными алгоритмами blast.
Поиск по алгоритму blastn.
Просто по данной последовательности blastn находил более 1000 находок, поэтому область поиска была ограничена семейством, к которому принадлала лучшая
находка - Terebellidae (taxid:32261). В результате было найдено 106 находок (Рис. 9).
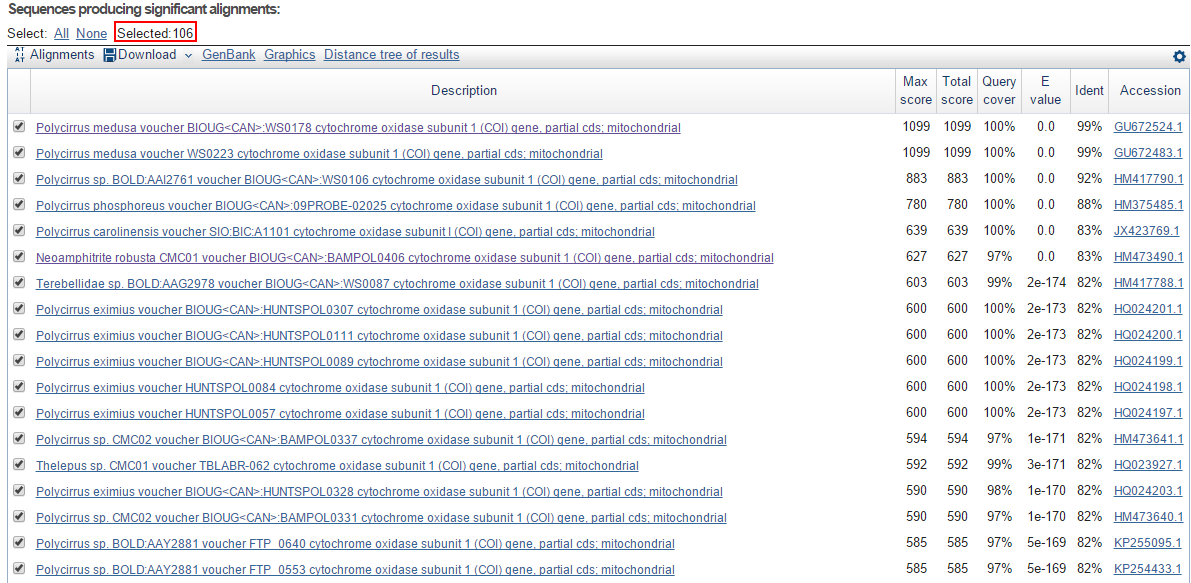 Рис. 9.
Рис. 9.
У худшей находки (Рис. 10) E-value составляет 1e-50; замен 196 на 595, то есть приблизительно 33 на 100 пар оснований (33%). Идентичных позиций 67%.
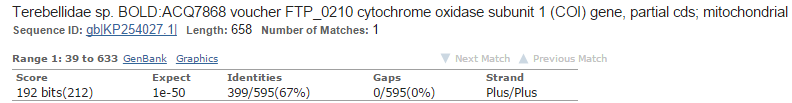 Рис. 10.
Рис. 10.
Поиск по алгоритму megablast (остальные параметры не были изменены).
Было найдено всего 10 находок (Рис. 11).
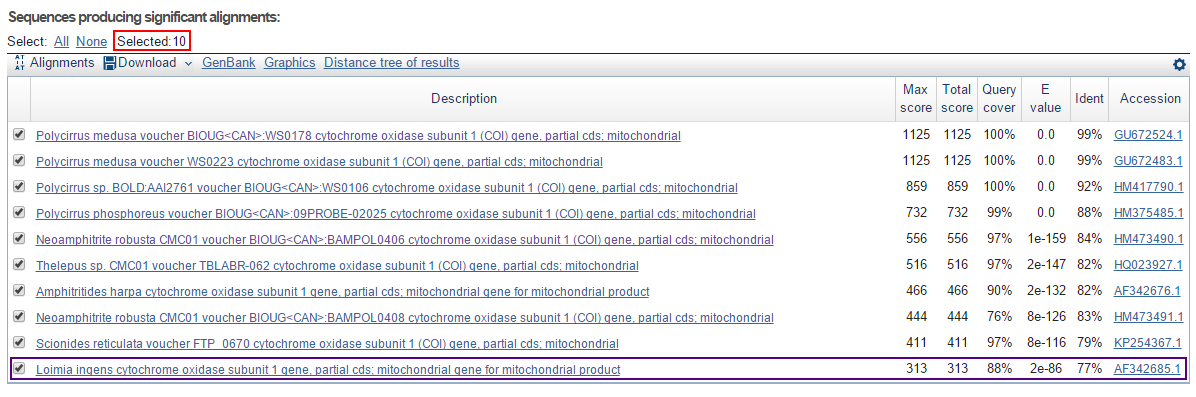 Рис. 11.
Рис. 11.
E-value худшей находки (Рис.12) составляет 2e-86; замен 124 на 543, то есть приблизительно 23 на 100 пар оснований (23%). Идентичных позиций 77%.
 Рис. 12.
Рис. 12.
Поиск по алгоритму discontiguous megablast (остальные параметры не были изменены).
Было найдено всего 108 находок (Рис. 13).
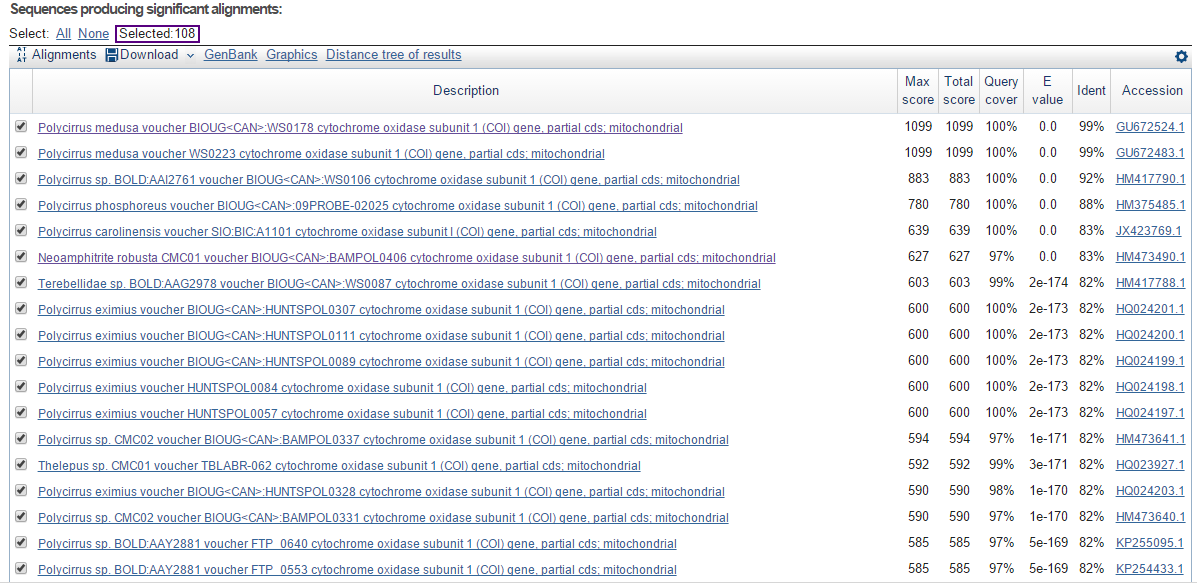 Рис. 13.
Рис. 13.
E-value худшей находки (Рис.14) составляет 2e-04; замен 20 на 71, то есть приблизительно 28 на 100 пар оснований (28%). Идентичных позиций 72%.
 Рис. 14.
Рис. 14.
По сравнению с blastn discontiguous megablast нашёл ещё две находки (выделены красным на Рис. 15.). Можно заметить, что у этих находок
query cover горздо меньше по сравнению с остальными (которые нашёл и blastn) - всего 11% (то есть всего 11% от длины последовательности выравнено с
входной последовательностью. Это удивительно, так как blastn ищет хоть что-нибудь сходное, и должен найти больше всех находок в сравнении с другими алгоритмами.
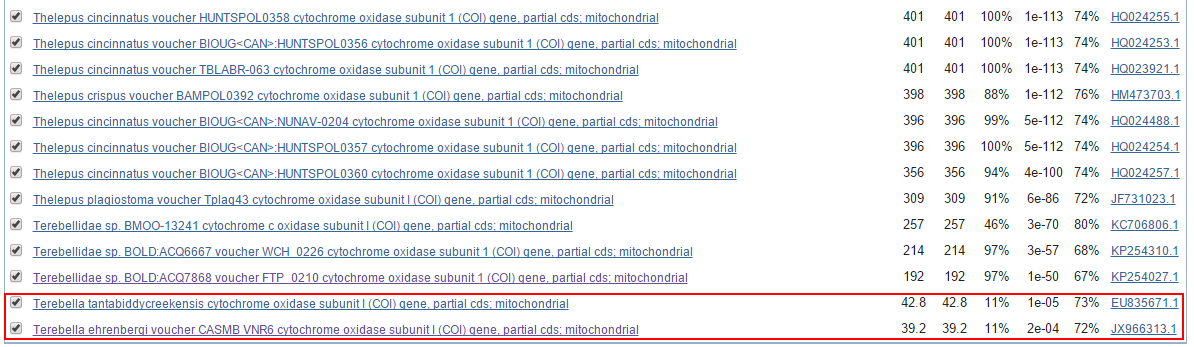 Рис. 15.
Рис. 15.
Megablast ищет гомологи, которые обладают очень большим сходством с входной последовательностью (Рис.17a). Но он, например, не нашёл находку, пятую
в списке находок blastn, которая кажется очень даже хорошей: E-value = 0, query cover 100%. (обведена в красный прямоугольник на Рис.17b).
Возможное объяснение этому заключается в том, что word size в megablast значительно превышает таковой в blastn (28 против 11). То есть он делит
входную последовательность на фрагменты длины 28 по ним ищет гомологов, постепенно "расширяя" выравнивание. Видимо, из рассматриваемой находки (которую нашёл blastn)
не нашлось ни одного фрагмента длины 28, полностью совпадающего с каким-либо фрагментом запроса длины 28, и, следовательно, megablast не нашёл её.
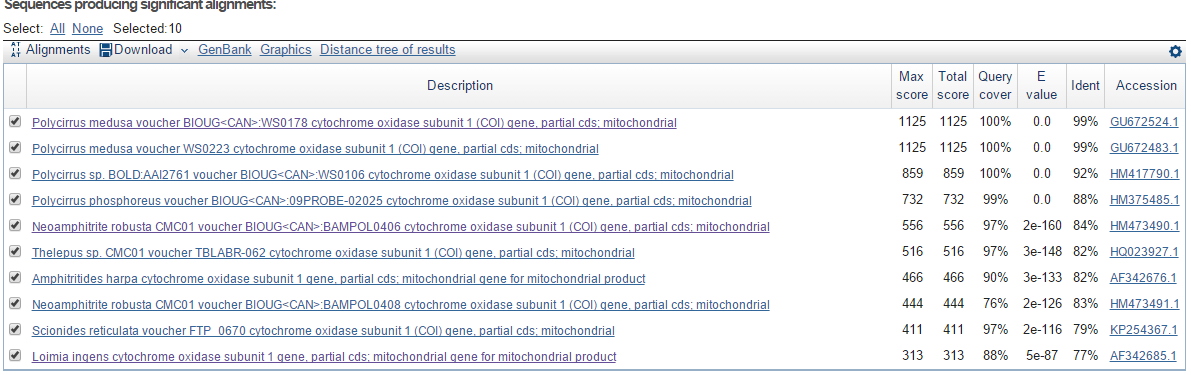 Рис. 17a.
Рис. 17a.
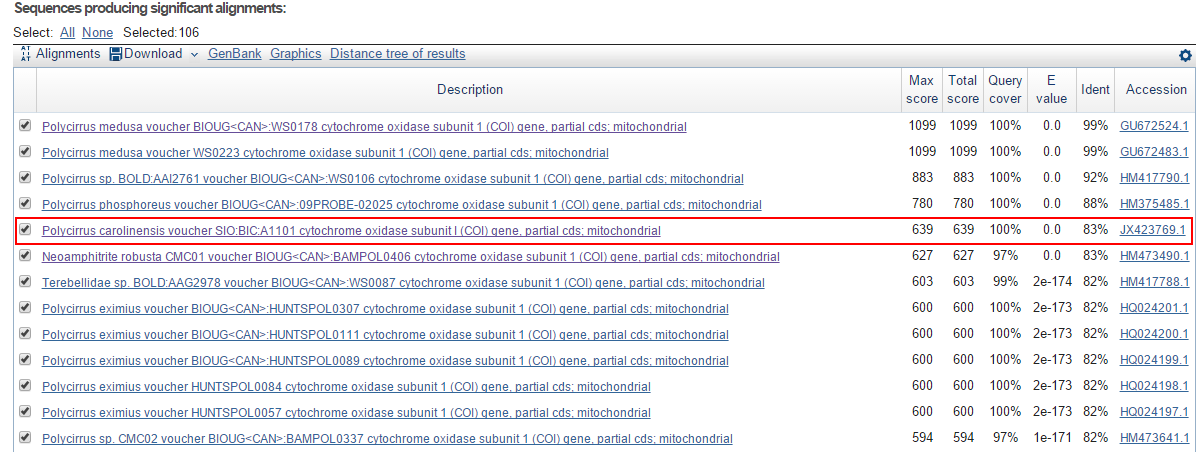 Рис. 17b.
Рис. 17b.
Задание №3.1
Проверка наличия гомологов пяти белков в геноме Acetobacter pomorum (TBLASTN) (организм из предыдущего практикума, очевидно, нелучший выбор
для поиска гомологов белков человека, тем не менее довольно интересно выяснить, какие из белков человека могут иметь сходных представителей в клетках
прокариот.
HSP7C_HUMAN (Heat shock 70 kDa protein 8) UniProtKB - P11142 (fasta-последовательность)- белок теплового шока человека, относящийся к шаперонам, который кодируется геном HSPA8 в восьмой хромосоме.
Основная функция - фолдинг белков, участие в апоптозе, так же предотвращение сворачивание белков в ходе посттрансляционного транспорта в митохондрии и хлоропласты.
C использованием tblastn поиска белковой последовательности данного белка против трансляции нуклеотидного банка данных в шести рамках.
Были обнаружены 2 гомолога данного белка из бактерии Acetobacter pomorum (Рис. 18).
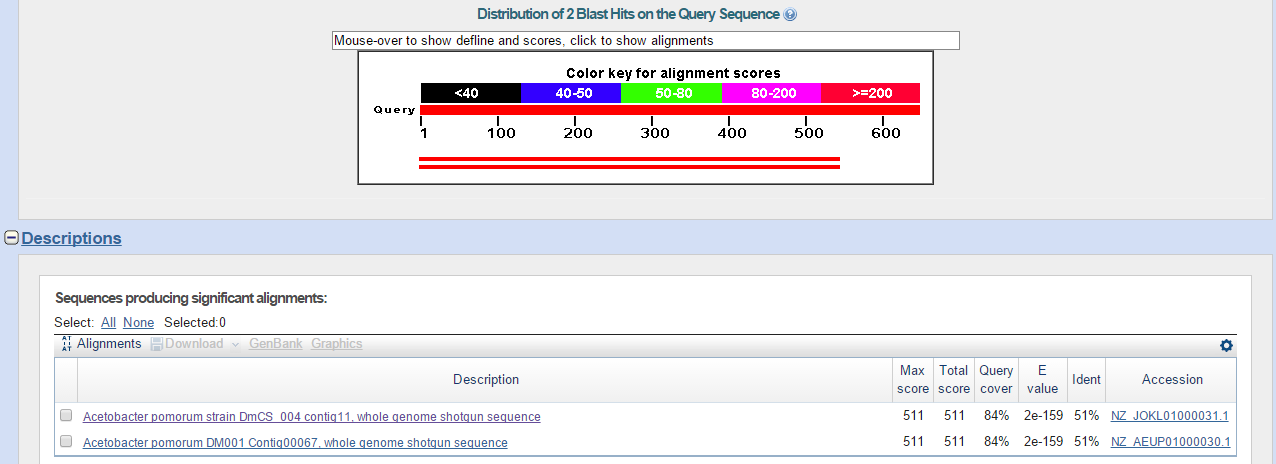 Рис. 18.
Рис. 18.
Обе находки довольно предсставляют собой идентичные выравнивания с одинаковыми значениями счёта, E-value и др., но одна - из штамма DmCS, а другая -
из штамма DM001.
Гомологи шаперонов эукариот действительно известны у бактерий,так, например, у прокариот есть шаперон DnaK с массой 70 кДа,роль которого сходна
с ролью человеческого Heat shock 70 kDa protein 8.
Параметры находок: E-value составляет 2e-159, процент идентичных позиций: 51%, query cover: 84%, счёт выравнивания: 511.
TERT_HUMAN (Telomerase reverse transcriptase) - UniProtKB - O14746 (fasta-последовательность)- каталитическая субъединица теломеразы, использует теломеразную РНК в качестве
матрицы, добавляя повторяющуюся последовательность из шести нуклеотидов 5'-TTAGGG к 3'-концу хромосом.
Для бактерии Acetobacter pomorum не было найдено ни одной находки. Тогда область поиска была расширена: я стал искать гомологи TERT_HUMAN
во всех бактериях. Снова ни одной находки. Объяснение довольно банальное: в клетках бактерий нет теломеразы, поскольку у
кольцевых бактериальных хромосом нет концов.
Даже у бактерий, имеющих линейные хромосомы (например, у Agrobacterium tumefaciens), все равно нет теломеразы.
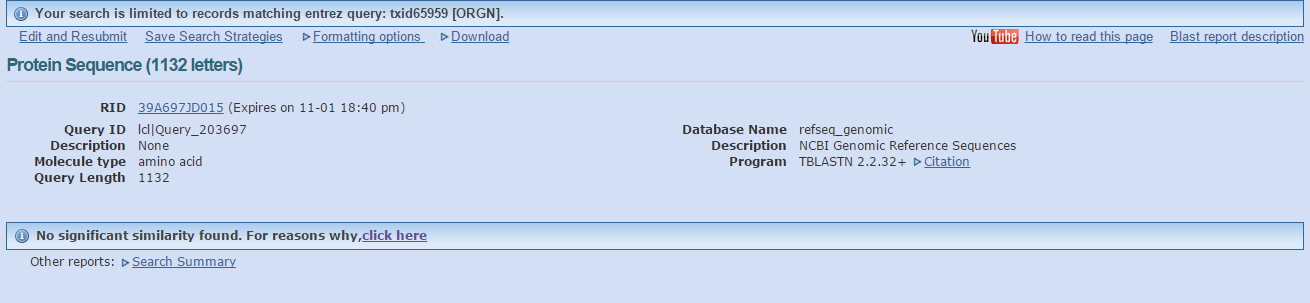 Рис. 19. Отсутствие гомологов TERT_HUMAN среди белков бактерий.
Рис. 19. Отсутствие гомологов TERT_HUMAN среди белков бактерий.
CISY_HUMAN (Citrate synthase) - UniProtKB - O75390 (fasta-последовательность)
- митохондриальный фермент цитратсинтаза, который участвует в Цикле трикарбоновых кислот,
катализируя реакцию конденсации ацетата (ацетил-CoA) и оксалоацетата, в результате чего образуется цитрат. Обнаружена
практически во всех клетках аэробных организмов, к которым относится и Acetobacter pomorum.
Было найдено 4 находки (Рис.20).
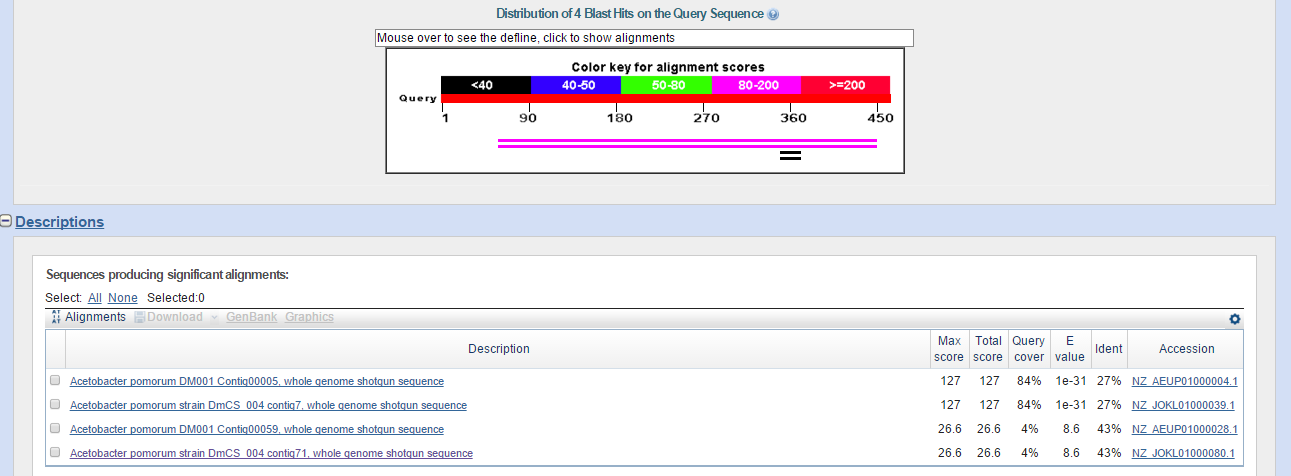 Рис. 20.
Две последние находки очень сомнительны, так как имеют очень большое значение E-value, и ничтожное покрытие.
Первые две (снова идентичные выравнивания из разных штаммов) получше. Их параметры следующие:
E-value составляет 1e-31, процент идентичных позиций: 27%, query cover: 84%, счёт выравнивания: 127.
Рис. 20.
Две последние находки очень сомнительны, так как имеют очень большое значение E-value, и ничтожное покрытие.
Первые две (снова идентичные выравнивания из разных штаммов) получше. Их параметры следующие:
E-value составляет 1e-31, процент идентичных позиций: 27%, query cover: 84%, счёт выравнивания: 127.
Тем не менее здесь гомология весьма условная: Довольно низкий счёт и % идентичных позиций. И это логично: у ацетобактерий есть фермент
цитратсинтаза, но он функционирует у них в прямо в цитоплазме клетки, в то время как у человека в специализированных органеллах - митохондриях.
Следовательно, ферменты в любом случае будут отличаться, так как работают в различных условиях.
RPB1_HUMAN (RNA polymerase II subunit B1) - UniProtKB - P24928 (fasta-последовательность)-
субъединица B1 ДНК-зависимой РНК-полимеразы II, фермента, катализирующего транскрипцию мРНК.
Найдено 2 находки (Рис.21).
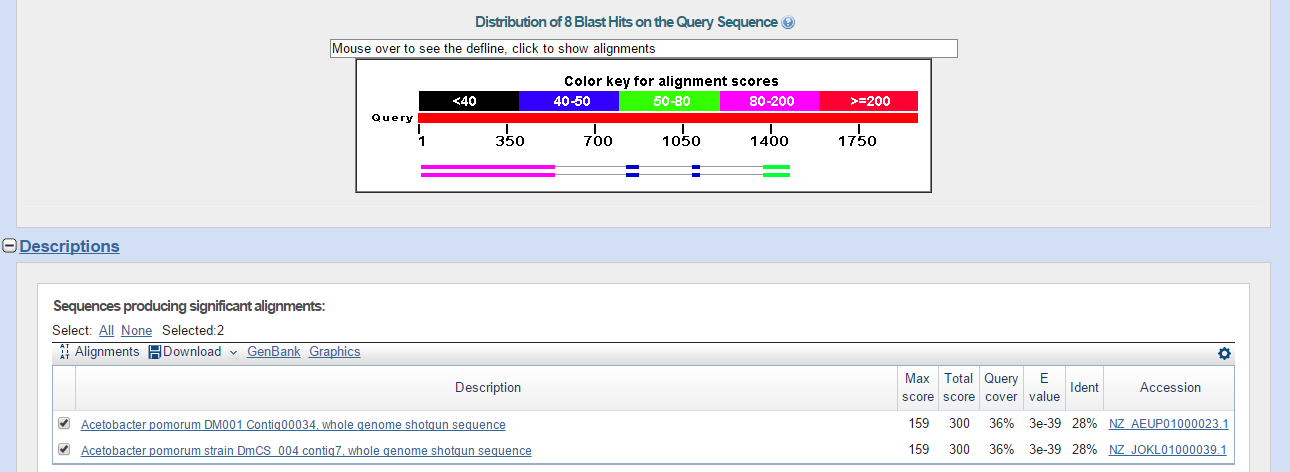 Рис. 21.
Рис. 21.
Они снова идентичные полностью из разных штаммов данной бактерии. Причём в каждой находке blast обнаружил по 4 сходных участка разной длины.
Параметры наиболее длинного выравнивания (Рис. 22): E-value составляет 3e-39, процент идентичных позиций: 28%, query cover: 36%,
счёт выравнивания: 159.
 Рис. 22.
Рис. 22.
И снова наблюдаем довольно условную гомологию. Стоит отметить, что у прокариот всего одна РНК-полимераза, которая катализирует синтез трёх типов РНК: мРНК, рРНК и тРНК.
Да и механизм транскрипции у человека и бактерии довольно сильно отличается. То есть РНК-полимераза бактерии и должна значительно отличаться
от РНК-полимеразы II человека.
PABP2_HUMAN (Polyadenylate-binding protein 2) UniProtKB - Q86U42 (fasta-последовательность)- локализованный в ядре
белок человека, который связывается с поли(А)хвостами. Белок необходим для постепенной
и эффективной полимеризации поли(А)хвостов на 3'-конце эукариотических генов и контролирует размер поли(А)хвоста на уровне примерно 250 нуклеотидов.
Данный белок локализуется в ядре, а этой структуры у бактерий вообще нет, как таковой. Следовательно, маловероятным представляется наличие
гомологов данного белка среди бактерий.
Гомологов из Acetobacter pomorum blast не нашёл, так же не было находок и в роду, и в семействе.
Это можно объяснить тем, что данный белок локализуется в ядре, а этой структуры у бактерий вообще нет, как таковой. Следовательно,
маловероятным представляется наличие гомологов данного белка среди прокариот.
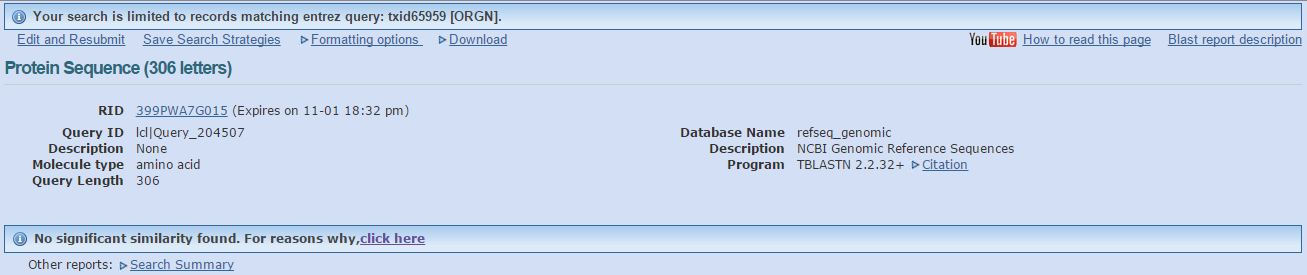 Рис. 23.
Рис. 23.
Задание №4
Оценка сходства геномов вирусов.
Выбранный вирус - Cherry rasp leaf virus. Данный вирус паразитирует на растениях, вызывая деформацию и скручивание их листьев (Рис.24).
 Рис. 24.
Рис. 24.
Далее были сохранены геном данного вируса Cherry rasp leaf virus (AJ621357.1), а так же пять геномов вирусов, принадлежащих к тому же семейству Secoviridae (в задании требуется взять
5 геномов вирусов из того же рода, но в данном роду Cheravirus были известны лишь сборки геномов 3 вирусов): Arabis mosaic virus
(идентификатор в INSDC: AY303786.1), Arracacha virus B (JQ437415.1), Bean pod mottle virus (U70866.1|BPU70866),
Beet ringspot virus (D00322.1|TBRRNA1), Tomato necrotic dwarf virus (KC999058.1). Все эти вирусы - двухцепочечные РНК-содержащие. Я брал для сравнения
только одну цепь РНК (RNA1 segment).
Затем все 6 полученных последовательностей были собраны в один файл ССЫЛКА с помощью команды seqret.
Была сделана база индексов blast для созданного fasta файла: makeblastdb -in virus.fasta -dbtype nucl.
Был запущен tblastx,на вход тот же самый fasta файл: tblastx -query virus.fasta -db virus.fasta -out blas.out -outfmt 7. В итоге
таблица с результатами была записана в файл blas.out.
С помощью скрипта были
удалены неинформативные и слабо сходные находки из полученной таблицы. Были установлены параметры для значение E-value, score и т.д. (Рис.25).
 Рис. 25. k.py - тот же скрипт.
Рис. 25. k.py - тот же скрипт.
В результате была получена: ТАБЛИЦА EXCEL
Далее была произведена сортировка по убыванию значения identity (процент идентичных позиций в находке). Оказалось,что первые пять находок
с наибольшим процентом идентичных нуклеотидов представляют собой сходные участки двух одних и тех же вирусов:
AJ621357.1 и JQ437415.1, (как раз нашего вируса Cherry rasp leaf virus и Arracacha virus B), что позволяет предположить, что именно геномы этих
двух вирусов наиболее схожи (Рис.26).
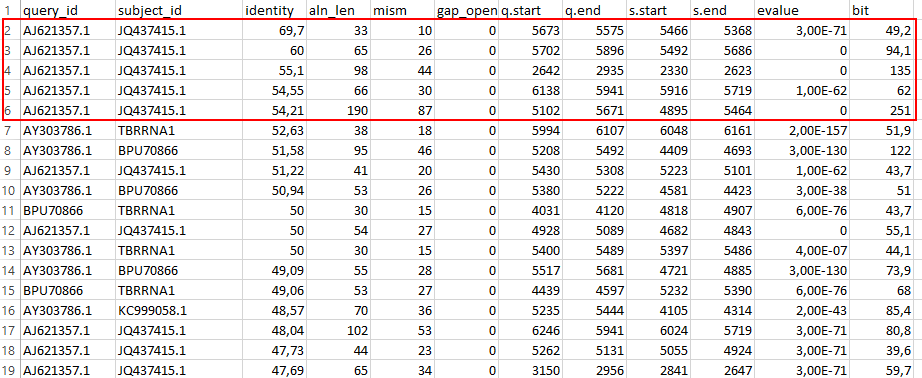 Рис. 26.
Рис. 26.
Далее сортировки по длине выравнивания (Рис.26a), его счёту (Рис.26b), E-value (Рис.26c) показали, что лучшие находки по
этим параметрам также представляют собой выравнивания данных вирусов.
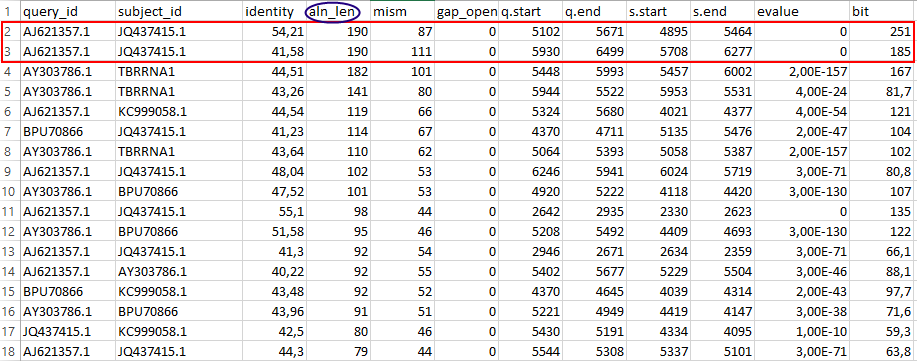 Рис. 26a.
Рис. 26a.
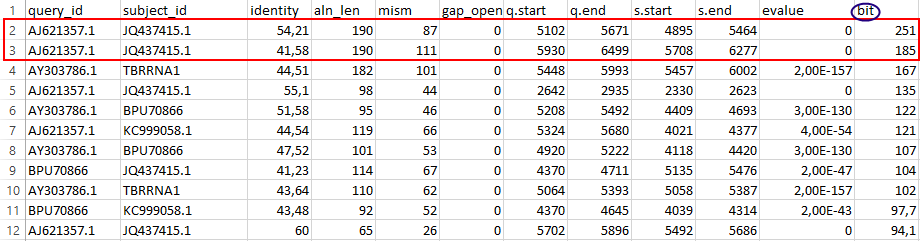 Рис. 26b
Рис. 26b
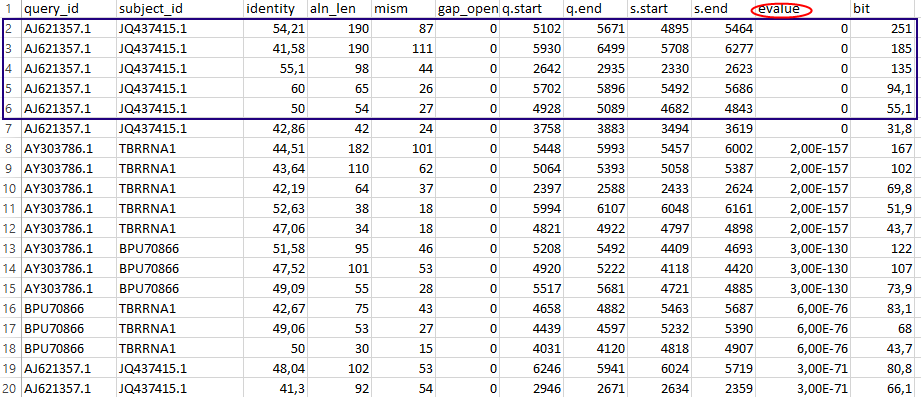 Рис. 26c
Таким образом, если оценивать сходство двух геномов вирусов по максимальной длине, максимальному скору, максимальному проценту идентичности
и минимальному E-value находок, тогда можно признать, что из данных вирусов наиболее сходные геномы имеют Cherry rasp leaf virus и Arracacha virus B.
Снова замечу, что для анализа были взяты лишь RNA1 сегменты данных вирусов. При аналогичном исследовании RNA2 сегментов этиъ же двухцепочных
вирусов возможны другие результаты.
Рис. 26c
Таким образом, если оценивать сходство двух геномов вирусов по максимальной длине, максимальному скору, максимальному проценту идентичности
и минимальному E-value находок, тогда можно признать, что из данных вирусов наиболее сходные геномы имеют Cherry rasp leaf virus и Arracacha virus B.
Снова замечу, что для анализа были взяты лишь RNA1 сегменты данных вирусов. При аналогичном исследовании RNA2 сегментов этиъ же двухцепочных
вирусов возможны другие результаты.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ


























