Fasta-файл с открытыми рамками ЗДЕСЬ.
Получение списка координат и ориентаций найденных открытых рамок с помощью infoseq.
Команда следующая: infoseq coli.orf -only -name -sprotein1 -length -description > result. С её помощью был получен список координат и ориентаций найденных открытых рамок. Однако здесь присутствовало много лишней информации, которая была отфильтрована с помощью Excel. Результат: Таблица Excel.
Получение списка аннотированных генов белков.
Сперва был скачан файл с fasta-последовательностями белков: ССЫЛКА.
Далее была скчана хромосомная таблица со списком генов белков, которая была обработана в Excel и отсортирована по "from": Таблица Excel.
Далее обе таблицы анализировались в одном файле: Сравнение таблиц.
Расхождения представлены на Рис. 1.
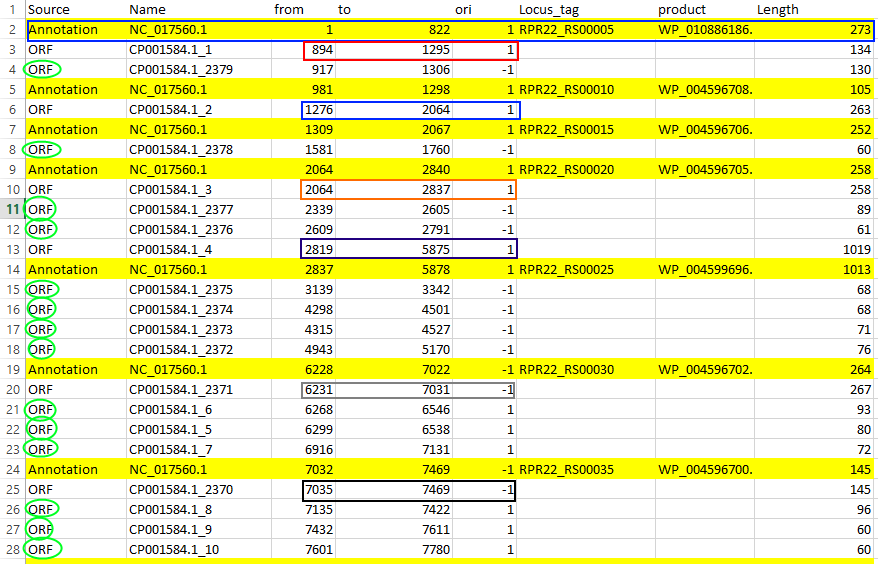
Сразу, в частности, можно заметить расхождение: для гена NC_017560.1 (выделен голубым цветом на Рис.1) отсутствует открытая рамка считывания. Это объясняется тем, что данный ген начинается сразу с 1 нуклеотида старт-кодоном и заканчивается стоп-кодоном. Так как getorf ищет участки между стоп-кодонами, то фрагмент хромосомы до первого стоп-кодона программа "проглядела".
Следующие за ним гены уже имеют каждый свою рамку считывания, которые выделены на Рисю.1 различными цветами. Однако ещё немаловажные отличия - каждый ген выходит за свою рамку считывания ровно на 3 нуклеотида (Рис.1). Это объясняется наличием стоп-кодонов в генах белков, которых нет в ORF. На Рис.1 представлены 6 таких открытых рамок.
Кроме того, помимо рамок, содержащих полный ген, существуют открытые рамки, в которых нет генов (12 таких рамок выделены зелёным на Рис.1). Такие рамки обычно короче (длина часто менее 100 остатков). Действительно, наличие открытой рамки является необходимым, но не достаточным условием существования гена на данном участке. Поэтому в геномах присутствие таких "пустых" орфов - обычное явление.
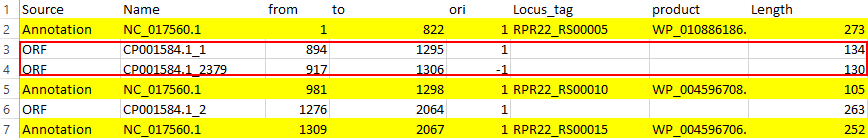
Так же можно найти гены белков, для которых нет орфов по причине их слишком маленькой длины (менее 60 остатков белок, так как мы задавали длину орфа не менее 180 п.н.) Один из таких примеров представлен на Рис.2.
На Рис.3 приведён пример перекрывания антипараллельных рамок размером 378 п.н. (гораздо больше, чем 150). На одна из этих рамок, расположенной на прямой цепи (CP001584.1_1), располагается ген NC_017560.1, который и транскрибируется с данной рамки. Другая рамка CP001584.1_2379 на комплементе, несмотря на то, что почти не устпает по длине CP001584.1_1), не содержит никакого гена. Действительно, при больших перекрываниях антипараллельных рамок, обычно ген есть лишь в одной из них, либо гена нет в обеих таких рамках: довольно сложно представить, чтобы гены нормально транскрибировались с обеих перекрывающихся рамок.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ