Главная
Практикум №12: "Предсказание генов эукариот"
Задание 1. Предскажите гены X5 с помощью AUGUSTUS.
Выбранный контиг из сборки X5: unplaced-888 c длиной 30534 пар нуклеотидов (больше 20 kb, но меньше 100 kb). Данный контиг был найден с помощью программы
infoseq (Рис. 1).
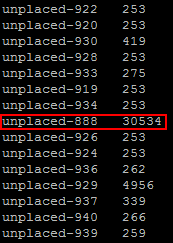 Рис. 1. Программа infoseq с опциями -only -name -snucleotide1 -length
Рис. 1. Программа infoseq с опциями -only -name -snucleotide1 -length
выдаёт название контига и его длину. Выбранный контиг выделен красным цветом.
С помощью программа seqret была получена нуклеотидная последовательность данного контига.
Предсказание генов X5 с помощью AUGUSTUS.
Для начала необходимо выбрать организм, наиболее близкий к какому-либо организму из списка, для которого уже произведено обучение сервиса AUGUSTUS.
Для этого был использован алгоритм blastx, который транслирует входную последовательность в шести рамках и ищет именно ПРОДУКТ гена.
На Рис. 2. представлены находки blast. Видно, что среди первых семи находок пять - из рода Schizosaccharomyces.
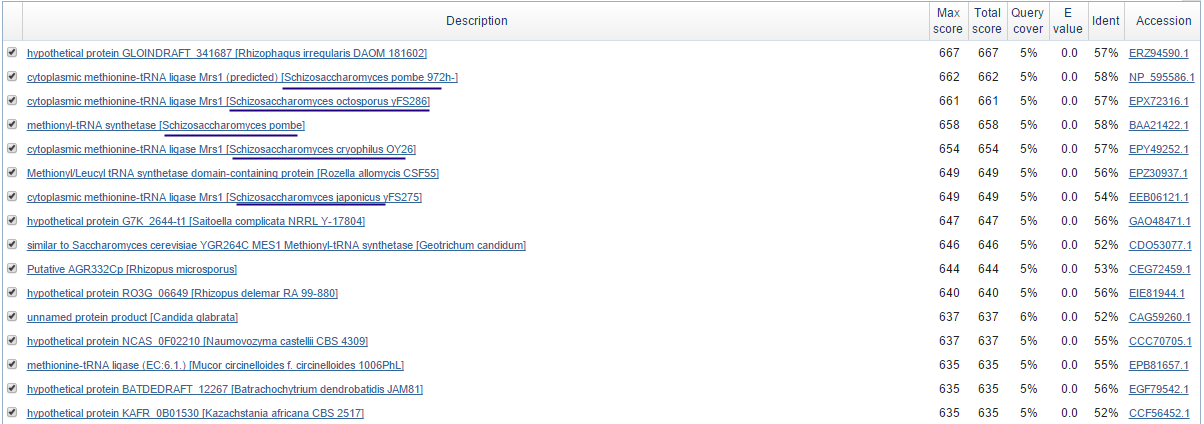 Рис. 2. Фрагмент выдачи blastx для входной последовательности unplaced-888.fasta
Рис. 2. Фрагмент выдачи blastx для входной последовательности unplaced-888.fasta
Находки рода Schizosaccharomyces подчёркнуты.
На основании полученных результатов для предсказания генов AUGUSTUS был выбран организм Schizosaccharomyces pombe, присутствующий в данном ресурсе.
Увидеть организмы, предлагаемые AUGUSTUS можно увидеть ЗДЕСЬ.
Ссылка на страницу с описанием статуса задания: ЗДЕСЬ.
В результате предсказания был получен архив: predictions.tar.gz. Данный архив содержит несколько файлов:
В файле augustus.aa находятся аминокислотные последовательности белков (fasta), продуктов предсказанных генов. Ссылка на файл:
augustus.aa.
В файле augustus.cdsexons находятся нуклеотидные последовательности экзонов (fasta), входящих в предсказанные гены. Ссылка на файл:
augustus.cdsexons.
В файле augustus.codingseq находятся кодирующие нуклеотидные последовательности генов (fasta). Ссылка на файл:
augustus.codingseq.
В файле augustus.gtf присутствует информация о предсказанных генах: координаты CDS, интронов, старт- и стоп-кодонов, цепь, координаты
инициаторной и терминальной последовательности гена. Ссылка на файл:
augustus.gtf.
В файле augustus.gbrowse присутствует информация о пре-мРНК синтезированных с предсказанных генов в процессе транскрипции.
Ссылка на файл:
augustus.gbrowse.
Файле augustus.gff суммирует информацию всех их всех упомянутых файлов.
Ссылка на файл:
augustus.gff.
Проверка предсказания с помощью BLAST.
Было предсказано 12 генов, кодирующих белки. Последовательности предсказанных белков в fasta формате содержатся в файле augustus.aa.
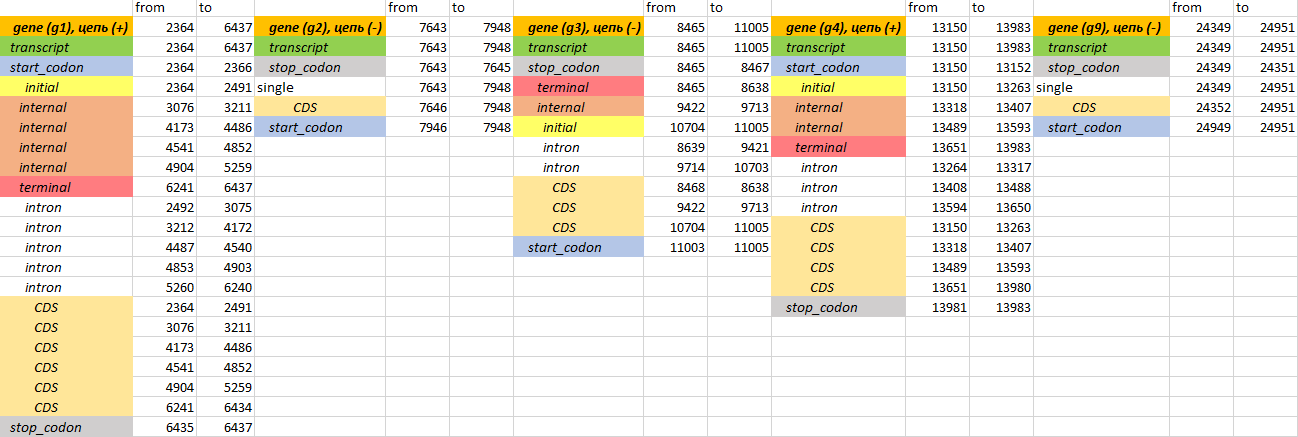
Далее пять генов (g1, g2, g3, g4 и g9) были выбраны для проверки с помощью BLAST. Предсказанная структура данных генов была взята из файла
augustus.gff и обработана в Excel (Таблица 1).
Таблица 1. Предсказание структуры генов g1, g2, g3, g4, g9 с помощью AUGUSTUS.
Далее для аминокислотных последовательностей предсказанных белков из файла augustus.aa проводился поиск гомологов с помощью blastp по
БД Swissprot. Область поиска была ограничена таксоном Fungi.
Самый длинный белок - продукт предсказанного гена g1. Для него blastp нашёл при данных условиях поиска 3 сравительно хорошие находки с
значением score более 200, покрытием более 70% и идентичностью более 40% (Рис. 3).
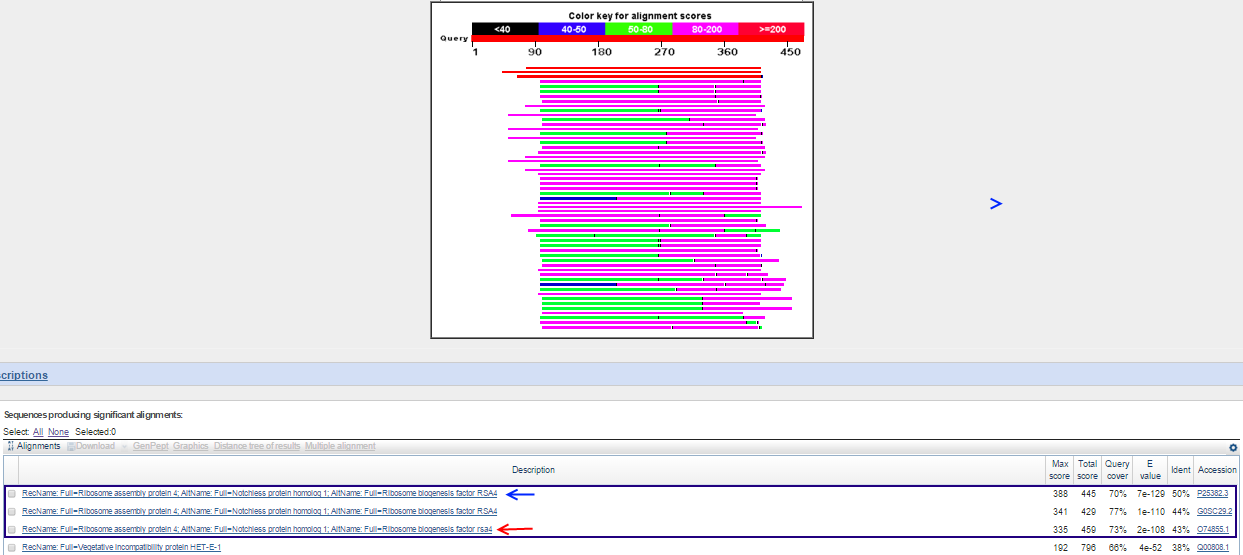 Рис. 3. Выдача blastp для белка g1. Хорошие находки выделены синей рамкой.
Рис. 3. Выдача blastp для белка g1. Хорошие находки выделены синей рамкой.
Так же стоит заметить, что одна из данных находок как раз принадлежит виду Schizosaccharomyces pombe, а этот организм как раз и был выбран для предсказания
генов данного контига (красная стрелка на Рис. 3. и Рис. 4.). Однако данный белок был предсказан по гомологии, что вызывает сомнения в достоверности
находки
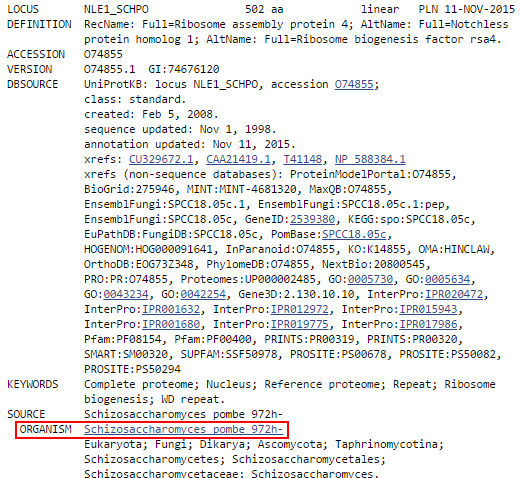 Рис. 4.
Рис. 4.
Так же очень важна первая, лучшая находка, принадлежащая Saccharomyces cerevisiae (синяя стрелка на Рис.3), так как данный продукт подтверждён на уровне белка экспериментально
(Рис. 5).
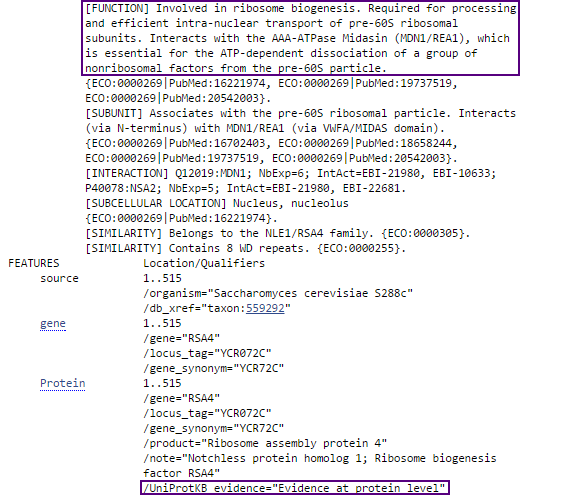 Рис. 5. Находка, принадлежащая Saccharomyces cerevisiae.
Рис. 5. Находка, принадлежащая Saccharomyces cerevisiae.
Можно предположить, что именно этот белок и является наиболее вероятным гомологом изучаемого продукта предсказанного гена g1. Данный белок представляет
собой белок сборки рибосомы 4 (RSA 4), информация о функции которого представлена на Рис.5. Он участвует в биогенезе рибосомы, необходим для
процессинга и эффективного транспорта предшественников 60S субъединиц рибосомы в ядро.
Попытаемся разобраться с экзон-интронной структурой, предсказанной AUGUSTUS. Можно заметить, что во всех трёх лучших находках есть две значительные области,
в которых во входной последовательности стоят гэпы, причём во всех трёх выравниваниях приблизительно в одних и тех же позициях: первая область -
гэп районе 193-196 остатков query длиной 20 остатков, вторая область с гэпами начинается приблизительно с 290-295 остатков длиной 26 остатков: в находке,
прнадлежащей Chaetomium thermophilum (вторая в выдаче blast), в данной области один гэп, в двух других данный регион разбит на два гэпа (Рис. 6a-6c).
 Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Saccharomyces cerevisiae
Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Saccharomyces cerevisiae
Области с вероятными ошибками в предсказанной экзон-интронной структурой выделены оранжевым цветом.
 Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Chaetomium thermophilum
Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Chaetomium thermophilum
Области с вероятными ошибками в предсказанной экзон-интронной структурой выделены оранжевым цветом.
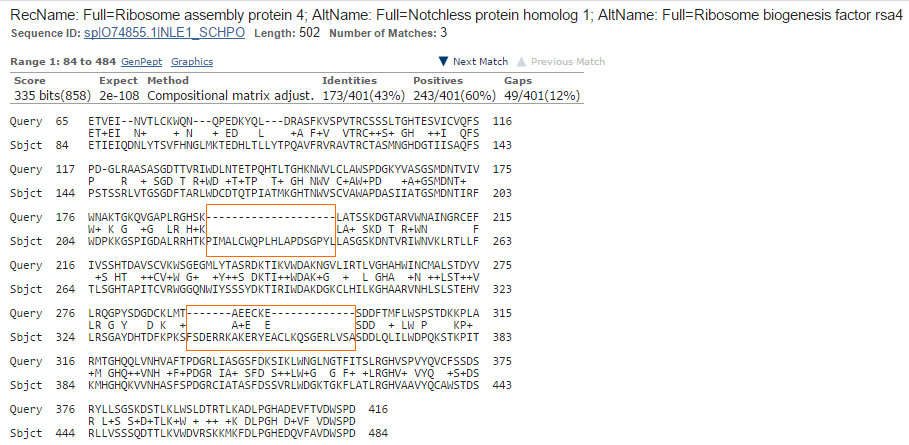 Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Schizosaccharomyces pombe
Рис. 6a. Выравнивания белка - продукта гена g1 с RSA 4 из Schizosaccharomyces pombe
Области с вероятными ошибками в предсказанной экзон-интронной структурой выделены оранжевым цветом.
Возможно, данные гэпы в последоватнльности белка, продукта гена g1, связаны с тем, что AUGUSTUS распознал часть экзона как интрон, вследствие чего
данный участок не был транслирован, что привело к образованию гэпа в данной области.
Следовательно, имеют место ошибки AUGUSTUS в определении экзон-интронной структуры.
Далее на вход blastp была подана последовательность белка - продукта гена g2. Не было найдено ни одной находки (Рис. 7).
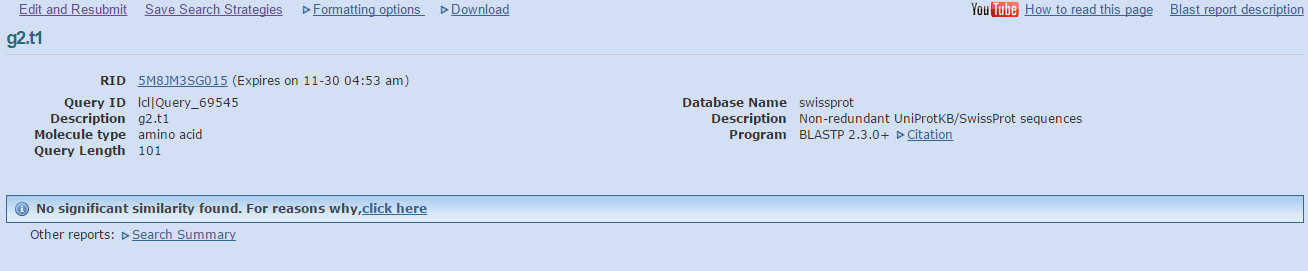 Рис. 7. Выдача blastp: отсутствие находок.
Рис. 7. Выдача blastp: отсутствие находок.
Таким образом, предсказание гена g2 - ошибка AUGUSTUS.
Далее проверим белок - продукт гена g3. Про поиске в банке nr (область поиска ограничена таксоном Fungi) blastp выдал серию гипотетических
белков и белков dopey (Рис. 8), причём лишь одна находка имела
query cover более 50% (54%). Значения Score так же очень низкие (менее 80). И лишь с большой натяжкой можно предположить, что рассматриваемый белок в самом деле
принадлежит к суперсемейству Dopey_N: тогда гораздо короче: состаляет приблизительно 1/3 длины белка, предсказанного AUGUSTUS (Рис. 9).
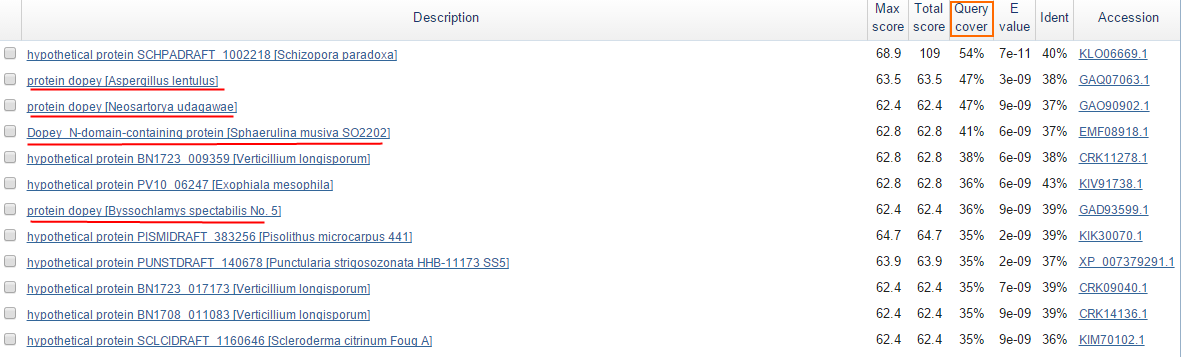 Рис. 8. Выдача blastp для продукта гена g3. Находки отсортированы по query cover. Белки из суперсемейства Dopey-N подчёркнуты красным.
Рис. 8. Выдача blastp для продукта гена g3. Находки отсортированы по query cover. Белки из суперсемейства Dopey-N подчёркнуты красным.
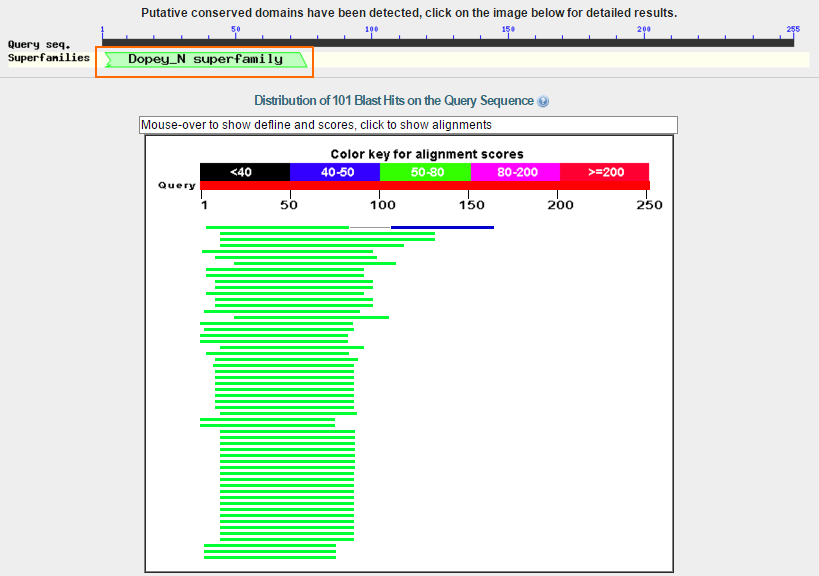 Рис. 9. Иллюстрирует предположение возможной принадлежности белка к суперсемейству Dopey_N.
Рис. 9. Иллюстрирует предположение возможной принадлежности белка к суперсемейству Dopey_N.
Чтобы это проверить, был проведён поиск по БД Swissprot для нахождения более достоверных гомологов. Снова область поиска была ограничена таксоном
Fungi. Blastp нашёл всего две находки (Рис.10). У одной E-value 8.1 - даже не рассматриваем, а вот другая (подчёркнута на Рис. 10) как раз представляет
собой белок dopey из Aspergillus nidulans, подтверждённый экспериментально на уровне белка, что очень важно (Рис. 11).
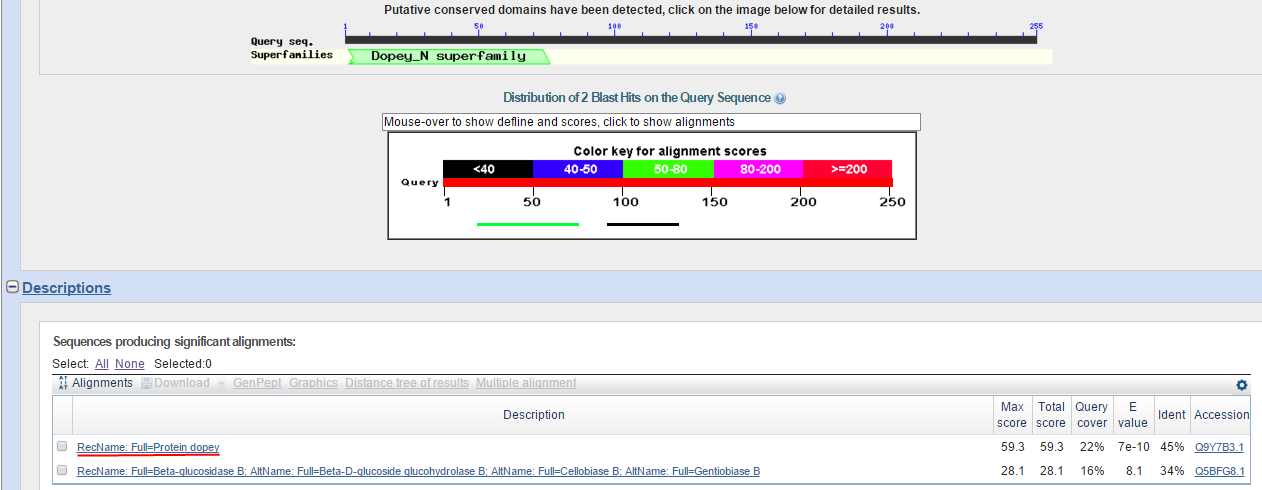 Рис. 10. Выдача blastp для банка Swissprot для продукта гена g3.
Рис. 10. Выдача blastp для банка Swissprot для продукта гена g3.
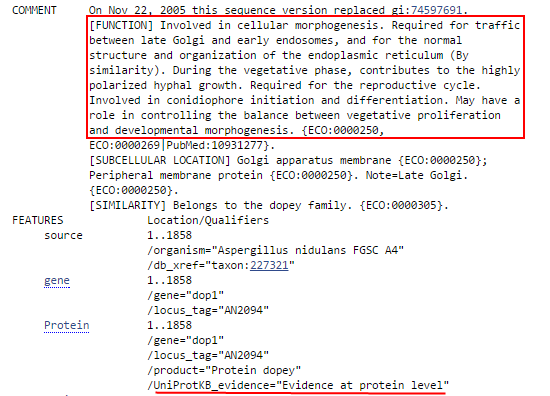 Рис. 11. Функция белка Protein dopey из Aspergillus nidulans.
Рис. 11. Функция белка Protein dopey из Aspergillus nidulans.
Функция данного белка: Вовлечён в клеточный морфогенез, необходим для нормальной организации и функционирования ЭПР, аппарата Гольджи, формирования эндосом.
На основании этих фактов можно сделать вывод, что действительно предсказанный ген - ген, кодирующий белок семейства Dopey_N, однако, видимо,
AUGUSTUS сильно ошибся в определении границ гена и экзон-интронной структуры: на самом деле белок в три раза короче предсказанного ресурсом.
Достоверных гомологов нет: домен Dopey_N далеко не высококонсервативный (небольшие значения Score, E-value).
Аналогично проверялся продукт белка g4 (Банк nr, ограничен поиск по Fungi, Рис. 12). Blast выдал всего 9 находок, все они плохие, к тому же не
было обнаружено никаких консервативных доменом вообще. Вывод: Предсказание ошибочно.
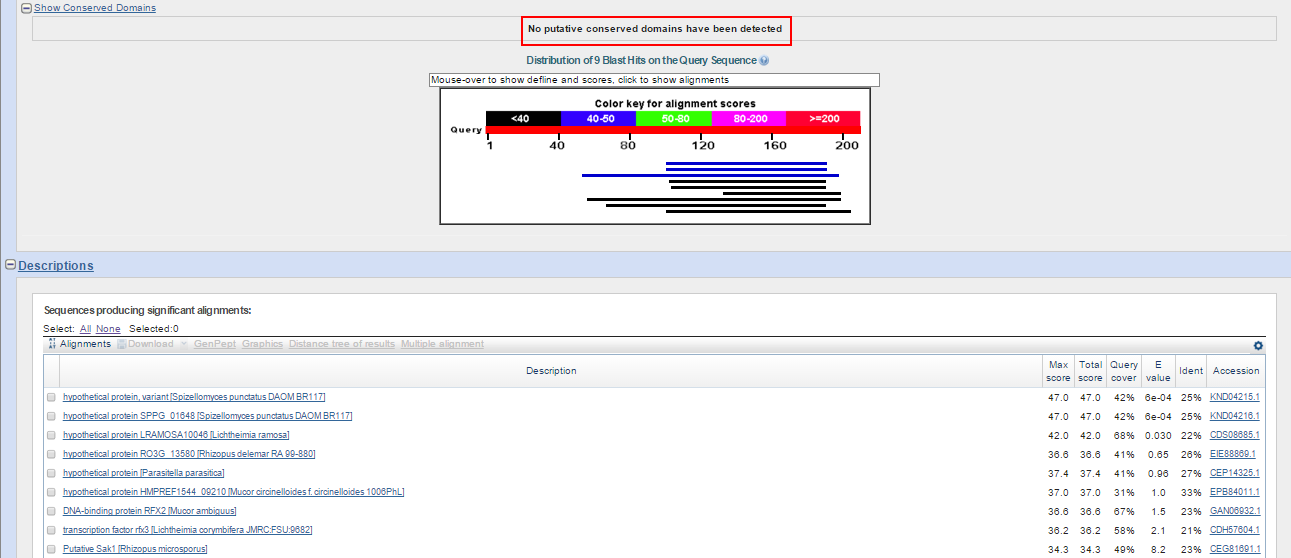 Рис. 12. Выдача blastp для последовательности белка - продукта гена g4.
Рис. 12. Выдача blastp для последовательности белка - продукта гена g4.
Проверка g9. При поиске в банке nr (поиск ограничен по Fungi, Рис. 13) blastp обнаружил высоко консервативный домен по ВСЕЙ
длине входной последовательности.
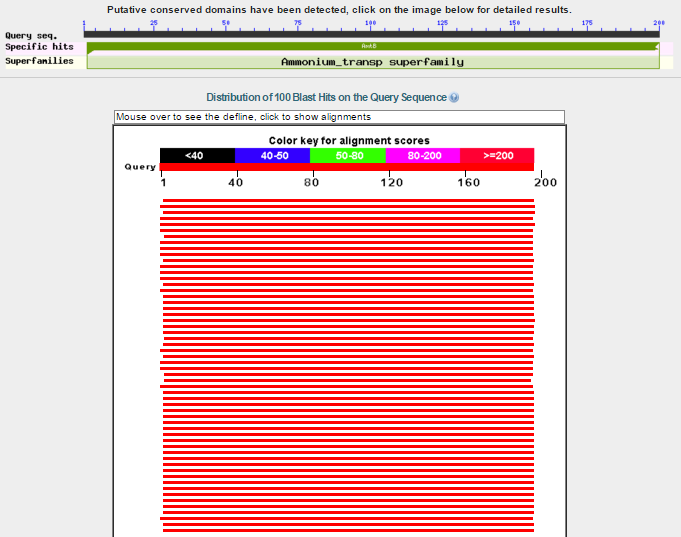 Рис. 13. Выдача blastp для последовательности белка - продукта гена g9.
Рис. 13. Выдача blastp для последовательности белка - продукта гена g9.
Далее гомологи я искал по базе Swissprot (Рис. 14).
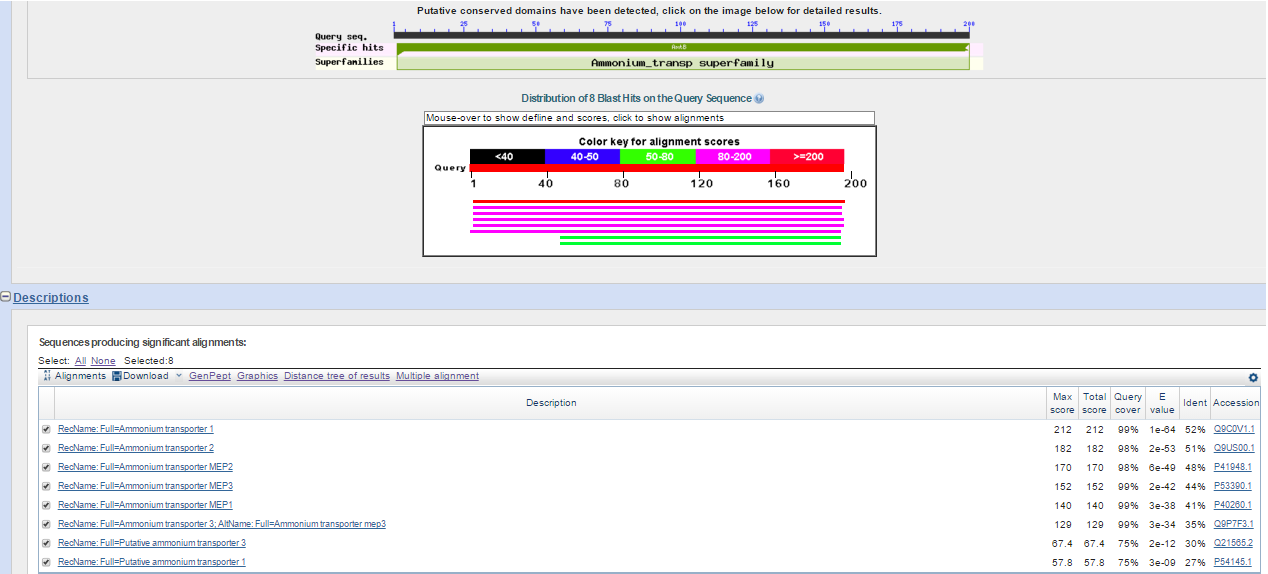 Рис. 14. Поиск гомологов продукта гена g9 по Swissprot.
Рис. 14. Поиск гомологов продукта гена g9 по Swissprot.
Было найдено 9 неплохих находок с очень хорошим query cover.
Причём лучшей находка со значением
Score за 200 представляет собой последовательность белка Ammonium transporter 1 из Schizosaccharomyces pombe (этот организм и был использован для поиска
гомологов нашего контига)!
Функция этого белка: транспортировка аммония для его использования в качестве источника азота. В условиях недостатка аммония способствует образованию
псевдогиф (Рис. 15).
Данную последовательность с большой долей вероятности можно считать гомологом входной последовательности, существуют лишь некоторые сомнения в достоверности,
так как данный белок подтверждён не экспериментально, а на уровне гомологии (Рис. 15).
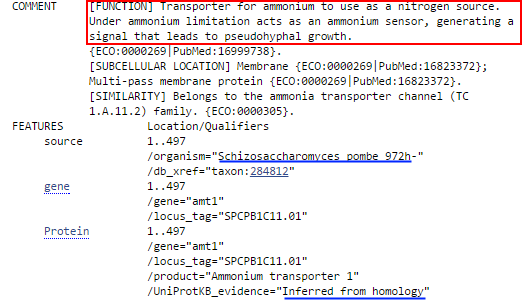 Рис. 14. Ammonium transporter 1 из Schizosaccharomyces pombe.
Рис. 14. Ammonium transporter 1 из Schizosaccharomyces pombe.
Задание 2. Сравнение аннотации Refseq и AUGUSTUS одного гена человека.
Выбранный ген: CCND1 (cyclin D1, Gene ID: 595).
Продукт данного гена, циклин D1, является регулятором циклин-зависимыми киназами CDK4 и CDK6, активируя их. Далее комплекс циклин D1 / CDK4 или
циклин D1 / CDK6 активирует (фосфорилирует) белок ретинобластомы Rb (опухолевый супрессор),
что способствует прогрессии клеточного цикла и переходу из G1 в S-фазу.
Координаты гена: хромосома 11; NCBI: 69641105-69654474, длина 13370 п.н. (UCSC Genome Browser: 69641087-69654474, длина 13388 п.н.); прямая цепь.
Для выполнения задания использовался UCSC Genome Browser. Была выбрана последняя сборка генома человека hg38.
Далее был найден выбранный ген CCND1
(поиском в Genome Browser). Так же были оставлены только три трэка: base position, Refseq и AUGUSTUS. В результате браузер выдал аннотации Refseq и AUGUSTUS гена (Рис. 15).
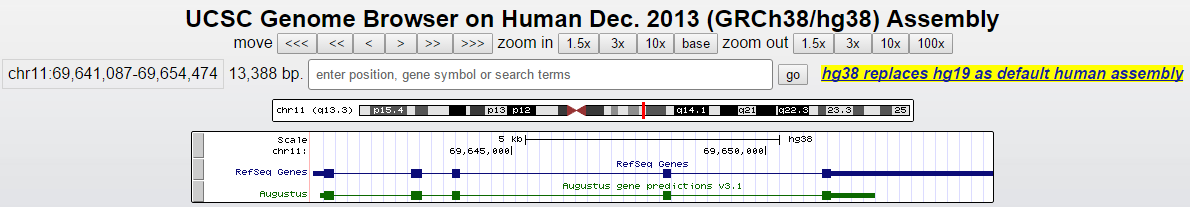 Рис. 15.
Рис. 15.
Далее были получены экзон-интронные структуры, предсказанные Refseq и AUGUSTUS.
В обоих предсказаниях координаты CDS: 69641313-69651282 и в обоих предсказано по 5 экзонов и по
4 интрона. Важно, что, так как первый и последний экзоны содержат 5'-и 3'-нетранслируемые
области, при вычислении остатка от деления на 3 использовались координаты CDS. Экзон-интронная структура, предсказанная Refseq и AUGUSTUS, представлена в Таблице 2.
Таблица 2. Экзон-интронная структура, предсказанная Refseq и AUGUSTUS. Для терминальных экзонов длина кодирующей обрасти (без UTR) показана в скобках.
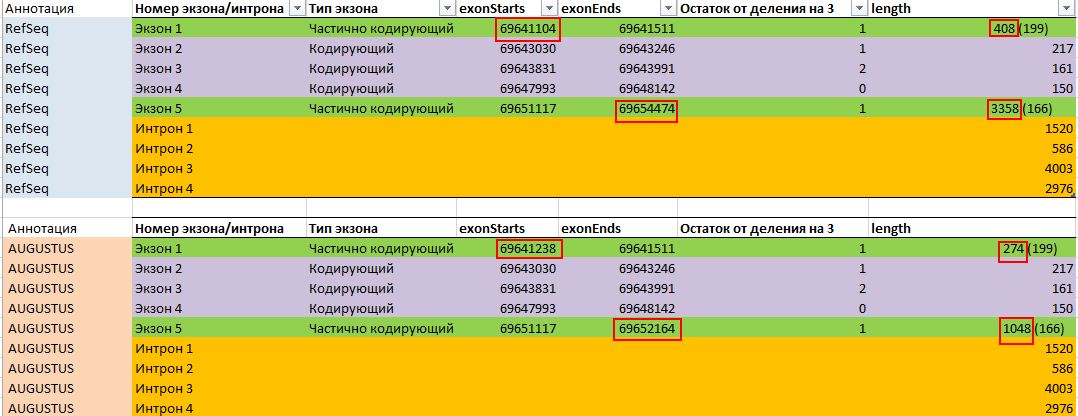
В данных предсказаниях все совпадает, кроме координаты начала первого экзона и координаты
конца последнего (отличия выделены в Таблице 2 красными прямоугольками). Видно, что и первый экзон, и последний
значительнее длиннее в аннотации Refseq, что позволяет сделать вывод, что Refseq выявил более длинные
5'-и 3'-нетранслируемые области, чем AUGUSTUS.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ



















