Главная
Практикум №11: "Поиск сигналов. Продолжение."
Задание 3. Определение сайтов связывания данного транскрипционного фактора в данном участке хромосомы человека
В данном задании я работал с файлом chipseq_chunk46.fastq.
К сожалению, качество всех чтений в данном файле было не очень хорошим, под 28, я решил взять для работы резервный файл chipseq_chunk46.fastq.
В данном файле качество ридов хорошее (Рис. 1.) и фильтрация данных не нужна. Всего последовательностей в этом файле 6543, длина 36 нуклеотидов.
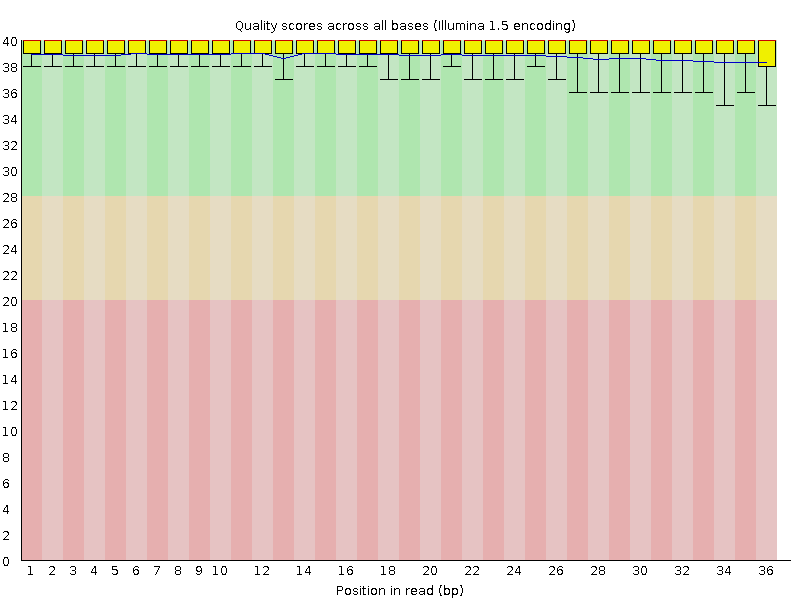 Рис. 1. Per base quality для chipseq_chunk7.fastq
Рис. 1. Per base quality для chipseq_chunk7.fastq
Далее чтения были картированы на заранее проиндексированный геном человека hg19. Команда:
bwa mem ../hg19/GRCh37.p13.genome.fa chipseq_chunk7.fastq > chipseq_chunk7.sam
Далее использовались команды:
samtools view -bSo chipseq_chunk7.bam chipseq_chunk7.sam - перевод выравнивание чтений с референсjv в бинарный формат.
samtools sort chipseq_chunk7.bam -T chip_temp -o chipseq_chunk7.sorted.bam - сортировка выравнивание по координате начала чтения в референсе.
samtools index chipseq_chunk7.sorted.bam - индексация полученного файла.
samtools idxstats chipseq_chunk7.sorted.bam > chipseq_chunk7.idxstats - запись информации о количестве откартированных чтений.
samtools view -c chipseq_chunk7.sorted.bam - информация о числе чтений в сумме откартированных на все элементы генома.
Все 6543 чтения были откартированы на геном (Рис. 2).
 Рис. 2. Число чтений, картированных на геном.
Рис. 2. Число чтений, картированных на геном.
Распределение чтений по хромосомам (Рис. 3., инофрмация из файла chipseq_chunk7.idxstats)
позволяет предположить, что у меня хромосома 1.
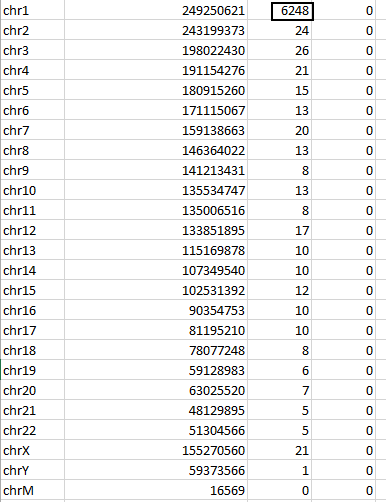 Рис. 3. Распределение чтений по хромосомам. Наибольшее число чтений (6248) картировалось
на хромосому 1 (чёрный прямоугольник).
Рис. 3. Распределение чтений по хромосомам. Наибольшее число чтений (6248) картировалось
на хромосому 1 (чёрный прямоугольник).
Поиск пиков с помощью программы MACS. Так как пиков было очень мало, команда:
macs2 callpeak -t chipseq_chunk7.sorted.bam -n chipseq_chunk7 --nomodel.
Результат - 3 файла:
chipseq_chunk7_peaks.narrowPeak
chipseq_chunk7_peaks.xls
chipseq_chunk7_summits.bed
Программа нашла 9 пиков в первой хромосоме примерно в одной области - около 59 млн п.н. Ширина пиков варьируется от 202 до 478 нуклеотидов.
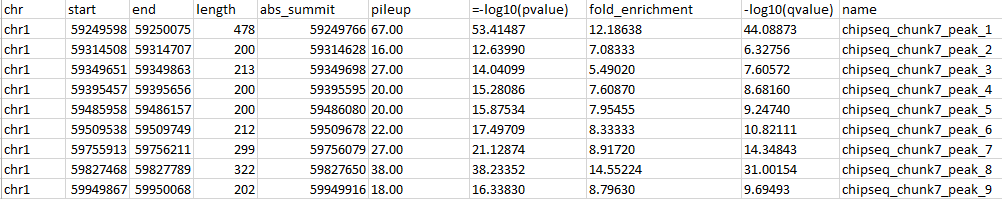 Таблица 1. Пики, найденные программой MACS.
Таблица 1. Пики, найденные программой MACS.
Результаты были визуализированы с помощью UCSC Genome Browser.
На Рис. 4. представлен скриншот окна геномного браузера, где в мелком масштабе отображены все найденные пики.
 Рис. 4.
Рис. 4.
Достоверность пиков
Значение p-value характеризует достоверность эксперимента: если его значение ниже порогового, что результат можно считать достоверным.
q-value аналогия p-value, но для множественных экспериментов. То есть тоже чем меньше q-value, тем достовернее результат. Но у нас в таблицах отрицательные
десятичные логарифмы p-value и q-value, соответственно, чем их значения больше, тем доставернее результат.
Из таблицы 1 следует, что наиболее достоверными следует считать пики 1 и 8. Для них наибольшие значения отрицательных логарифмов p- и q-value
(53.41487 и 44.08873 для первого пика и 38.23352 и 31.00154 для восьмого). Наименее достоверный пик (с наименьшими этими значениями) - второй
(12.63990 и 6.32756, соответственно).
Ширина пиков
Из таблицы 1 видно, что ширина пиков варьируется от 202 до 478 нуклеотидов. Вершина большинства пиков расположена приблизительно посередине пика с
небольшими отклонениями в ту или другую сторону,
но не всегда: например, у третьего и девятого пиков вершина расположена в его первой четверти
(расстояние от старта до вершины составляет 22% и 24% от общей ширины пика, Таблица 2).
 Таблица 2. Расположение вершины пиков относительно его крайних точек.
Здесь первая строка соответствует первому пику, вторая - второму и т.д
Таблица 2. Расположение вершины пиков относительно его крайних точек.
Здесь первая строка соответствует первому пику, вторая - второму и т.д
Расположение относительно функциональных элементов генома
Пик 1 расположен в конце гена JUN (Homo sapiens Jun proto-oncogene, AP-1 transcription factor subunit). Так же я включил
отображение регуляторных элементов (ENCODE project). Интересно, что как раз в области пика значительно снижена модификация H3K27Ac, которая в целом
активирует экспрессию генов
Видно, что в области 1 пика находятся сайты связывания нескольких регуляторных факторов, найденные с помощью ChIP-seq (Рис. 5).
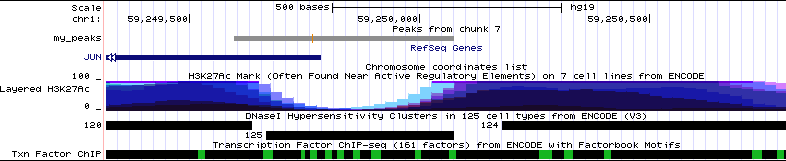 Рис. 5. Расположение первого пика в геноме.
Рис. 5. Расположение первого пика в геноме.
Пик 2 расположен в интроне длинной некодирующей РНК LINC01135. В его области также находится некоторое количество сайтов связывания ргуляторных
элементов. И в его области также H3K27Ac меньше, чем в прилежащих областях (Рис. 6).
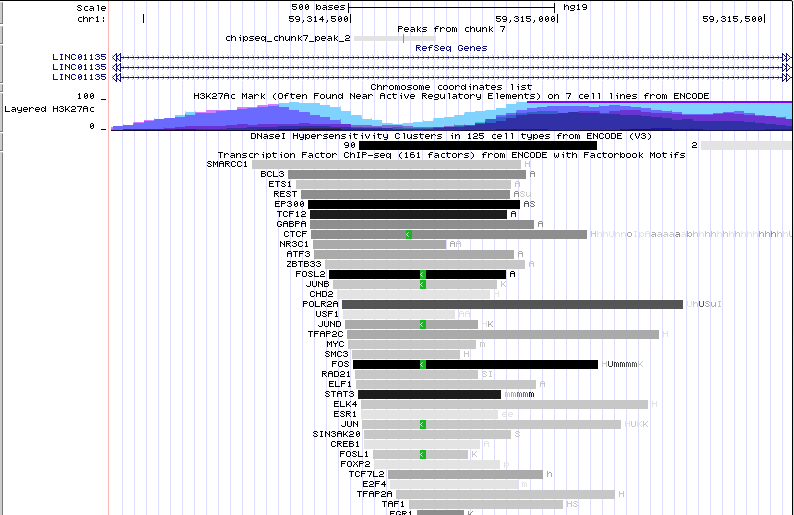 Рис. 6. Расположение второго пика в геноме.
Рис. 6. Расположение второго пика в геноме.
Пик 3 также расположен в длиной некодирующей РНК LINC01135. Модификации H3K27Ac не снижена в области пика, в отличие от предыдущих случаев.
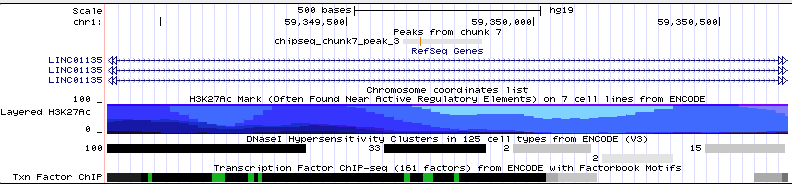 Рис. 7. Расположение третьего пика в геноме.
Рис. 7. Расположение третьего пика в геноме.
Пик 4 расположен уже вне гена LINC01135, на расстоянии около 90 тысяч п. н. до следующего гена другой длинной некодирующей РНК (LINC01358).
Снова модификации H3K27Ac в области пика меньше, много сайтов связывания различных регуляторных элементов (например, JUN, FOSL1, EGR1 и другие)
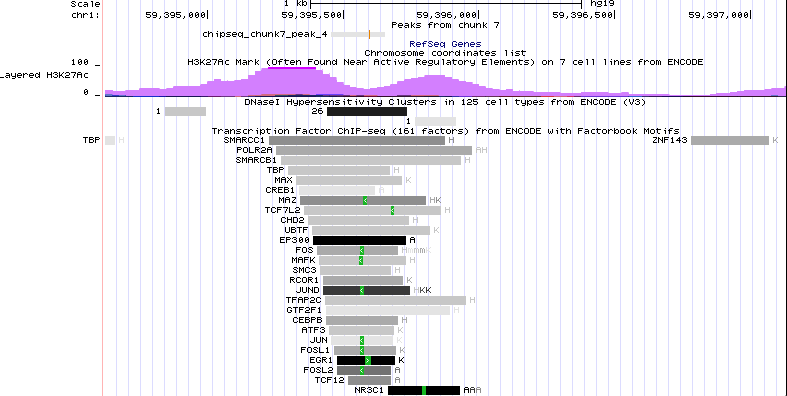 Рис. 8. Расположение четвёртого пика в геноме.
Рис. 8. Расположение четвёртого пика в геноме.
Пик 5 расположен перед геном LINC01358. Снова модификации H3K27Ac в области пика меньше, чем в его окрестности, много сайтов связывания регуляторных
элементов, например, сайты связывания транскрипционных фактором TCF12 и TCF3 попадают прямо в област пика.
 Рис. 9. Расположение пятого пика в геноме.
Рис. 9. Расположение пятого пика в геноме.
Пик 6 расположен уже в гене LINC01358. Модификация H3K27Ac в данной области не так выражена, как в предыдущих случаях. Есть несколько сайтов связывания,
различных транскрипционных факторов
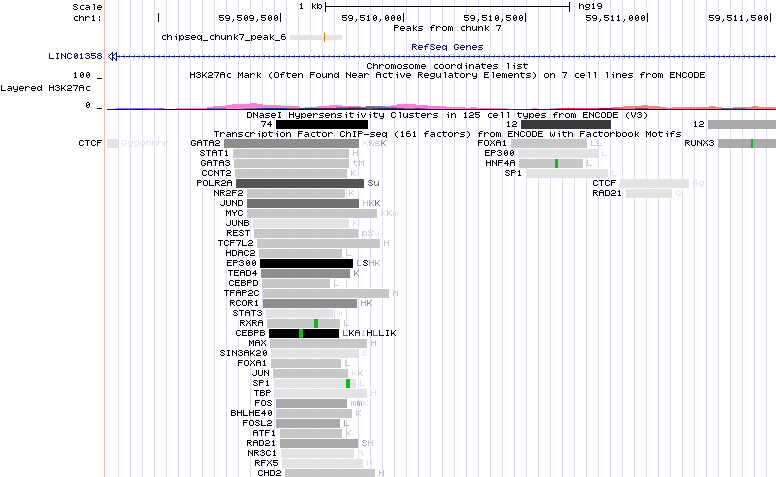 Рис. 10. Расположение шестого пика в геноме.
Рис. 10. Расположение шестого пика в геноме.
В области пика 7 также содержится несколько сайтов связывания регуляторных элементов (CTCF, JUND, например). Модификации третьего гистона здесь
практически нет. Расположен перед геном карбогидраткиназы (FGGY).
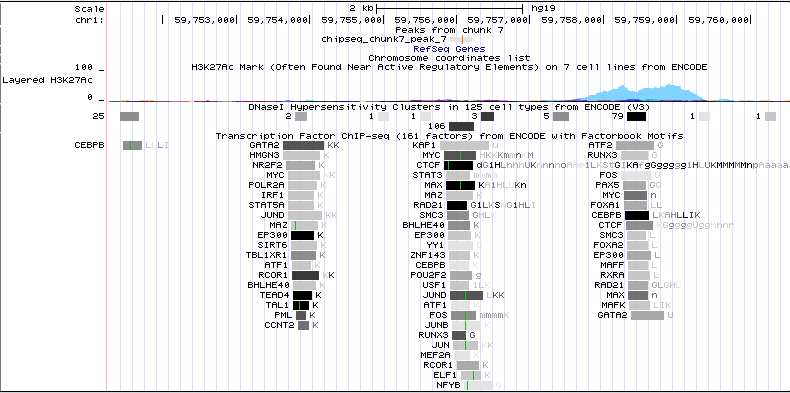 Рис. 11. Расположение седьмого пика в геноме.
Рис. 11. Расположение седьмого пика в геноме.
В области пика 8 содержится довольно много сайтов связывания регуляторных элементов (JUN, FOS, JUND, ATF1). Так же расположен в гене FGGY.
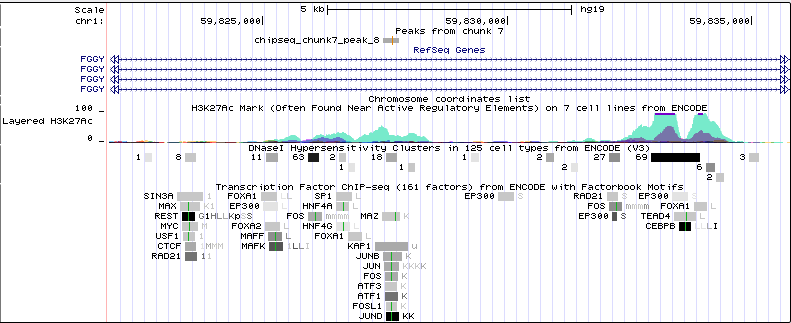 Рис. 12. Расположение восьмого пика в геноме.
Рис. 12. Расположение восьмого пика в геноме.
В области пика 9 содержится довольно много сайтов связывания регуляторных элементов (JUN, FOS, JUND). Так же расположен в гене FGGY.
 Рис. 13. Расположение девятого пика в геноме.
Рис. 13. Расположение девятого пика в геноме.
Задание 4. Нахождение трёх генов, транскрипция которых инициируется с помощью TATA-бокс связывающего белка, и одного -
без сигнала TATA-бокса в промоторной области
TATA-бокс связывающий фактор TBP - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК с консенсусом TATAWAAR.
Он является одним из ключевых ДНК-узнающих белков при образовании на промоторе генов комплекса TFIID инициации
транскрипции с помощью Pol II. Тем не менее, лишь часть промоторов имеет сигнал TATA-box, связываемый TBP.
Описание выбранного эксперимента представлены на Рис. 14. Взята клеточная линия HeLa-S3.
 Рис. 14. Параметры выбранного эксперимента.
Рис. 14. Параметры выбранного эксперимента.
Ген G6PC3
Название: Homo sapiens glucose 6 phosphatase catalytic subunit 3
17 хромосома, старт: 42148098, стоп: 42153712, прямая цепь, длина 1637 п.н., последовательность TATAAA расположена на расстоянии приблизительно
20 нуклеотидов от старта транскрипции (Рис. 15, Рис. 16).
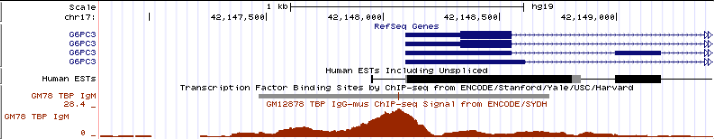 Рис. 15. Промоторная область гена G6PC3 в мелком масштабе. Виден пик (немного пологий правда).
Рис. 15. Промоторная область гена G6PC3 в мелком масштабе. Виден пик (немного пологий правда).
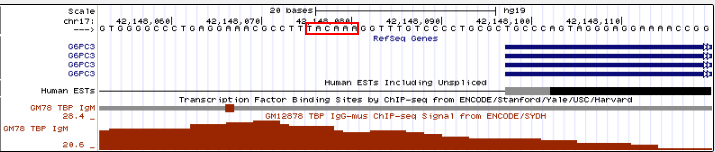 Рис. 16. Промоторная область гена G6PC3 в увеличенном масштабе. Красным выделена
последовательность TATA-бокса.
Рис. 16. Промоторная область гена G6PC3 в увеличенном масштабе. Красным выделена
последовательность TATA-бокса.
Пик отчётливо выделяется на уровне общего шума, в промоторной области
в 16 нуклеотидах от старта транскрипции была найдена TACAAA. ТАТА-бокс очень сходен с консенсусом, что позволяет предположить, что промотор сильный.
Ген ATAD5
Название: Homo sapiens ATPase family, AAA domain containing 5
17 хромосома, старт: 29158988, стоп: 29222883, прямая цепь, длина 6865 п.н. Рис. 17, Рис. 18.
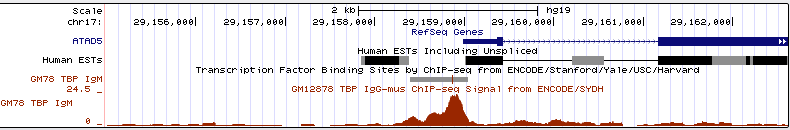 Рис. 17. Промоторная область гена ATAD5
в мелком масштабе. Виден отчётливый пик.
Рис. 17. Промоторная область гена ATAD5
в мелком масштабе. Виден отчётливый пик.
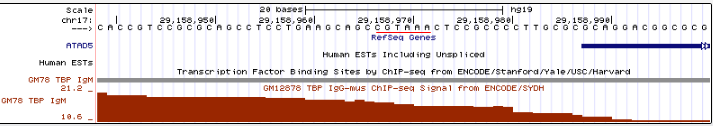 Рис. 18. Промоторная область гена ATAD5 в увеличенном масштабе. Явной последовательности
ТАТА-бокса, близкой к консенсусу нет.
Рис. 18. Промоторная область гена ATAD5 в увеличенном масштабе. Явной последовательности
ТАТА-бокса, близкой к консенсусу нет.
Пик связывания антител в промоторной области для данного гена очень высокий, гораздо выше уровеня шума. Это говорит о его высокой достоверности.
Однако последовательности TATA-бокса в промоторном регионе, видимо, нет. Есть последовательность (выделена красным на Рис.18) CGTAAA, 4 основаниями
схожая с консенсусной, но всё же её вряд ли
можно отнести к последовательности TATA-бокса. Поэтому для этого гена, по-видимому, ТАТА-бокса в промоторной области нет.
Ген ITPR1
Homo sapiens inositol 1,4,5-trisphosphate receptor type 1
3 хромосома, старт: 4535032, стоп: 4889524, прямая цепь, длина 10053 п.н. Рис. 19, Рис. 20.
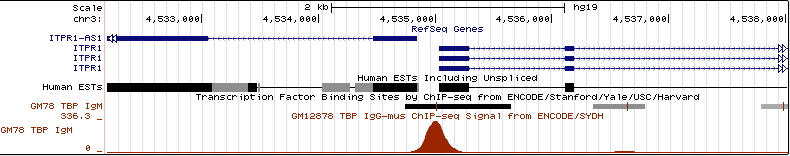 Рис. 19. Промоторная область гена ITPR1
в мелком масштабе. Виден отчётливый пик.
Рис. 19. Промоторная область гена ITPR1
в мелком масштабе. Виден отчётливый пик.
 Рис. 20. Промоторная область гена ITPR1 в увеличенном масштабе. Последовательность
ТАТА-бокса выделена красным
Рис. 20. Промоторная область гена ITPR1 в увеличенном масштабе. Последовательность
ТАТА-бокса выделена красным
Высокий пик, что говорит о высокой достоверности, присутствует последовательность TATA-бакса (TATATA), близкая к консенсусу. Расположена примерно
в 20 нуклеотидах от старта транскрипции.
Ген TPI1
triosephosphate isomerase isoform 1
12 хромосома, старт: 6976693 , стоп: 6980110, прямая цепь, длина 1366 п.н. Рис. 21, Рис. 22.
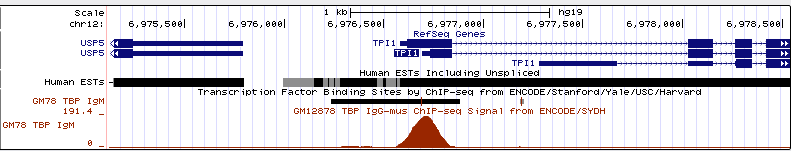 Рис. 20. Промоторная область гена TPI1
в мелком масштабе. Виден отчётливый пик.
Рис. 20. Промоторная область гена TPI1
в мелком масштабе. Виден отчётливый пик.
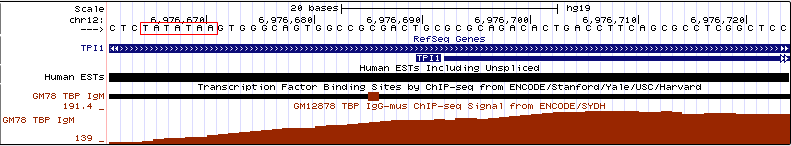 Рис. 21. Промоторная область гена TPI1 в увеличенном масштабе. Последовательность
ТАТА-бокса выделена красным
Рис. 21. Промоторная область гена TPI1 в увеличенном масштабе. Последовательность
ТАТА-бокса выделена красным
Виден сильный сигнал, позволяющий предполагать, что есть высоко консервативный ТАТА бокс. Так же из обзора я узнал, что, действительно,
в последовательности данного гена есть ТАТА бокс: ТАТАТАА. И именно этот мотив был найден перед стартом транскпипции 1 изоформы (вторая имеет
другое направление и перекрывается с первой) TPI1. Данная последовательность расположена на расстоянии 15 нуклеотидов от старта транскрипции, что согласуется
с дитературными данными






















