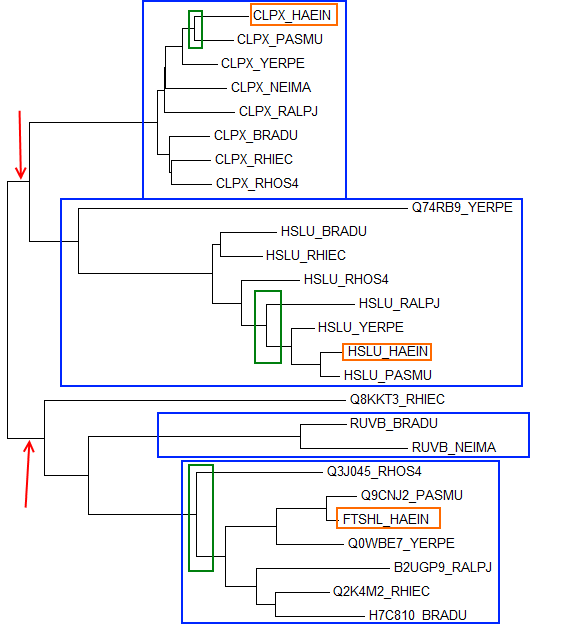
Далее был проанализирован полученный файл . Бласт выдал 34 находки, среди которых стоит отметить, что во всех 8 бактериях был найден CLPX(ATP-dependent Clp protease ATP-binding subunit ClpX) в единичном экземпляре.
По две находки для каждой бактерии (кроме NEIMA) было найдено HSLU (ATP-dependent protease ATPase subunit HslU).
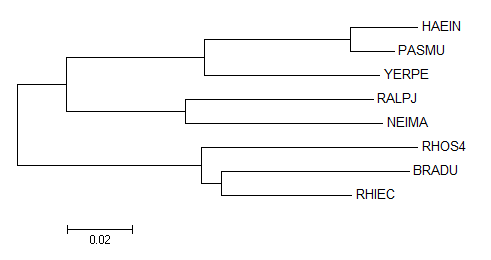
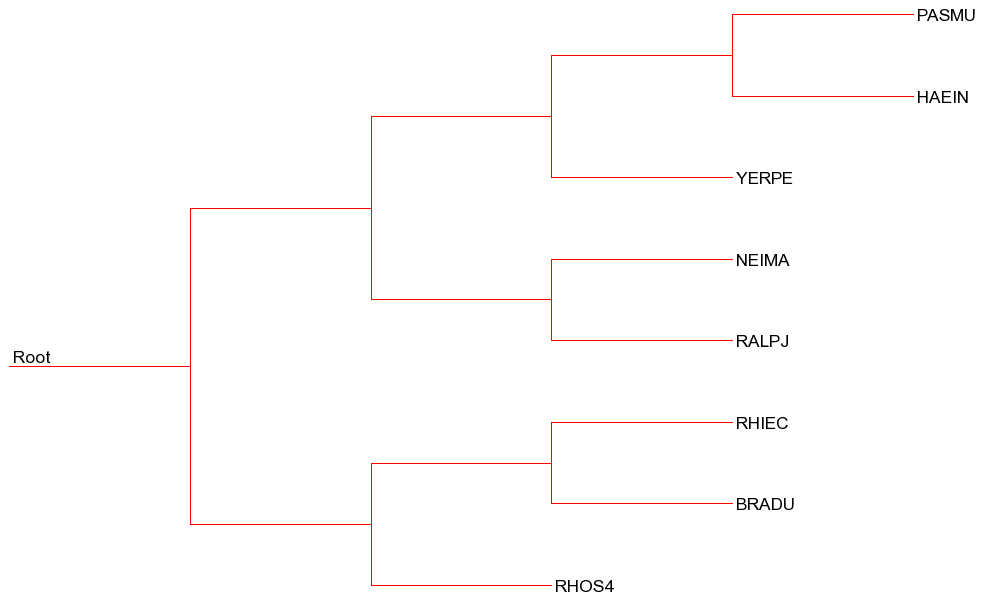
Далее для данных последовательностей гомологов было построено выравнивание (Muscle), импортировано в MEGA и там уже поспроено дерево (Neighbor-Joining, Рис. 3).