Главная
Практикум №6: "Мембранные белки"
Задание 1. Анализ множественного выравнивания трансмембранных белков
Белок NAD(P) трансгидрогеназа. PDB: 4O9U (цепь D), Uniprot: Q72GS0. Данному АС Uniprot (который был найден по
PDB) соответствует аминокислотная последовательность лишь бета-субъединицы (цепь B) (последовательности дельта, которая нужна по заданию в uniprot не оказалось).
Поэтому я рискнул дальше работать не с дельта, а с бета-субъединицей данного белка... Последовательность: здесь.
Для белка была получена репрезентативная выборка гомологов с помощью поиска BLAST на сайте EMBL EBI по кластерам UniRef50.
Выравнивание получалось очень плохое при запуске BLAST при указанных в задании параметрах (очень много плохо выровненных последовательностей),
поэтому я изменил порог E-value (1e-50), и уменьшил число находок до 150.
Репрезентативная выборка содержит 149 гомологов, E-value худшей находки: 1.7e-51.
Затем было получено множественное выравнивание гомологов и самого белка программой Muscle.
Из выравнивания были удалены короткие белки:
UniRef50_UPI0004739C68 (265)
UniRef50_A0A0E9L6H6 (174)
UniRef50_A0A0P1ERC5 (219)
UniRef50_A8TCJ5 (279)
UniRef50_A0A0F4J1B4 (194)
UniRef50_Q8N5H5 (236)
UniRef50_K1XA78 (192)
UniRef50_T1B9S7 (260)
UniRef50_X0X1C2 (250)
UniRef50_T0Z9G3 (209)
UniRef50_T0Y452 (132)
UniRef50_B8PZW5 (259)
UniRef50_T1ABX1 (202)
UniRef50_A0A0T9AW15 (140)
UniRef50_A0A0T9B5R2 (186)
UniRef50_A0A0P5TBN9 (270)
Плохо выравненные последовательности, которые также были удалены:
UniRef50_A0A0T6A602
UniRef50_W7K965
UniRef50_A0A093YIF7
UniRef50_A0A0L1HIX3
UniRef50_M2S4D0
UniRef50_A0A0F9ARR4
UniRef50_BOEQA9
Файл с итоговым выравниванием: здесь.
Выравнивание было добавлено в программу JalView.
Первая последовательность - последовательность исходного белка, с ней ассоциирована 3D-структура данного белка,
в соответствии с которой была построена разметка ("М" в "TM_REAL").
Позиции, соответствующие трансмембранным спиралям,
в основном представлены гидрофобными аминокислотами и являются высоко консервативными (но опять же далеко не всегда)
(правда, в случае гидрофобных остатков по большей части сохраняется лишь функциональная консервативность).
Однако участки между спиралями далеко не всегда вариативные, есть довольно довольно консервативные.
Интересно, что в трансмембранных участках присутствуют полярные остатки. Не очень понятно, с чем это связано, возможно это имеет большое
значания для функционирования фермента (возможно, полярные аминокислоты входят в активный центр фермента).
Ссылка на JalView прект: здесь.
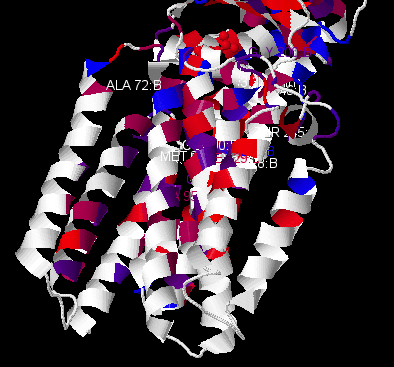 Рис. 1. Пространственная структура NAD(P) трансгидрогеназы (цепь B, PDB: 4O9U). Раскраска:Hydropgobisity - наиболее гидрофобные остатки - красным,
наименее - ярко-синим, промежуточные, такие, как Tyr, Gly - имеют промежуточные оттеки.
Above Identity Threshold (порог 20%).
Ориентация белка: часть, ориентированная в n-сторону мембраны - сверху (положительно заряженных остатков много)
, а ориентированная в p-сторону - снизу (преобладают отрицательно заряженные аминокислоты).
Рис. 1. Пространственная структура NAD(P) трансгидрогеназы (цепь B, PDB: 4O9U). Раскраска:Hydropgobisity - наиболее гидрофобные остатки - красным,
наименее - ярко-синим, промежуточные, такие, как Tyr, Gly - имеют промежуточные оттеки.
Above Identity Threshold (порог 20%).
Ориентация белка: часть, ориентированная в n-сторону мембраны - сверху (положительно заряженных остатков много)
, а ориентированная в p-сторону - снизу (преобладают отрицательно заряженные аминокислоты).
Затем для гомолога A1A1C4 было получено предсказание трансмембранных
доменов с помощью сервиса TMHMM. Результат предсказания - разметка в строке аннотаций "TM_PREDICTED" ("X").
В целом, предсказанные и реальные трансмембранные участки совпадают. Отличия возникают, возможно, потому что белки
все-таки немного разные. Общее число трансмембранных спиралей
(9) предсказано верно. Некоторые спирали оказываются длиннее, но они расположены в основном в одном месте последоватлеьности.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ
