Главная
Практикум №8: "Совмещение структур"
С помощью сервиса PDBeFold был произведен поиск структурных гомологов белка GlmU из бактерии Streptococcus cerevisiae (PDB-код: 4aaw).
Было выбрано 2 гомолога из разных организмов:
GlmU из Acinetobacter baumannii (5vmk) и GlmU из Mycobacterium tuberculosis (3foq). Сразу отметим, что хороших гомологов не было найдено, тут
длиной выравнивания менее 50% от длины вашего белка для обоих.
На Рис. 1 показано полученное совмещение структур.
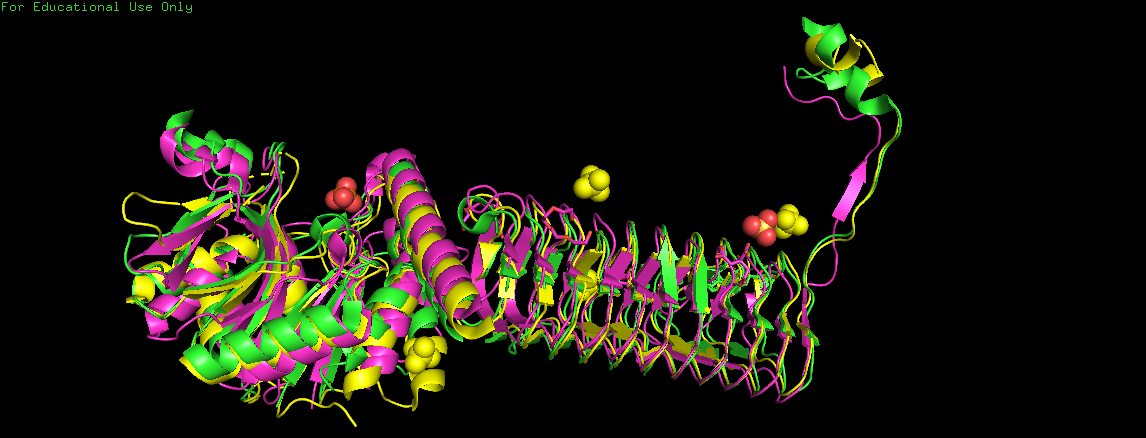 Рис. 1. Совмещение структур (4aaw показана зеленым, 3foq - желтым, 5vmk - сиреневым)
Видим, что в целом структуры белков достаточно хорошо согласуются не очень хорошо, есть отличия не только в петлях, но и в регулярных участках.
Рис. 1. Совмещение структур (4aaw показана зеленым, 3foq - желтым, 5vmk - сиреневым)
Видим, что в целом структуры белков достаточно хорошо согласуются не очень хорошо, есть отличия не только в петлях, но и в регулярных участках.
На Рис. 2 представлено полученное структурное выравнивание.
Рис. 2. Выравнивание по структуре.
Было построено выравнивание последовательностей программой Muscle
с параметрами по умолчанию (рис. 3).
Рис. 3. Выравнивание, построенное программой Muscle.
Пример несовпадения выравнивания по структуре с выравниванием по последовательности - позиция 15 (Muscle дает глицины для всех трех последовательностей,
а в выравнивании по структуре в структуре 5vmk на данном месте стоит аланин). Рис. 4 демонстрирует положение аминокислот в данной позиции в структурном
выравнивании.
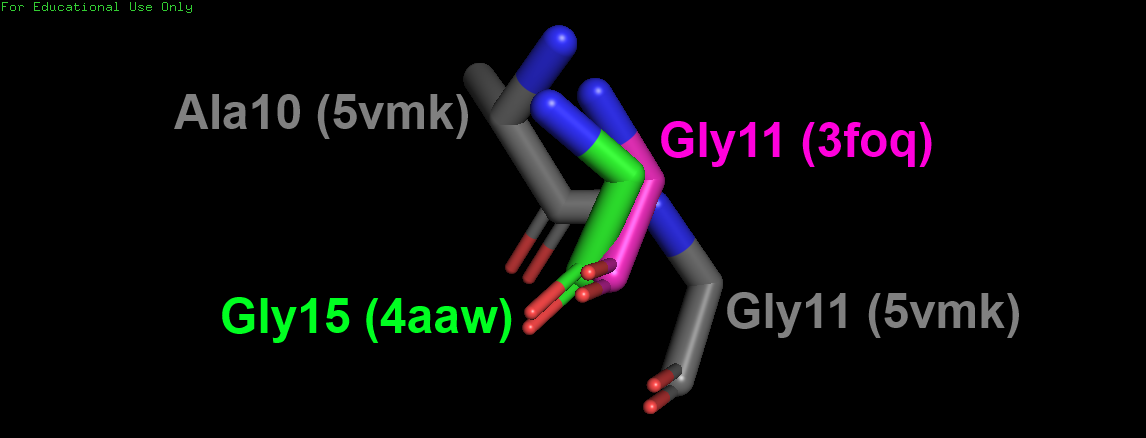 Рис. 4. положение аминокислот в позиции 15 в структурном
выравнивании. Дополнительно показан Ala16 для структуры 5vmk.
Рис. 4. положение аминокислот в позиции 15 в структурном
выравнивании. Дополнительно показан Ala16 для структуры 5vmk.
Видим, что Ala16 в структуре 5vmk лучше, чем Gly15 выровнен с глицинами двух других структур, что позволяет говорить о том, что структурное
выравнивание является более точным.
Совмещение по заданному выравниванию
Для задания была выбрана одна структура константного домена T-клеточного рецептора из цепи альфа (2BNQ, участок D: 115 - 204)
и одна — из цепи бета (2AXH, участок A: 119 - 244).
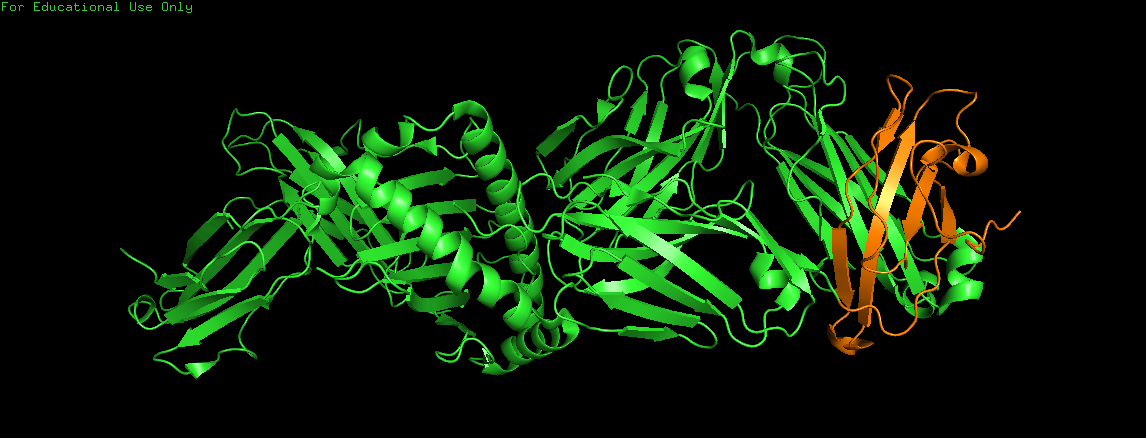 Рис. 5. Структура Структура 2BNQ, оранжевым выделен выбранный участок.
Рис. 5. Структура Структура 2BNQ, оранжевым выделен выбранный участок.
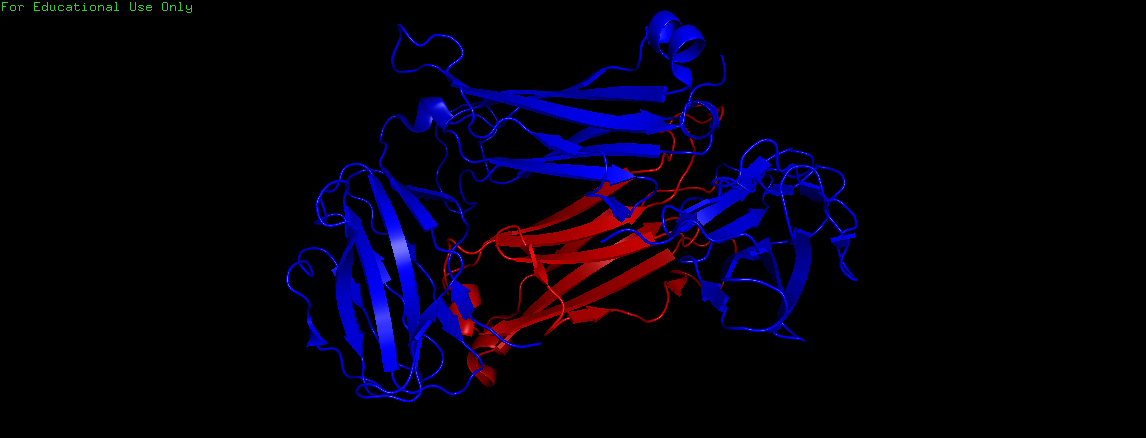 Рис. 6. Структура Структура 2AXH, красным выделен выбранный участок.
Рис. 6. Структура Структура 2AXH, красным выделен выбранный участок.
С помощью сервера SheeP были получены карты β-листов для выбранных участков (Рис. 7-8). Для того чтобы ориентация бета-листов совпадала,
к карте для цепи бета была применена команда flip columns. Из двух бета-листов для цепи бета верхний совпадает с листом на карте для цепи альфа.
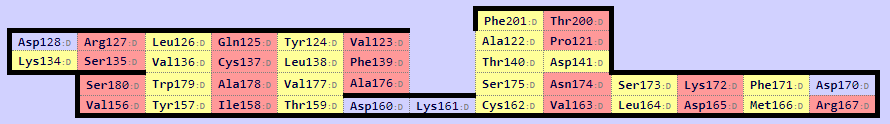 Рис. 7. Карта β-листов для цепи альфа.
Рис. 7. Карта β-листов для цепи альфа.
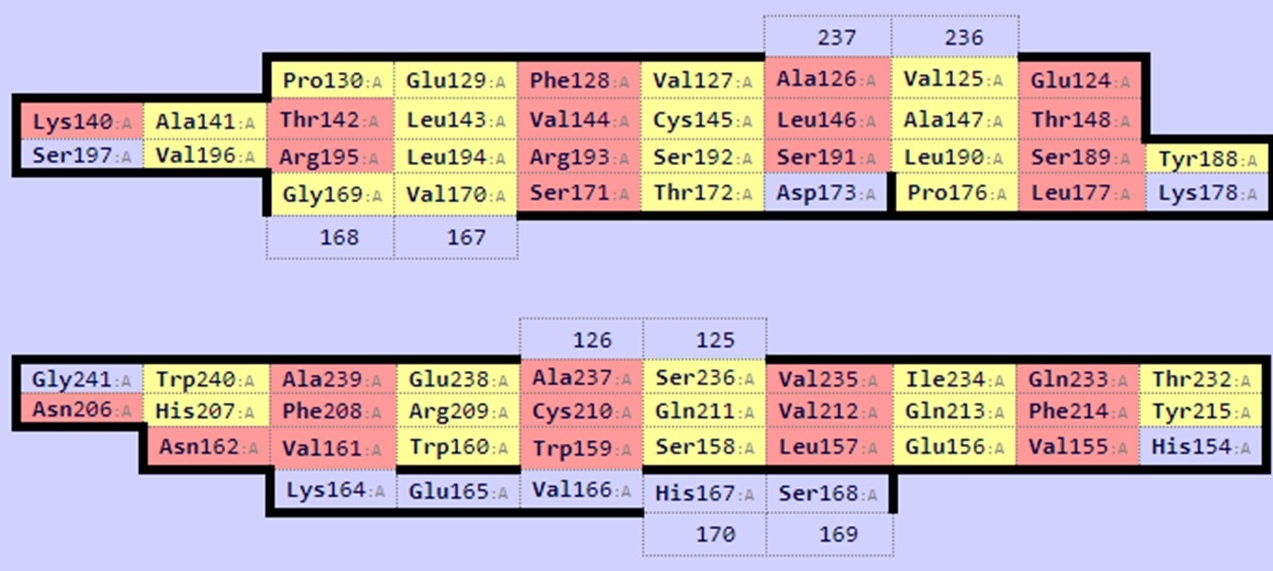 Рис. 8. Карта β-листов для цепи альфа.
Рис. 8. Карта β-листов для цепи альфа.
В каждой карте был найден консервативный остаток цистеина:
Cys137 в цепи альфа и Cys145 в цепи бета. По ним и окружающим их остаткам затем строилось выравнивание. Файл с совмещенными структурами: здесь.
Использовались следующие команды:
select alpha, alpha_2bnq and chain D and resi 124-126+136-138+177-179 and name CA
select beta, beta_2axh and chain A and resi 126-128+144-146+191-193 and name CA
pair_fit alpha, beta
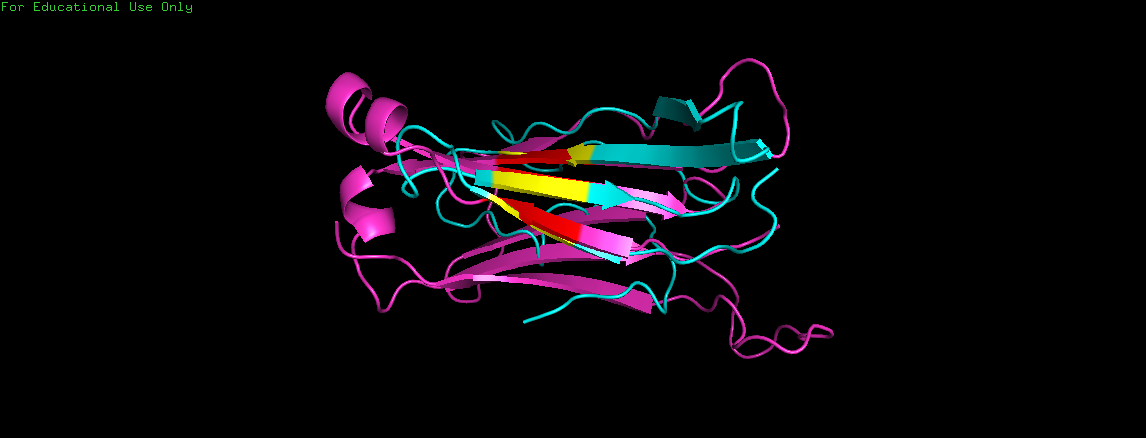 Рис. 9. Совмещение участков цепей альфа и бета с помощью команды pair_fit.
Альфа-цепь покрашена светло-голубым цветом, бета - сиреневым. Остатки, по которым производилось совмещение,
выделены желтым (на альфа-цепи) и красным (на бета-цепи).
Рис. 9. Совмещение участков цепей альфа и бета с помощью команды pair_fit.
Альфа-цепь покрашена светло-голубым цветом, бета - сиреневым. Остатки, по которым производилось совмещение,
выделены желтым (на альфа-цепи) и красным (на бета-цепи).
Выдим, что несмотря на то, что направление и расположение β-тяжей одинаковы, далеко не все элементы вторичной структуры совпадают,
вследствие чего говорить об идентичности топологий нельзя.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ









