На этой странице проводится сравнение структуры белка CPAF с похожими белками. Сравнение проводится с помощью сервиса PDBeFold www.ebi.ac.uk/msd-srv/ssm/. Сначала провелось сравнение со стандартными параметрами (70% сходства по вторичным структурам), но сервис нашел только белки, прописанные как один и тот же. Сделав сходство 50%, удалось найти еще несколько белков, но их сходство было довольно слабо (<15% по последовательности аминокислотных остатков). Всего сервис проверил 95775 структур и 247218 цепочек. Таблицу с результатами можно скачать по ссылке: Таблица (на первом листе представлены результаты с 70% сходства, на втором - с 50%.
Я выбрала структуру #9, белок с неизвестной функцией BACUNI_00178 (PDB-идентификатор 4GHN). BACUNI_00178 - родственник CPAF (пептидаза из той же белковой семьи), но достаточно отдаленный, так как бактерия, синтезирующая его - Bacteroides uniformis, не являющаяся родственником хламидии. Изображение совмещенных белков и более близкое приближение гэпа в накладывающейся структуре изображено на рисунках 1 и 2.
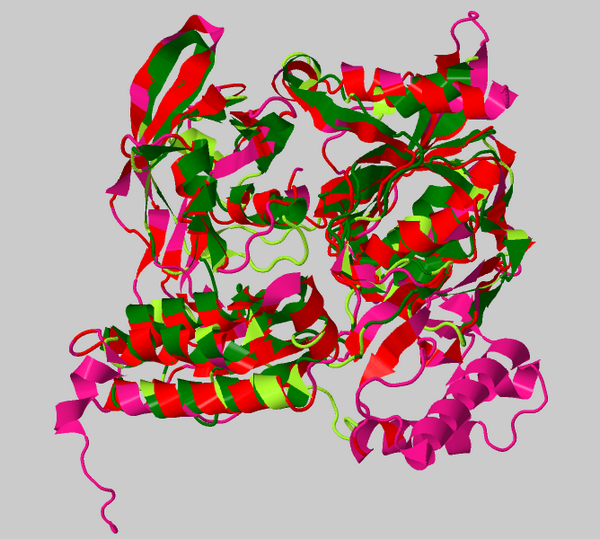
Рис.1 Наложение структур белков CPAF и BACUNI_00178. Зеленым показана цепь CPAF, красным - BACUNI_00178. Участки с расстоянием между CA-атомами двух белков больше 1.5 ангстрем показаны соответственно желто-зеленым и розовым. Видно, что совпадение элементов вторичной структуры достаточно неплохое.
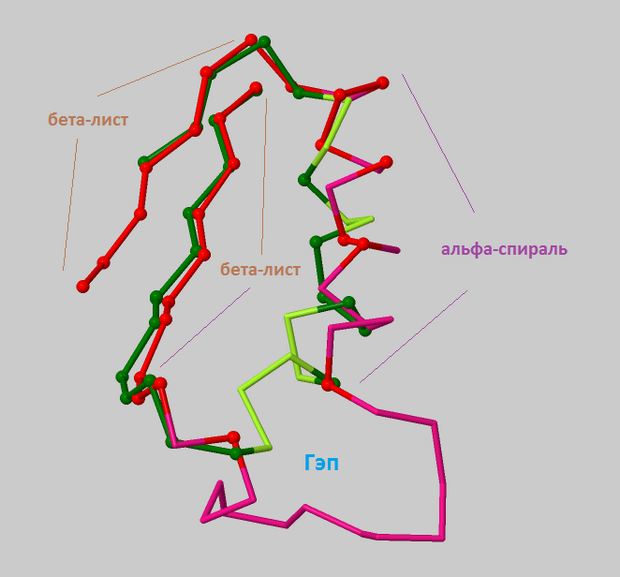
Рис.2 Участок наложенных структур. Видно совпадение некоторых элементов, таких как альфа-спирали и бета-листы, а также участок, где структуры расходятся (гэп).