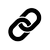Выравнивание - это постановка в соответствие аминокислот из разных последовательностей. Для этого практикума было взято готовое выравнивание MHCI_C1.alignment.fasta. Обработка выравниваний производилась программой JalView. Готовый проект можно скачать здесь в формате .jar
1. Выделение блока
Сначала были выбраны два вертикальных блока. Изображения блоков приведены на рисунках 1 и 2.
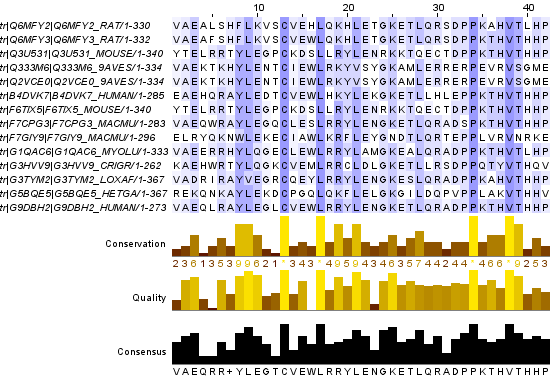
Рис.1 Первый вертикальный блок в выравнивании последовательностей. Используется раскраска BLOSUM62. |
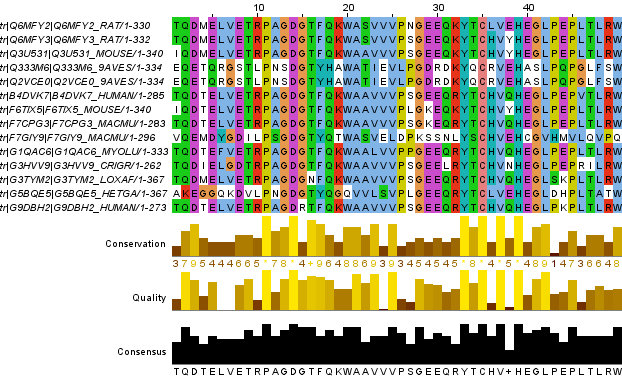
Рис.2 Второй вертикальный блок в выравнивании последовательностей. Используется раскраска ClustalX. |
На рисунке 3 представлен участок с малой сходимостью последовательностей. Как видно из рисунка, на участке две последовательности полностью совпадают, а остальные им мало гомологичны.
Рис.3 Участок с малой сходимостью. Две совпадающие последовательности выделены.
2. Консервативность в выбранном блоке
Подсчитано количество консервативных позиций в первом вертикальном блоке. Среди них:
- консервативные на 70% - 19/42
- функционально консервативные на 70% – 26/42
- абсолютно функционально консервативные – 7/42
- абсолютно консервативные - 4/42
3. Близко расположенные вертикальные блоки
Я нашла два вертикальных блока, разделенных участком с гэпами. Поскольку столбцов с гэпами всего 3, и они разделены сравнительно консервативной областью, можно считать 2 блока объединенными в кластер.
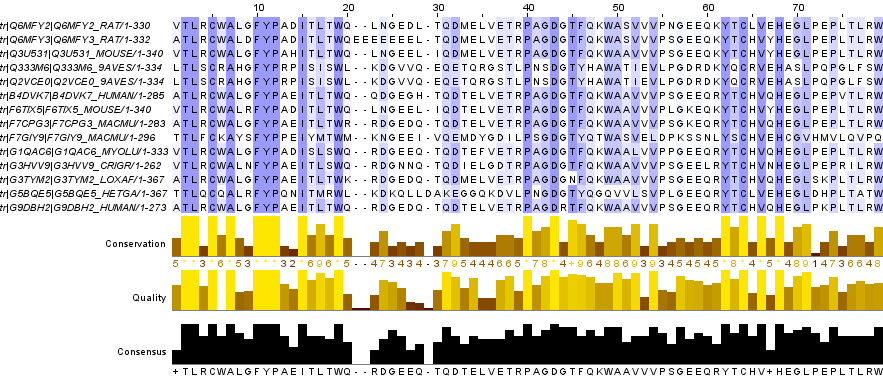
Рис.4 Кластер из двух блоков.
4. Выравнивание последовательности
Последовательность была выравнена со вторым блоком. Выравнивание приведено на рисунке 5.
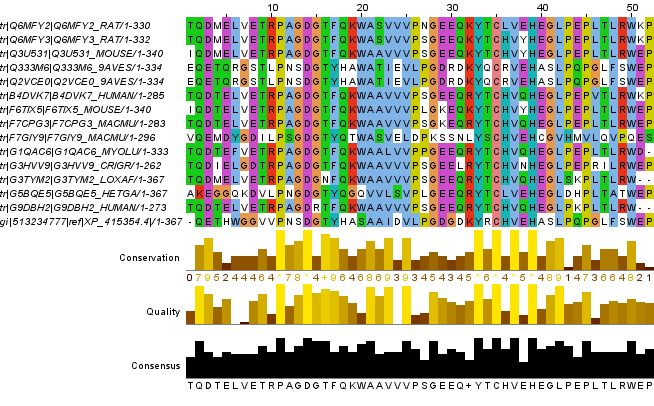
Рис.5 Выравненная со вторым вертикальным блоком последовательность.
5. Консенсусная последовательность и LOGO
Для полученного блока (включающего оригинальные последовательности и добавленную к ним) была получена консенсусная последовательность (из шкалы "Consensus" в JalView).
>Consensus/1-52 Percentage Identity Consensus TQDTELVETRPAGDGTFQKWAAVVVPSGEEQKYTCHVEHEGLPEPLTLRWEP
Также на сайте WebLogo было получено LOGO этого блока.

6. "Выравнивание" негомологичных белков
Для "выравнивания" были взяты последовательности шести моих однокурсников. Идентификаторы были переведены в формат UniProt на сайте UniProt, и последовательности был получены через File -> Fetch Sequence(s).
Были взяты последовательности:
- Оли Васюткиной (D7DTP4)
- Ярослава Лозинского (A1A2B5)
- Светы Яровенко (A9WC34)
- Иры Хвень (Q8KDH8)
- Сони Буяновой (Q8ZUP0)
- Димы Пензара (D7GXG0)
Выравнивание производилось программой MUSCLE. Выбраны два "блока", где программа обнаружила кажущуюся сходимость аминокислот. "Блоки" приведены на рисунках 6 и 7. Видно, что никакой сходимости вне построенных колонок между последовательностями нет.
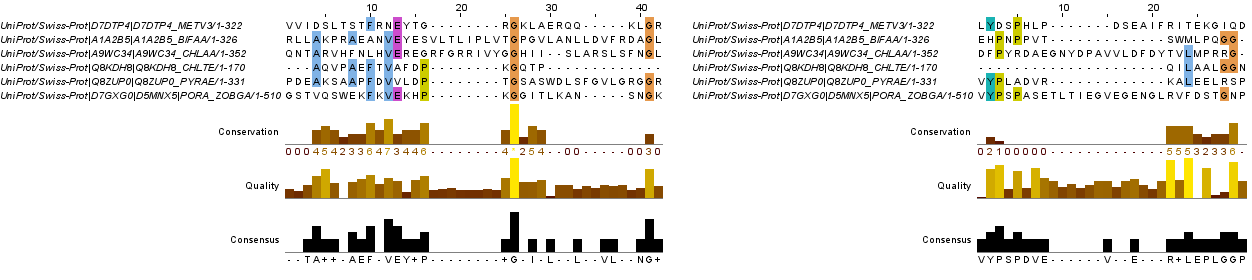
Рис.6 и 7. Ложное выравнивание негомологичных последовательностей. Использована раскраска ClustalX с порогом консервативности 50%.